20 用测试数据进行一键式WGCNA没问题,但换自己的数据运行后,Report显示“WGCNA 尚未运行完毕,请稍后再试!”
老师,我想请教一下我在使用一键式WGCNA分析数据时遇到的问题。
我用调研图老师给的测试数据(https://shengxin.ren/article/443)进行一键式WGCNA没有问题,但将自己的数据(15个样品,24400个基因,30个生理指标)转化成相同格式后用默认参数跑,得到的Report就显示“WGCNA 尚未运行完毕,请稍后再试!”,请问是什么原因,是我上传的数据格式有问题吗?
补充:
我把SD值改成0或0.6再跑都是“尚未运行完毕”,只是结果里比SD值1.2多分出十几个模块。
我的数据跟测试数据跑出来的结果比,只运行到ModuleTraitRelationships这一步,而且ModuleTraitRelationships的pdf文件不完整,打不开,缺失txt和jpg。 后面的ModuleHubGene_xx.pdf、ModuleHubGeneDetail_xx.txt、ModuleRelationships.jpg、ModuleRelationships.pdf、CytoEdge_xx.txt、CytoNode_xx.txt、inputJs.js、Network_count.txt这些文件都没有跑出来。
下附软件截图及代码
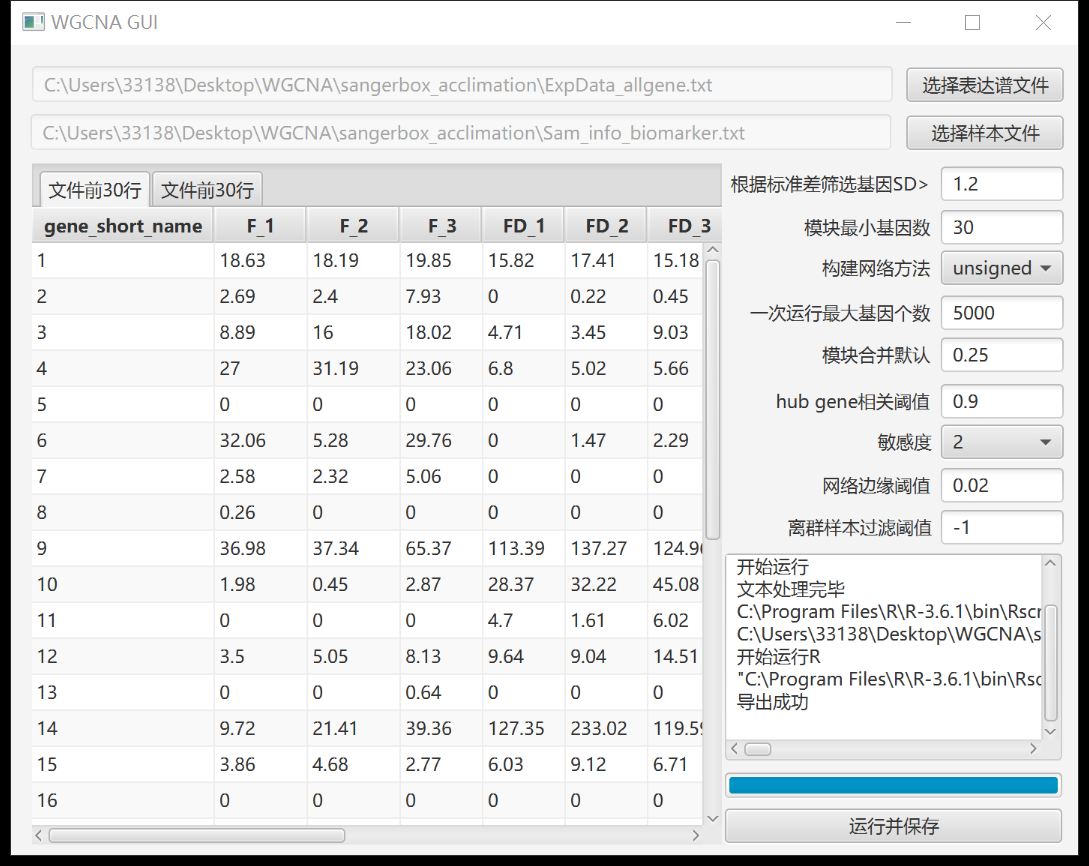
读取文件完毕
读取文件完毕
开始运行
文本处理完毕
C:\Program Files\R\R-3.6.1\bin\Rscript.exe
C:\Users\33138\Desktop\WGCNA\software\Sanger_V1.0.9\source\softs\5c7344303916c\WGCNA_Pipline.R
开始运行R
"C:\Program Files\R\R-3.6.1\bin\Rscript.exe" "C:\Users\33138\Desktop\WGCNA\software\Sanger_V1.0.9\source\softs\5c7344303916c\WGCNA_Pipline.R" "C:\Users\33138\Desktop\result_acclimation" "success_ExpData_allgene.txt" "success_Sam_info_biomarker.txt" "1.2" "30" "unsigned" "5000" "0.25" "0.9" "2" "0.02" "-1" "1"
导出成功
- 3 关注
- 1 收藏,3265 浏览
- zhuganglie 提出于 2019-11-26 16:33