在R中求一致性指数( Harrell'concordance index:C-index)案例
看教程不够直观,那就看视频吧! >>点击加载视频
什么是一致性指数?
C-index,英文名全称concordance index,中文里有人翻译成一致性指数,最早是由范德堡大学(Vanderbilt University)生物统计教教授Frank E Harrell Jr 1996年提出,主要用于计算生存分析中的COX模型预测值与真实之间的区分度(discrimination),和大家熟悉的AUC其实是差不多的;在评价肿瘤患者预后模型的预测精度中用的比较多。一般评价模型的好坏主要有两个方面,一是模型的拟合优度(Goodness of Fit),常见的评价指标主要有R方、-2logL、AIC、BIC等;另外一个是模型的预测精度,顾名思义就是模型的真实值与预测值之间差别大小,均方误差,相对误差等。在临床应用上更注重预测精度,建模的主要目的是用于预测,而C-index它就属于模型评价指标中的预测精度。
C-index的计算方法是把所研究的资料中的所有研究对象随机地两两组成对子,以生存分析为例,两个病人如果生存时间较长的一位其预测生存时间长于另一位,或预测的生存概率高的一位的生存时间长于另一位,则称之为预测结果与实际结果相符,称之为一致。
C-index的计算步骤为:
一、所有样本互相配对,共有N*(N-1)/2对,其中N为样本数
二、去除配对中两个病人都没有达到事件终点(比如死亡),或者其中的一个病人A的生存时间短于另一个病人B,然而病人A还没有到达事件终点(死亡)~ps:这种配对无法判断出谁先死的。此时剩下的配对数记为:M
三、计算剩下的配对中,预测结果和实际相一致的配对数记为K,即(两个病人如果生存时间较长的一位其预测生存时间长于另一位,或预测的生存概率高的一位的生存时间长于另一位,则称之为预测结果与实际结果相符,称之为一致)
(4)计算C-index=K/M。
从上述计算方法可以看出C-index在0.5-1之间(任意配对随机情况下一致与不一致刚好是0.5的概率)。0.5为完全不一致,说明该模型没有预测作用,1为完全一致,说明该模型预测结果与实际完全一致。一般情况下C-index在0.50-0.70为准确度较低:在0.71-0.90之间为准确度中等;而高于0.90则为高准确度,跟相关系数有点类似。
光从C-index一个数字上还是很难以衡量到底是准确度高还是低,所以人们就想着用一个统计学检验来说服证明这个高低,正如筛选基因差异是光看差异倍数来判断表达差异还过于武断,此时引入重抽样技术(Bootstrap)来检验预测模型的准确度。Bootstrap是非参数统计中一种重要的估计统计量方差进而进行区间估计的统计方法。
Bootstrap方法核心思想和基本步骤如下:
(1)采用重抽样技术从原始样本中抽取一定数量的样本,此过程允许重复抽样。
(2)根据抽出的样本计算给定的统计量T。
(3)重复上述N次(一般大于1000),得到N个统计量T。
(4)计算上述N个统计量T的样木方差,得到统计量的方差。
另如果数据集很大的话可以按照不同的比例将数据集拆分,一部分用于建模一部分用于验证。关于交叉验证(Cross-validation),如5-fold、10-fold等。
虽然看起来很复杂,但是事实上已经有人做了这些事情,在R中有包可以直接计算一致性指数:Hmisc 、compareC,两个包都可以计算c-index
今天就用一个例子来说明一下,数据准备:
表达谱矩阵:
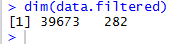 生存时间和死亡事件
生存时间和死亡事件
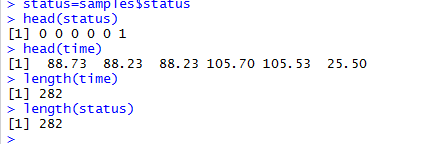
具体代码如下:
方法一、
library(survival)
library(Hmisc)
fmla <- as.formula(paste0("Surv(time, status) ~",paste0(row.names(data.filtered)[1:10],collapse = '+')))
cox <- coxph(fmla, data = as.data.frame(t(data.filtered)))#构建多因素生存模型
fp <- predict(cox)#模型预测
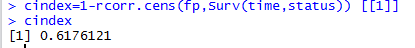
cindex=1-rcorr.cens(fp,Surv(time,status)) [[1]]#计算一致性指数
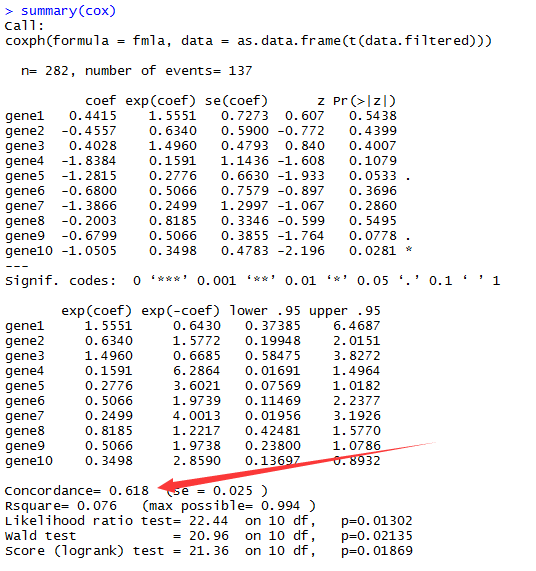
方法二、
summary(cox)
 方法三
方法三
library(survcomp)
library(survival)
fmla <- as.formula(paste0("Surv(time, status) ~",paste0(row.names(data.filtered)[1:10],collapse = '+')))
cox <- coxph(fmla, data = as.data.frame(t(data.filtered)))#构建多因素生存模型
cindex <- concordance.index(predict(cox),surv.time = time, surv.event = status
,method = "noether")
cindex$c.index; cindex$lower; cindex$upper
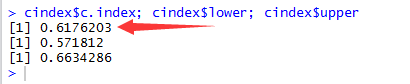 从以上结果可以看出方法三直接输出了95%的置信区间
从以上结果可以看出方法三直接输出了95%的置信区间
- 发表于 2017-06-26 15:32
- 阅读 ( 38640 )
- 分类:软件工具
你可能感兴趣的文章
- 分享一段比较不同模型的C-index和RMS的代码 6075 浏览
