拿TCGA肝癌的RNA-Seq数据来做差异分析例子
看教程不够直观,那就看视频吧! >>点击加载视频
刚写了一篇如何用GEO数据来做差异的例子如:https://www.shengxin.ren/article/207
这次来用TCGA的肝癌的数据来演示以下,我们选择大家比较喜欢的RNA-Seq Counts数据
使用TCGA简易下载工具选择肝癌RNA-Seq Counts数据进行下载如:
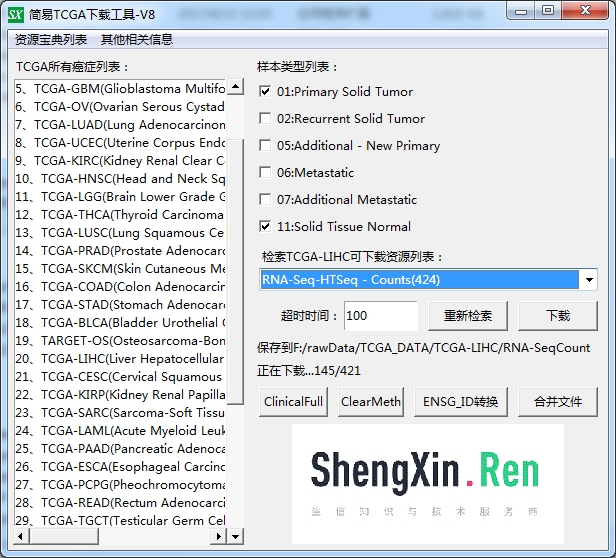 下载完成后我们共得到421个样本数据如:
下载完成后我们共得到421个样本数据如:
 进一步的我们需要合并这些样本数据,因为我们下载的是癌与癌旁的样本,使用TCGA简易下载工具的合并矩阵功能就可以将这些样本合并在一个矩阵当中,但是这里涉及到一个问题样本合并的顺序是什么样的呢,有些同学经常问能不能将癌症样本合并到前面,癌旁样本合并在后面呢,这个问题今天就来给大家操作一下
进一步的我们需要合并这些样本数据,因为我们下载的是癌与癌旁的样本,使用TCGA简易下载工具的合并矩阵功能就可以将这些样本合并在一个矩阵当中,但是这里涉及到一个问题样本合并的顺序是什么样的呢,有些同学经常问能不能将癌症样本合并到前面,癌旁样本合并在后面呢,这个问题今天就来给大家操作一下
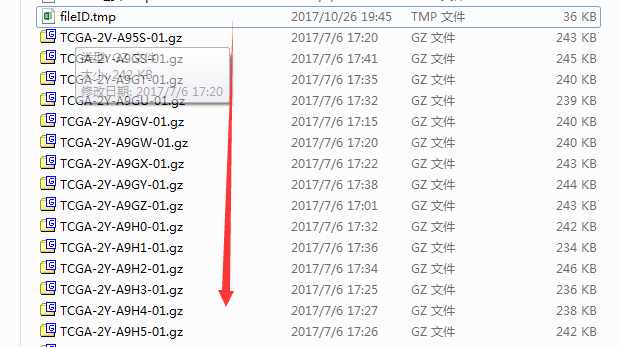
首先我们需要编辑一下fileID.tmp文件,因为合并样本的顺序以及合并哪些样本完全由fileID.tmp控制,所以我们编辑fileID.tmp就可以满足我们的合并需要,打开fileID.tmp可以看到共有三列,421行,即每行代表一个样本。
从第二行可以看出样本信息,即-01.gz结尾的表示癌症样本,-11.gz结尾的表示癌旁样本
我们先把第二列为-01.gz的行筛选并复制出来如:

共有369个样本
紧接着第二列为-11.gz的文件筛选并复制出来,共50行,放到-01.gz的那369行后面共419个样本,剩下的样本我们就舍弃了,为什么呢,仔细看可以发现少了两个样本,这两个样本其实是重复样本,简单处理直接丢弃,样本量够大,少一两个样本不会影响结果。
 得到了一个新表格,我们使用这个表格的数据替换fileID.tmp里的内容。
得到了一个新表格,我们使用这个表格的数据替换fileID.tmp里的内容。
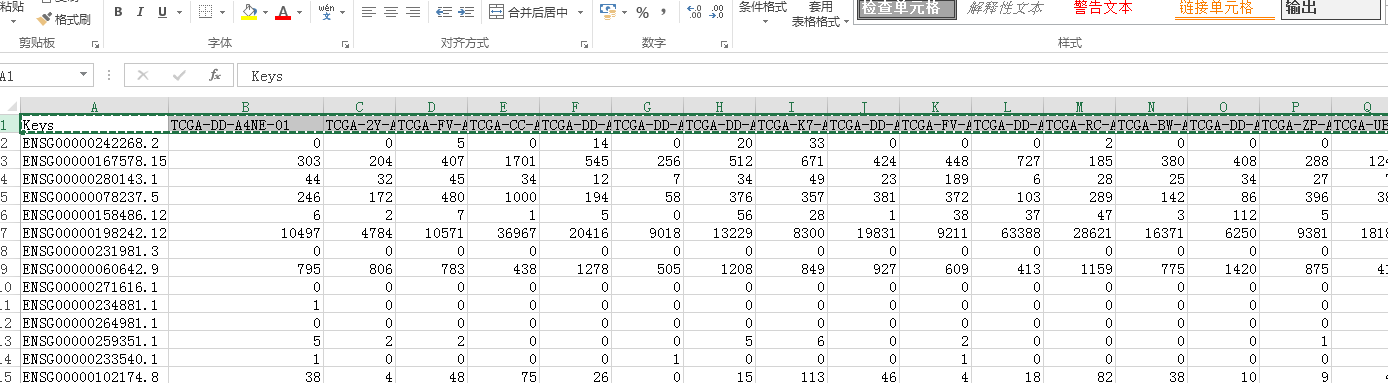
然后我们点击 简易TCGA下载工具 的合并文件功能进行合并得到最终的表达矩阵如:Merge_matrix.txt
打开看看如下:
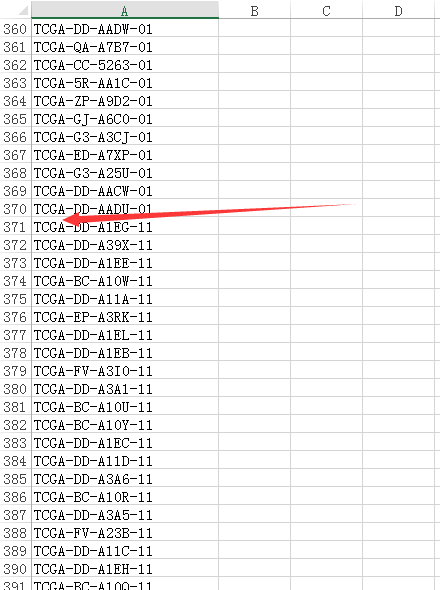
 我们把第一行样本复制出来,转置一下看看样顺序
我们把第一行样本复制出来,转置一下看看样顺序
很明显 正常样本在最后了
 进一步的我们编辑样本分组表以便进行差异筛选,如:
进一步的我们编辑样本分组表以便进行差异筛选,如:
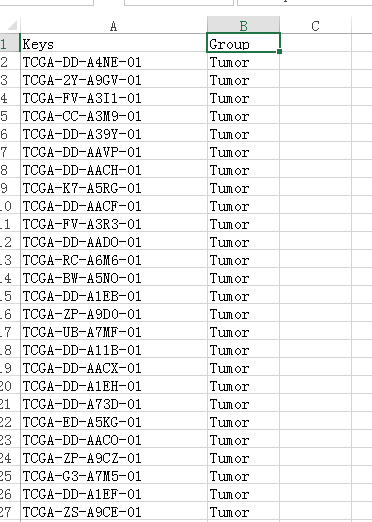 将样本分组表保存为一个新的文本文件,我们命名为tcga_group.txt
将样本分组表保存为一个新的文本文件,我们命名为tcga_group.txt
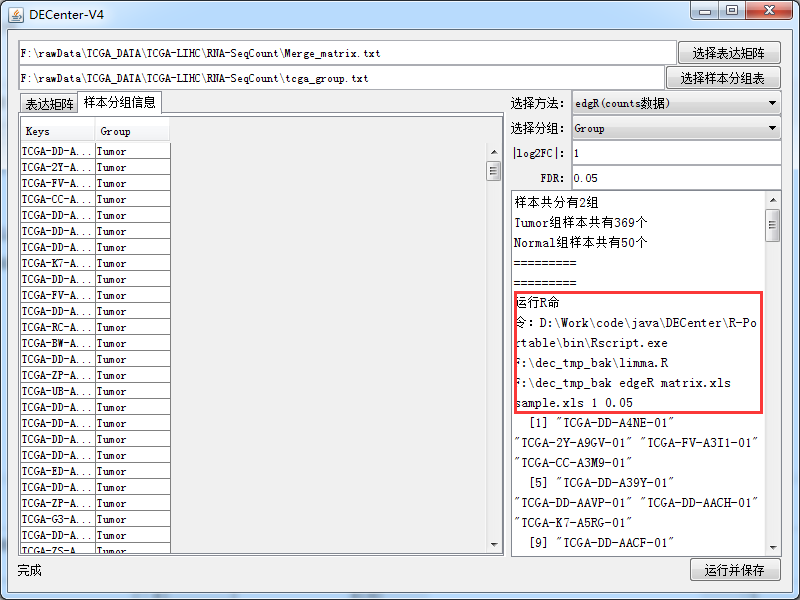
进一步的使用DECenter来筛选差异,因为样本数比较多,所以这一步会比较卡。
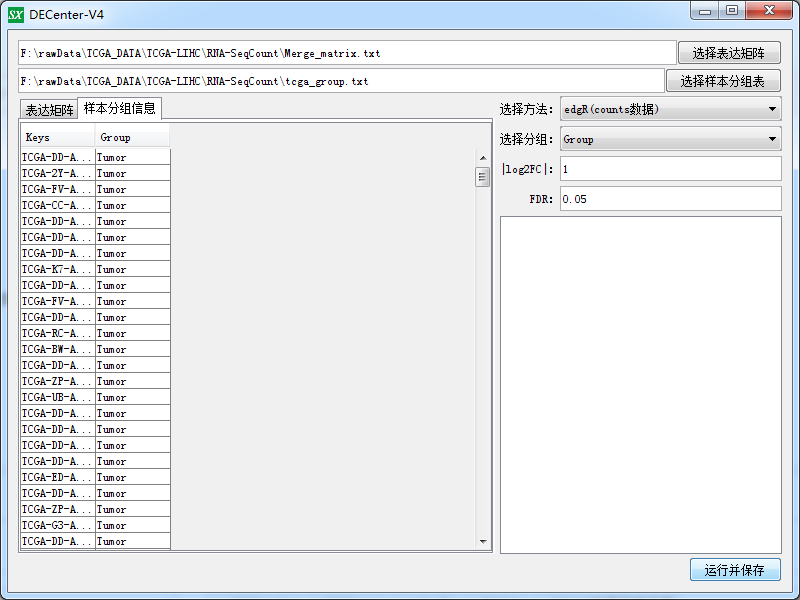 做完之后生成了三个文件如下:
做完之后生成了三个文件如下:
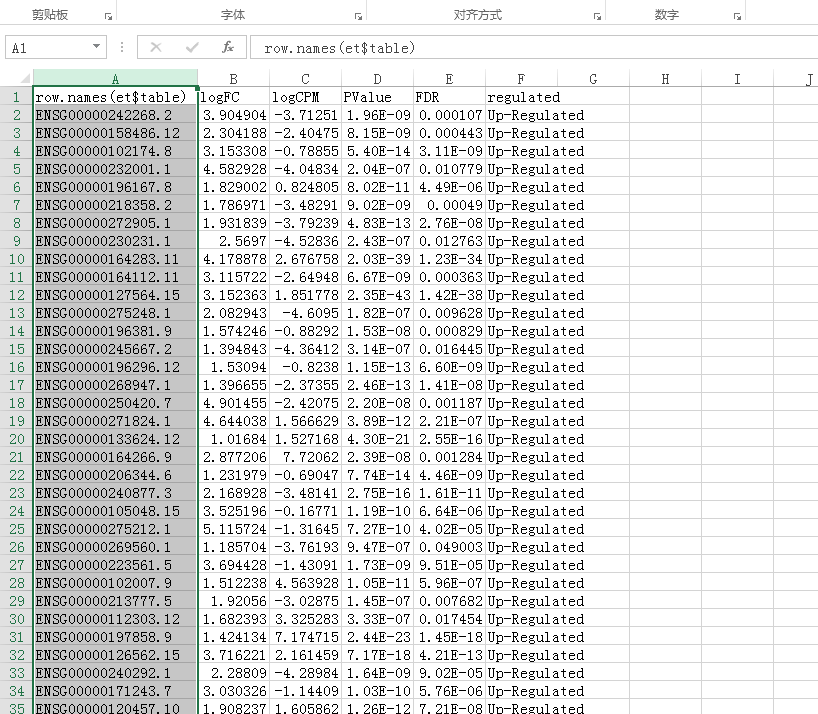
 打开Tumor-vs-Normal.edgeR_Dif.txt文件可以看到所有的差异都在里面了
打开Tumor-vs-Normal.edgeR_Dif.txt文件可以看到所有的差异都在里面了
到这里差异就筛选完了,但是里面的ID不是基因的,该怎么办
老办法,使用TCGA简易下载工具的ENSG_ID转换功能
但是我们需要做一下修改,TCGA简易下载工具的ENSG_ID转换功能支持的是表格矩阵,我们的Tumor-vs-Normal.edgeR_Dif.txt文件也是表格矩阵,但是最后一列不是数字,所以我们将最后一列换成数字,比如Up-Regulated替换成1,Down-Regulated替换成-1,如: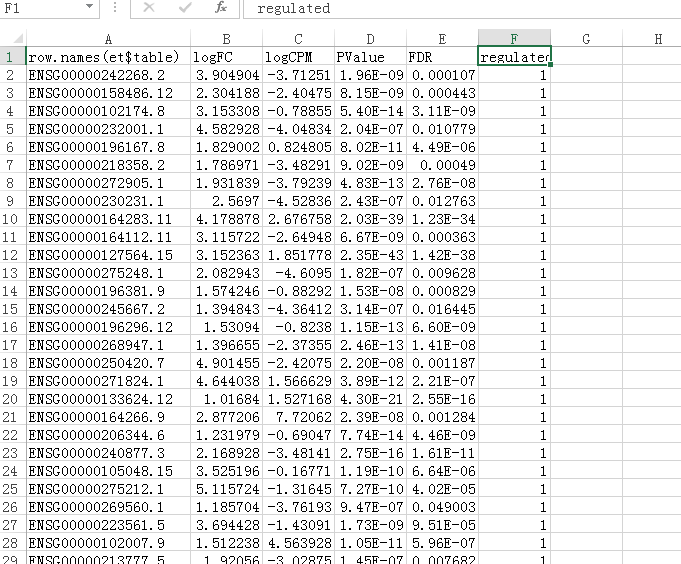
然后倒入 TCGA简易下载工具 的ENSG_ID转换
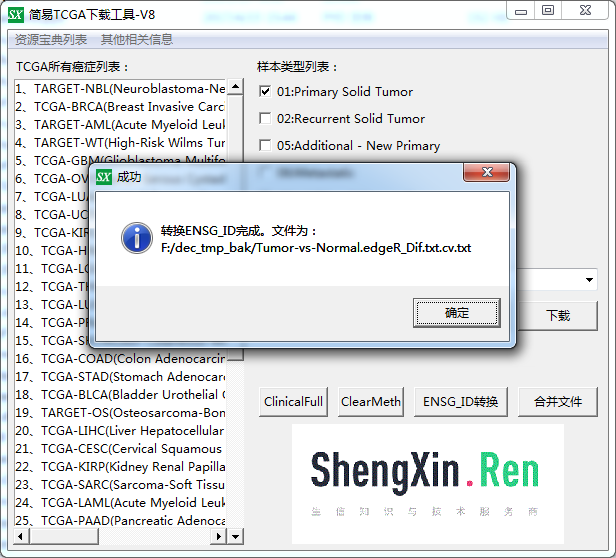
得到了编码基因和lncRNA的差异表达表格
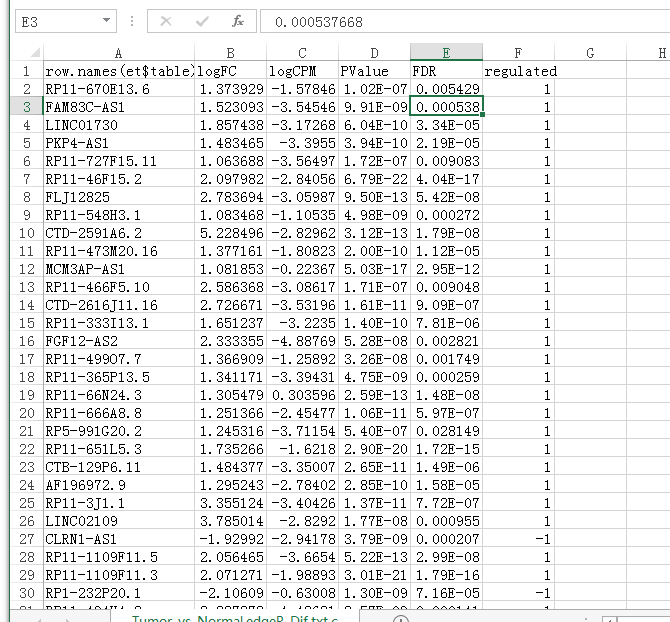
 lncRNA差异表达:
lncRNA差异表达:
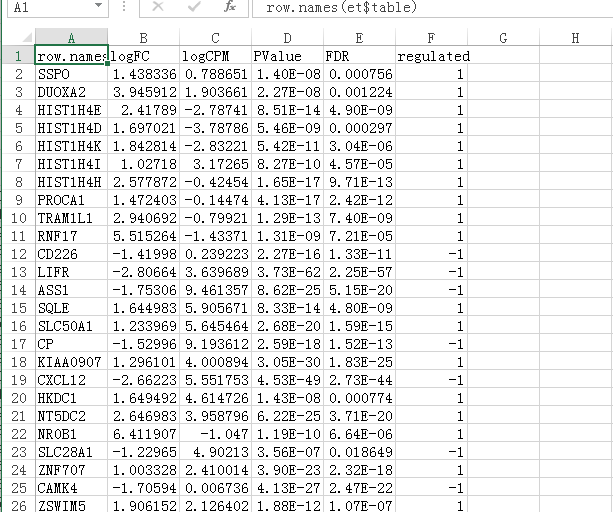
编码基因差异表达:
当然使用DESeq也可以,只是比较慢,要多等会,我的电脑筛这四百多个样本用了大概一两个小时。。。。。哭~~
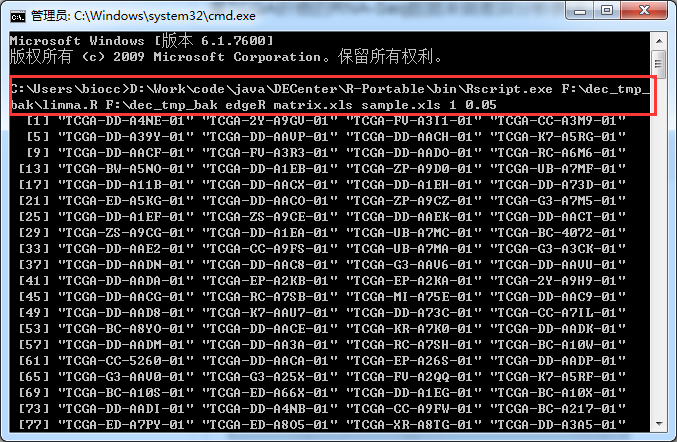
另外一个小技巧就是如果你觉得DECenter卡死,你可以复制那个cmd命令到你的cmd里面运行即可如:
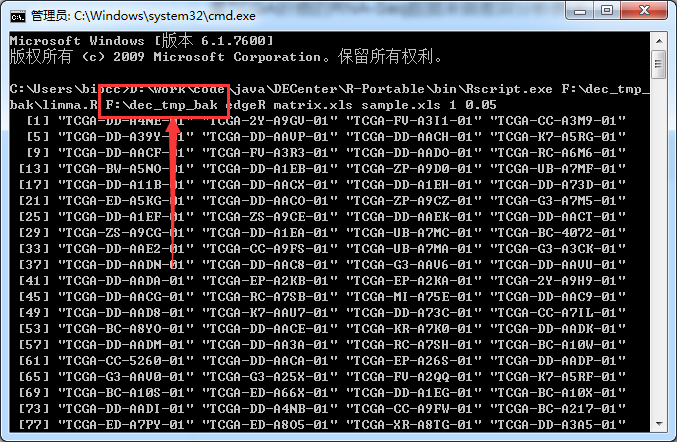
 最后结果会保存在这个目录下:
最后结果会保存在这个目录下:
- 发表于 2017-10-26 20:27
- 阅读 ( 34623 )
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9676 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10621 浏览
- 转录组Count数据差异分析工具 7249 浏览
- TCGA数据库 7841 浏览
- TCGA下载工具超级版,在线秒下载 7652 浏览
- 生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物 10170 浏览
