记录clusterProfiler GO分析的一些疑问
闲暇时突然想写一个GO富集分析的小工具,说到富集分析的话不得不提Y叔的clusterProfiler了,简单的使用clusterProfiler提供的GO富集分析的例子:
## Not run: data(geneList)de <- name...
看教程不够直观,那就看视频吧! >>点击加载视频
闲暇时突然想写一个GO富集分析的小工具,说到富集分析的话不得不提Y叔的clusterProfiler了,简单的使用clusterProfiler提供的GO富集分析的例子:
## Not run:
data(geneList)
de <- names(geneList)[1:100]
yy <- enrichGO(de, 'org.Hs.eg.db', ont="BP", pvalueCutoff=0.01)
head(yy)
## End(Not run)
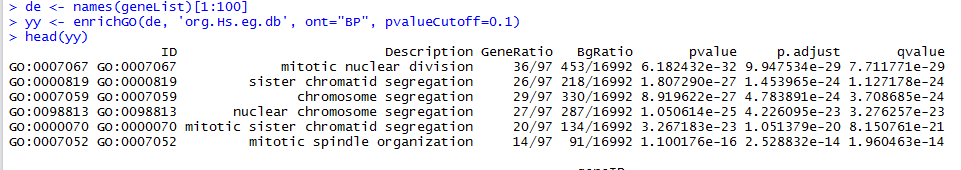
得到了富集分析的结果,写工具嘛首先需要的是结果可靠,所以根据富集分析的算法我们可以自己实现一个富集分析的功能,主要在于前期数据处理这块,从上述例子可以得到这六个GO term的ID,我们去GO数据库下载了go-basic.obo文件,通过自己的脚本进行解析出来(见教程:GO的obo文件解析Python代码)。
完成了GO Term的解析之后,我们要看一下我们解析的对不对,基于clusterProfiler的例子数据,我们可以进行模拟一下,首先这个例子是人的,所以我们先得到ENTREZID与GO的关系,怎么获得呢
通过这么一句R代码:
select(org.Hs.eg.db,keys(org.Hs.eg.db),columns = c('GO'))
我们就得到了人的所有ENTREZID与GO的关系,导出之后我们就得到了ENTREZID与GO的关系文件
有了这个关系文件,然后又有了GO Term之间的层次关系文件go-basic.obo,我们就可以统计出每个GO term对应的ENTREZID个数了,比如从例子数据结果中的BgRatio那一列,453/16992表示的意思就是GO:0007067中共有453个基因,所有的BP中的基因共有16992个,为了验证我们的结果我们看看我们程序匹配到的这个结果是否一致,我们的程序输出入:
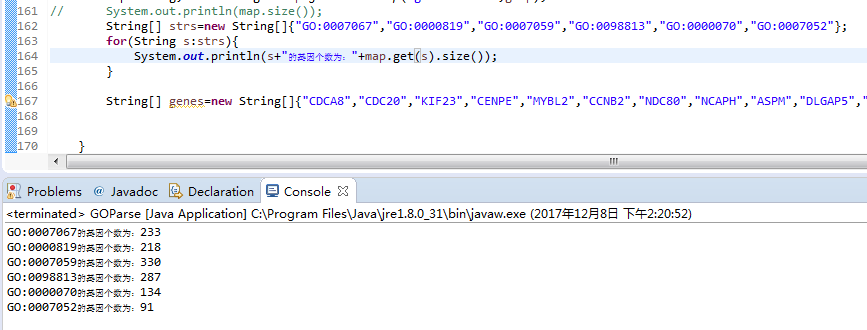
从中可以看出有五个是完全一致的,第一个不一致,为什么不一致,我们先使用GO检索一下这个GO ID,如:
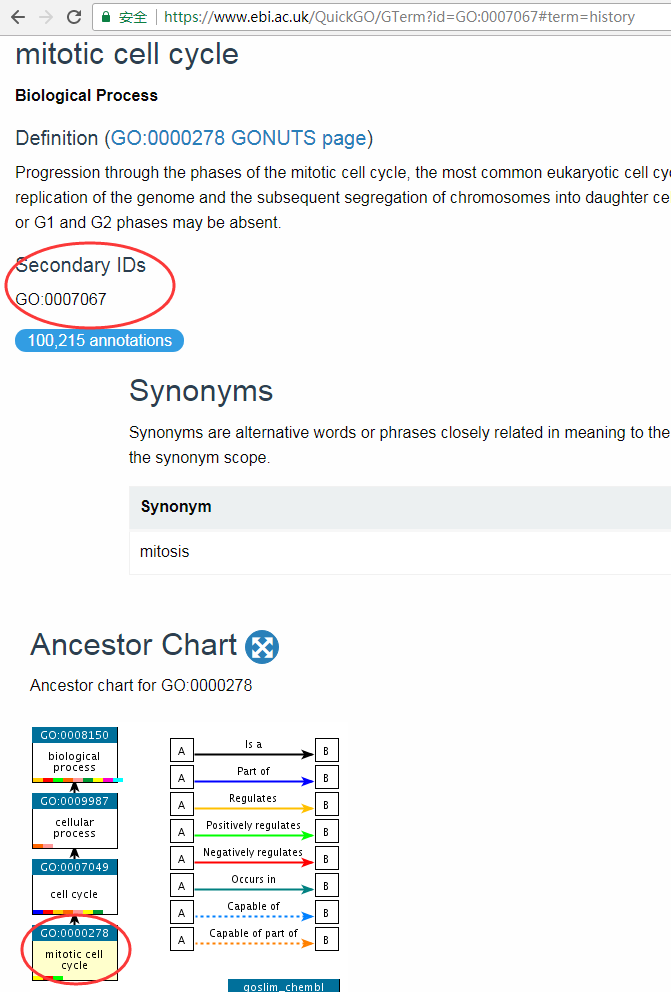
从中可以看出原来是这个ID已经过时不用了,我们的程序因为使用最新版的obo文件,所以结果也有点差别
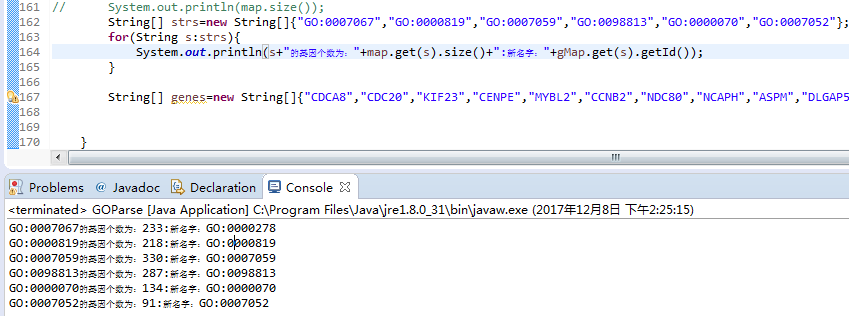
此记自己写的程序与clusterProfiler的一些微小差别,这里的233个基因并未考虑到层次关系所连接进来的基因,新的GO term的基因个数如:
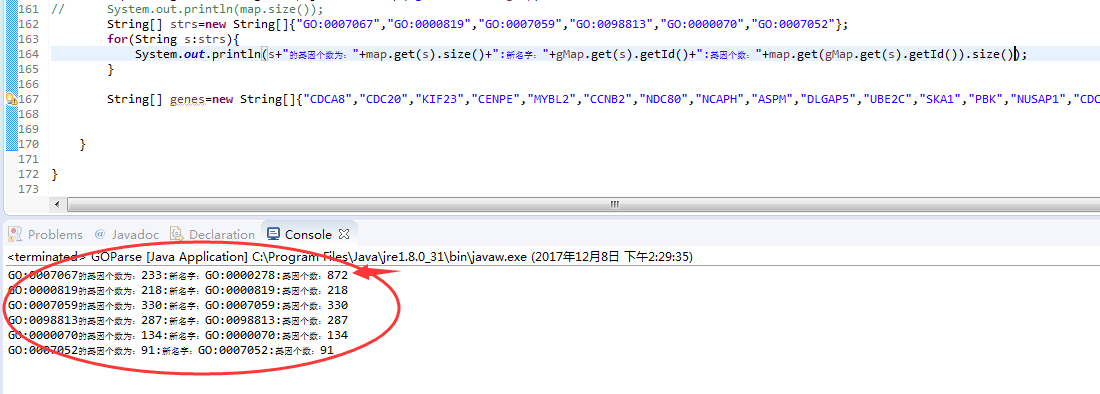 然后关于背景BP中的基因共有16992个,这个和程序解析出来的也是有出入的,在org.Hs.eg.db包中能注释上GO的ENTREZID有18907个,然而根据NCBI提供的ENTREZID转GeneSymbol中只有18892个ENTREZID对应有geneSymbol,故此背景数目有一些出入,但是这些出入并不会影响结果。
然后关于背景BP中的基因共有16992个,这个和程序解析出来的也是有出入的,在org.Hs.eg.db包中能注释上GO的ENTREZID有18907个,然而根据NCBI提供的ENTREZID转GeneSymbol中只有18892个ENTREZID对应有geneSymbol,故此背景数目有一些出入,但是这些出入并不会影响结果。- 发表于 2017-12-08 14:32
- 阅读 ( 18961 )
- 分类:软件工具
你可能感兴趣的文章
- GO的obo文件解析Python代码 9969 浏览
1 条评论
请先 登录 后评论
