批量计算相关系数工具使用教程
看教程不够直观,那就看视频吧! >>点击加载视频
相关表和相关图可反映两个变量之间的相互关系及其相关方向,但无法确切地表明两个变量之间相关的程度。
相关系数是用以反映变量之间相关关系密切程度的统计指标。相关系数是按积差方法计算,同样以两变量与各自平均值的离差为基础,通过两个离差相乘来反映两变量之间相关程度;着重研究线性的单相关系数。
在生物信息领域中相关系数使用很广泛,比如观察两个基因的表达模式,分析基因的共表达等等
我们开发了这个批量计算相关系数的工具,可以很便捷的计算多个基因的相关性,软件集成SangerBox中,他可以让你不需要编程即可实现相关系数的批量计算
谍照在此:
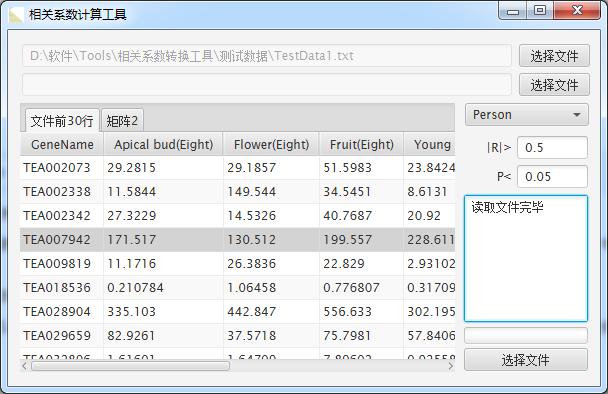
软件界面很简单
分三步:
1、选择数据矩阵
2、选择阈值和相关系数的方法
3、导出
我们的小工具一贯的思路就是简单明了
要使用小工具,首先我们得准备数据,需要记住的是 这款软件 是以行进行计算相关系数的,如果你传给他一个数据矩阵,他会计算每一行之间的相关性,两两计算,如案例1;如果你传给他的是两个数据矩阵,那么他会计算这两个数据矩阵中每一行之间的相关性如案例2
案例1:
比如我们有一个测试数据TestData1.txt
选择文件导入TestData1.txt如下图:

使用0.5和0.05作为相关系数阈值(这里根据你的实际需要选择阈值)
然后点击 选择文件 按钮,选择保存的目录,等待运行完成
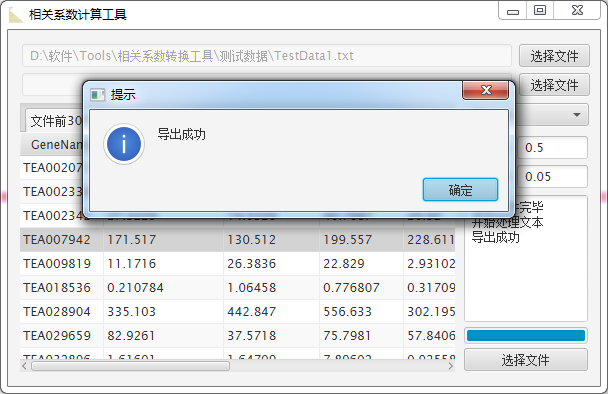
就得到了这个矩阵中每行之间的相关系数结果了,如图:
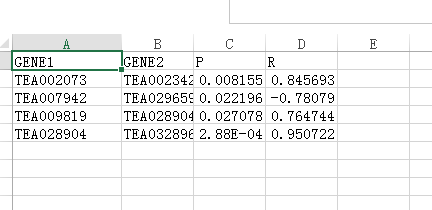
案例2:
有时候我们可能不仅仅只有一个数据文件,可能我们有两套数据,想看一下两套数据中元素的相关性,比如 想看一下miRNA和mRNA的表达相关性,这个时候我们有两个表达谱,所以此时我们可以将两个表达谱同时导入到软件中
首先导入矩阵1,如图
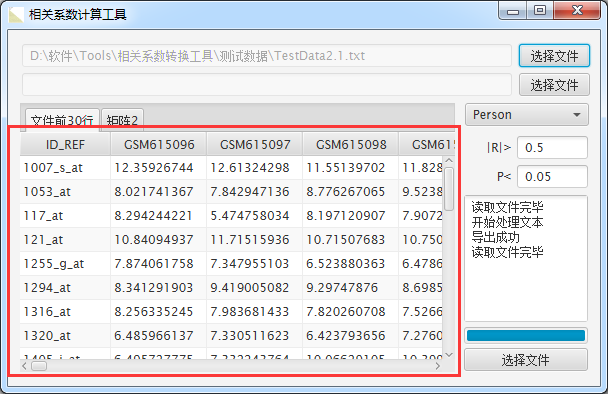 再导入矩阵2,如图
再导入矩阵2,如图
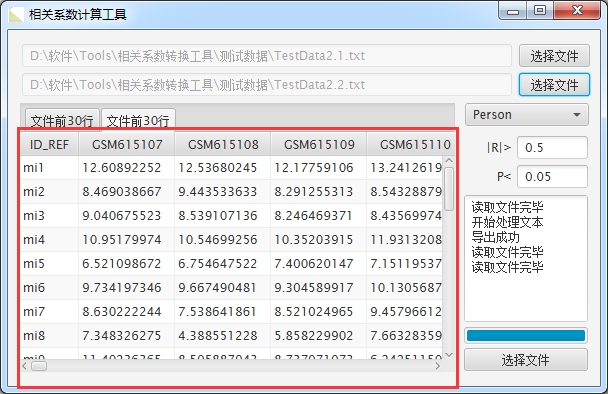
使用默认的参数,点击 选择文件 按钮 保存结果
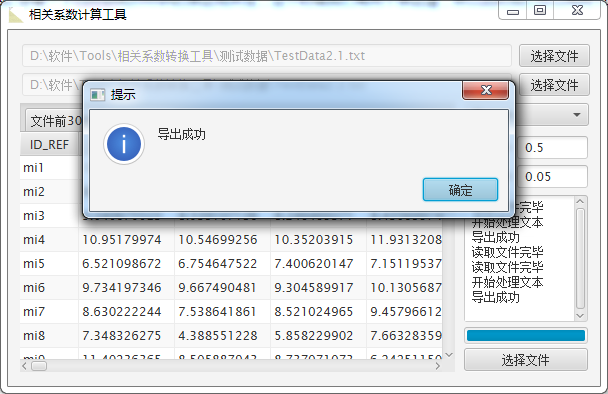
最终在目录下生成了三个文件,如图
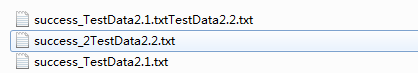
从中可以看出,其中有两个文件是咱们输入的表达谱,success_TestData2.1.txtTestData2.2.txt才是最终的相关系数结果如图:
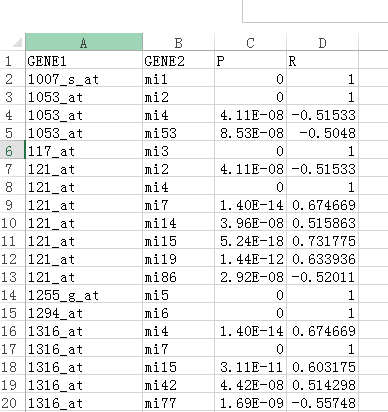
那么问题来了,这两个表达矩阵该怎么准备呢,其实这两个表达矩阵的表头要有共同的,软件是根据你导入的两个表达矩阵的表头,提取出共同的样本才能计算相关系数的
最后测试数据已经打包上传,下载在这里:一分钱下载(为了防止被人无限制下载拖垮网站,故设置一分钱下载)
- 发表于 2018-04-04 21:56
- 阅读 ( 15576 )
- 分类:软件工具
你可能感兴趣的文章
- 怎样使用SangerBox进行投稿选刊 5138 浏览
- 怎样使用Sangerbox对搜索结果进行统计 3392 浏览
- 怎样使用Sangerbox筛选目标杂志 3902 浏览
- 怎样使用Sangerbox进行文献分析 4838 浏览
- 怎样利用Sangerbox收藏文献,高效读文章 3429 浏览
- 怎么高效的使用Sangerbox查文献 3816 浏览
