RCricos绘制简单圈图
看教程不够直观,那就看视频吧! >>点击加载视频
RCricos绘制简单圈图
程序1:
library(RCircos)
#导入内建人类染色体数据
data(UCSC.HG38.Human.CytoBandIdeogram)
cyto.info <- UCSC.HG38.Human.CytoBandIdeogram
RCircos.Set.Core.Components(cyto.info, chr.exclude=NULL,tracks.inside=10, tracks.outside=0 )
#chr.exclude <- NULL; 设置不显示的染色体,如 c(1,3)
#cyto.info <- UCSC.HG19.Human.CytoBandIdeogram; 设置染色体数据
#tracks.inside <- 10; 设置内部track 个数
#tracks.outside <- 0; 设置外部track 个数
#设置外部track 个数
RCircos.Set.Plot.Area()
RCircos.Chromosome.Ideogram.Plot() #绘制染色体表意文字,染色体的默认方法亮点和染色体名称。
程序2:
#导入内建人类染色体数据
data(UCSC.HG19.Human.CytoBandIdeogram);
head(UCSC.HG19.Human.CytoBandIdeogram);
chr.exclude <- NULL; #设置不显示的染色体,如 c(1,3)
cyto.info <- UCSC.HG19.Human.CytoBandIdeogram; #设置染色体数据
tracks.inside <- 10; #设置内部track 个数
tracks.outside <- 0; #设置外部track 个数
#导入上面四个基本参数
RCircos.Set.Core.Components(cyto.info, chr.exclude, tracks.inside, tracks.outside);
#列出 所有绘图参数
RCircos.List.Parameters()
#Following are an example to change RCircos plot parameters:
#===========================================================
params <- RCircos.Get.Plot.Parameters(); #获得参数列表
params$radius.len <- 2.0; #更改其中的参数
params$base.per.unit <- 5000; #更改其中的参数
RCircos.Reset.Plot.Parameters(params) #参数重置
#定义要生成的图像文件,pdf,png,tiff 等 ,注意:最后要用dev.off()关闭
out.file <- "D:/RCircosDemoHumanGenome.tiff";
pdf(file=out.file, height=8, width=8, compress=TRUE);
RCircos.Set.Plot.Area();
#Plot Chromosome Ideogram
RCircos.Chromosome.Ideogram.Plot();
#Gene Labels and connectors on RCircos Plot
#RCircos.Gene.Connector.Plot 绘制染色体表意文字和基因名称之间的连接
#RCircos.Gene.Name.Plot 在数据轨道上绘制基因名称
data(RCircos.Gene.Label.Data);
name.col <- 4;
side <- "in";
track.num <- 1;
RCircos.Gene.Connector.Plot(RCircos.Gene.Label.Data, track.num, side);
track.num <- 2;
RCircos.Gene.Name.Plot(RCircos.Gene.Label.Data, name.col,track.num, side);
#Heatmap, Histogram, Line, Scatter, and Tile Plot
#Heatmap--------------
#RCircos.Heatmap.Plot绘制一个数据轨道的热图
data(RCircos.Heatmap.Data);
data.col <- 6;
track.num <- 5;
side <- "in";
RCircos.Heatmap.Plot(RCircos.Heatmap.Data, data.col, track.num, side);
#Scatter--------------
#RCircos.Scatter.Plot 一个数据轨迹的扫描图
data(RCircos.Scatter.Data);
data.col <- 5;
track.num <- 6;
side <- "in";
by.fold <- 1;
RCircos.Scatter.Plot(RCircos.Scatter.Data, data.col,track.num, side, by.fold);
#Line-----------------
#RCircos.Line.Plot绘制线为一个数据轨道
data(RCircos.Line.Data);
data.col <- 5;
track.num <- 7;
side <- "in";
RCircos.Line.Plot(RCircos.Line.Data, data.col, track.num, side);
#Histogram------------
#RCircos.Histogram.Plot 绘制一个数据轨迹的直方图
data(RCircos.Histogram.Data);
data.col <- 4;
track.num <- 8;
side <- "in";
RCircos.Histogram.Plot(RCircos.Histogram.Data, data.col, track.num, side);
# Tile-----------------
data(RCircos.Tile.Data);
track.num <- 9;
side <- "in";
RCircos.Tile.Plot(RCircos.Tile.Data, track.num, side);
#Links-----------------
#RCircos.Link.Plot绘制两个或多个基因组位置之间的链接线
data(RCircos.Link.Data);
track.num <- 11;
RCircos.Link.Plot(RCircos.Link.Data, track.num, TRUE);
data(RCircos.Ribbon.Data);
RCircos.Ribbon.Plot(ribbon.data=RCircos.Ribbon.Data, track.num=11, by.chromosome=FALSE, twist=FALSE);
dev.off();
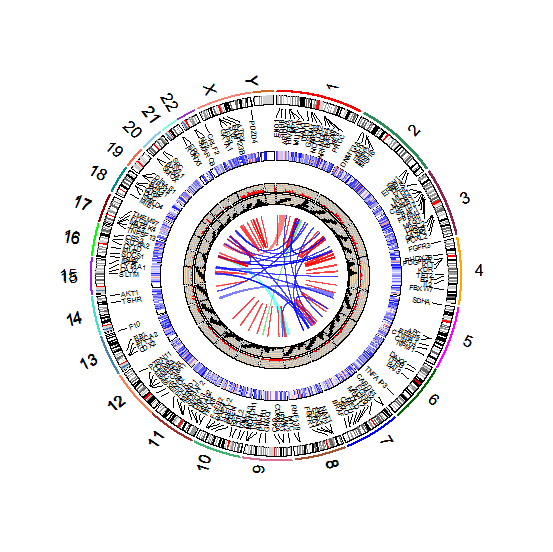
##由于R版本不同部分函数有差异
- 发表于 2017-05-20 10:25
- 阅读 ( 8001 )
- 分类:软件工具
