生信文献解读(2)What? 传统识别基因预后标志物的方法存在鲁棒性差,可移植性差的问题得到解决了?
看教程不够直观,那就看视频吧! >>点击加载视频
癌症作为一种复杂疾病,是多基因异常情况下,最终导致通路的失调,进而机体异常[1]。对预后相关基因的识别不仅可以提高对预后的判断能力,而且可以更好地理解癌症发生的潜在机制。同时,通过生物学通路去探索癌症发生、诊断以及预后的机制[2,3]。但是,一个通路包含的基因太多了,并不是所有的基因都是重要的基因,因此,从这些通路中进一步挖掘子通路,这些通路与单个基因一样具有预测性,但子通路在生物学背景下功能更强,更易于解释[4]。然而,目前传统识别基因预后标志物的方法存在鲁棒性差的问题,可移植性差。所以今天小编就和大家分享一篇关于癌症预后的文章【Briefings in Bioinformatics -- A novel high-frequency sub-pathway mining approach to identify robust prognostic gene signatures -- 发表时间:2019.7.27 --IF:6.72】来解决这个问题

 癌症作为一种复杂疾病,是多基因异常情况下,最终导致通路的失调,进而机体异常[1]。对预后相关基因的识别不仅可以提高对预后的判断能力,而且可以更好地理解癌症发生的潜在机制。同时,通过生物学通路去探索癌症发生、诊断以及预后的机制[2,3]。但是,一个通路包含的基因太多了,并不是所有的基因都是重要的基因,因此,从这些通路中进一步挖掘子通路,这些通路与单个基因一样具有预测性,但子通路在生物学背景下功能更强,更易于解释[4]。然而,目前传统识别基因预后标志物的方法存在鲁棒性差的问题,可移植性差。因此,针对这一现象,在2019年7月,Li等人将随机策略和子通路挖掘的方法相结合,开发出HiFreSP(high-frequency sub-pathways mining approach,高频子通路挖掘方法)算法去识别更鲁棒的癌症预后标志物,该方法对训练集噪声鲁棒,并能有效防止过拟合。并在ESCC(食管鳞状细胞癌),PAAD(胰腺癌)、BRCA(乳腺癌)以及TCGA数据库中包含20种癌症(n = 8137)的数据集中得到验证。其中,在ESCC疾病中,Li等人识别出了由6个基因(CCND1,CSF3R,E2F2,JUP,RARA和TCF7)构成的预后标志物。接下来,他们进一步将HiFreSP算法识别到的预后标志物与Lasso cox以及RSF(随机森林)预后模型识别到的标志物的预后能力进行比较,发现HiFreSP预后评估性能和鲁棒性最好。Li等人将HiFreSP算法思想的代码存放在GitHub上(https://github.com/chunquanlipathway/HiFreSP),以便感兴趣的研究员方便使用。
癌症作为一种复杂疾病,是多基因异常情况下,最终导致通路的失调,进而机体异常[1]。对预后相关基因的识别不仅可以提高对预后的判断能力,而且可以更好地理解癌症发生的潜在机制。同时,通过生物学通路去探索癌症发生、诊断以及预后的机制[2,3]。但是,一个通路包含的基因太多了,并不是所有的基因都是重要的基因,因此,从这些通路中进一步挖掘子通路,这些通路与单个基因一样具有预测性,但子通路在生物学背景下功能更强,更易于解释[4]。然而,目前传统识别基因预后标志物的方法存在鲁棒性差的问题,可移植性差。因此,针对这一现象,在2019年7月,Li等人将随机策略和子通路挖掘的方法相结合,开发出HiFreSP(high-frequency sub-pathways mining approach,高频子通路挖掘方法)算法去识别更鲁棒的癌症预后标志物,该方法对训练集噪声鲁棒,并能有效防止过拟合。并在ESCC(食管鳞状细胞癌),PAAD(胰腺癌)、BRCA(乳腺癌)以及TCGA数据库中包含20种癌症(n = 8137)的数据集中得到验证。其中,在ESCC疾病中,Li等人识别出了由6个基因(CCND1,CSF3R,E2F2,JUP,RARA和TCF7)构成的预后标志物。接下来,他们进一步将HiFreSP算法识别到的预后标志物与Lasso cox以及RSF(随机森林)预后模型识别到的标志物的预后能力进行比较,发现HiFreSP预后评估性能和鲁棒性最好。Li等人将HiFreSP算法思想的代码存放在GitHub上(https://github.com/chunquanlipathway/HiFreSP),以便感兴趣的研究员方便使用。

(1)训练集:ESCC芯片数据GSE121931(n = 125),对该数据通过RMA标准化和log2转化;
(2)测试集:额外的3套ESCC数据集、2套PAAD数据集、4套BRCA(ER+)数据集和TCGA数据库中20种癌症(n = 8137)数据集。
(3)HiFreSP算法思想,如Fig 1所示:
i.构造训练集
ii.识别预后相关的基因
iii.识别预后相关的通路
iv.计算高频基因(HFG)以及高频通路得分(HFP)
v.结合HFG和HFP识别预后相关子的通路
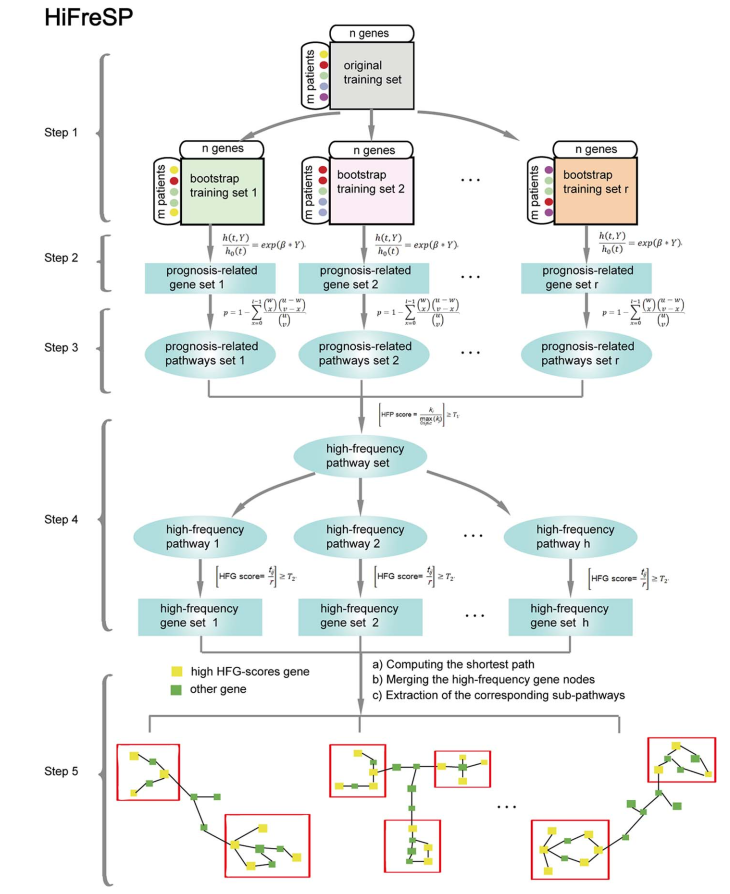 Fig 1. HiFreSP方法图
Fig 1. HiFreSP方法图 (1)在ESCC训练集中识别由19个基因构成的预后标志物:对GSE5362数据集,按照Fig 1的思想,首先循环5000(r=5000)次,每次挑选119个样本挑选构造训练集(基因数为17434)。在每一次循环中,都通过单因素cox回归模型找和预后相关的基因,接下来将预后相关基因注释到通路,识别预后相关通路,并计算每个候选基因的HFG(>0.05)和每个通路的HFP (>0.5),然后在每一个HFP中寻找HFG,从而得到相应的子通路(子通路基因数不能超过90)。最终,Li等人在GSE5362数据中识别了15条子通路,发现05200_24子通路中包含的19个基因log-rank p值最显著。并将这19个基因构成的模型在独立数据集中得到验证(如Fig 2)。
(1)在ESCC训练集中识别由19个基因构成的预后标志物:对GSE5362数据集,按照Fig 1的思想,首先循环5000(r=5000)次,每次挑选119个样本挑选构造训练集(基因数为17434)。在每一次循环中,都通过单因素cox回归模型找和预后相关的基因,接下来将预后相关基因注释到通路,识别预后相关通路,并计算每个候选基因的HFG(>0.05)和每个通路的HFP (>0.5),然后在每一个HFP中寻找HFG,从而得到相应的子通路(子通路基因数不能超过90)。最终,Li等人在GSE5362数据中识别了15条子通路,发现05200_24子通路中包含的19个基因log-rank p值最显著。并将这19个基因构成的模型在独立数据集中得到验证(如Fig 2)。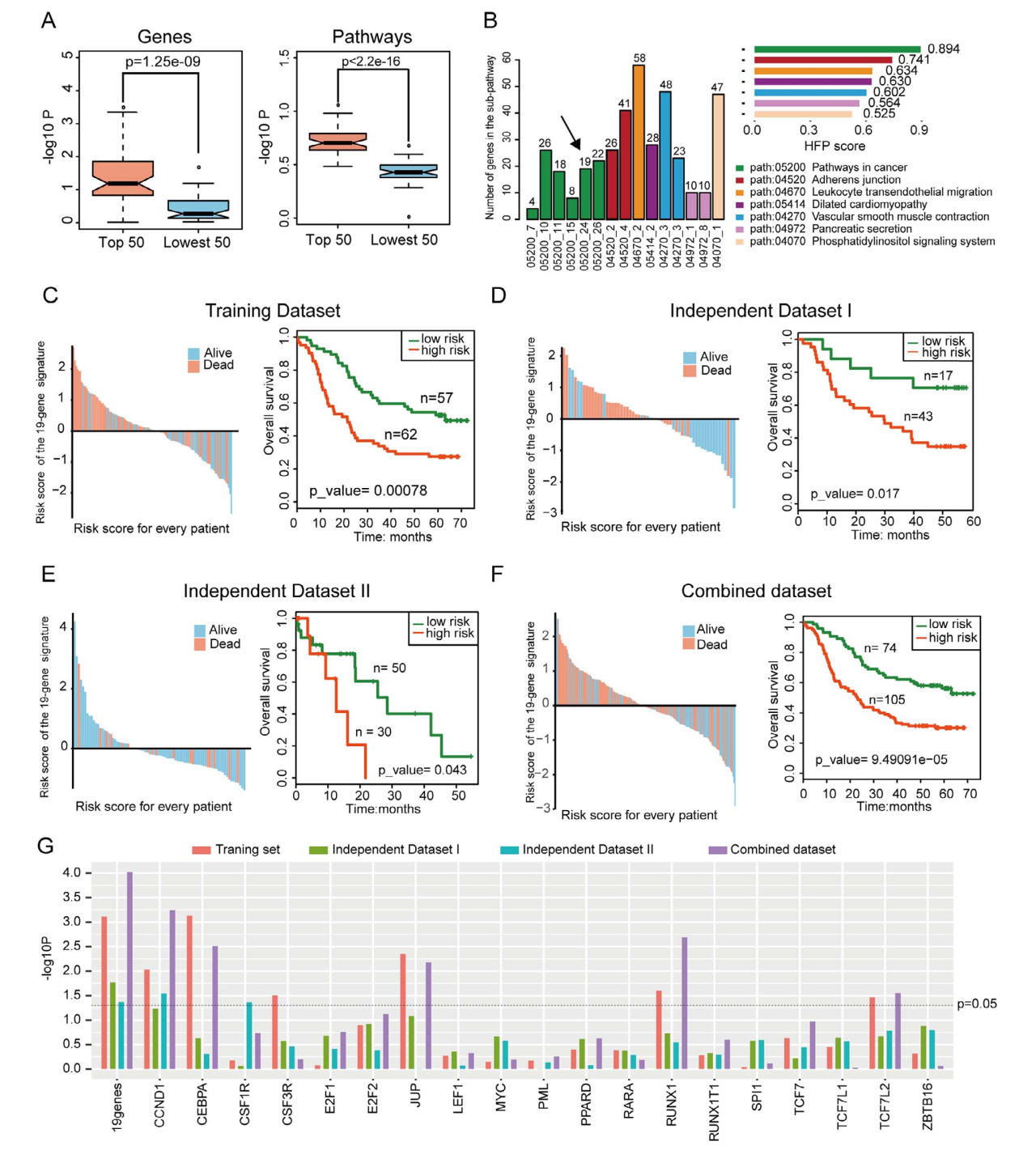 Fig 2.
19个基因的预后性能。A) Top50(高频)和Bottom50基因单因素cox
p值以及富集通路p值的条形图;B) 子通路;C)
19个基因构成的预后标记物在训练集预后性能的log-rank检验。D-F) 19个基因构成的预后标记物在测试集1、测试集2以及训练集和测试集1的混合数据集中预后性能的log-rank检验。G)
19个基因分别在训练集、测试集1、测试集2以及训练集和测试集1的混合数据集中预后评估展示。
Fig 2.
19个基因的预后性能。A) Top50(高频)和Bottom50基因单因素cox
p值以及富集通路p值的条形图;B) 子通路;C)
19个基因构成的预后标记物在训练集预后性能的log-rank检验。D-F) 19个基因构成的预后标记物在测试集1、测试集2以及训练集和测试集1的混合数据集中预后性能的log-rank检验。G)
19个基因分别在训练集、测试集1、测试集2以及训练集和测试集1的混合数据集中预后评估展示。(2)6个基因组成的基因标志物:通过组合的方法,一共有219-1种基因组合方式,通过评价每一个基因模型在训练集中的ROC值,从19个基因中挑选出由6个基因(CCND1,CSF3R,E2F2,JUP,RARA和TCF7)构成的基因集合,并在训练集以及测试集中去验证这个新的预后标志物模型的预后能力。(如Fig 3所示)
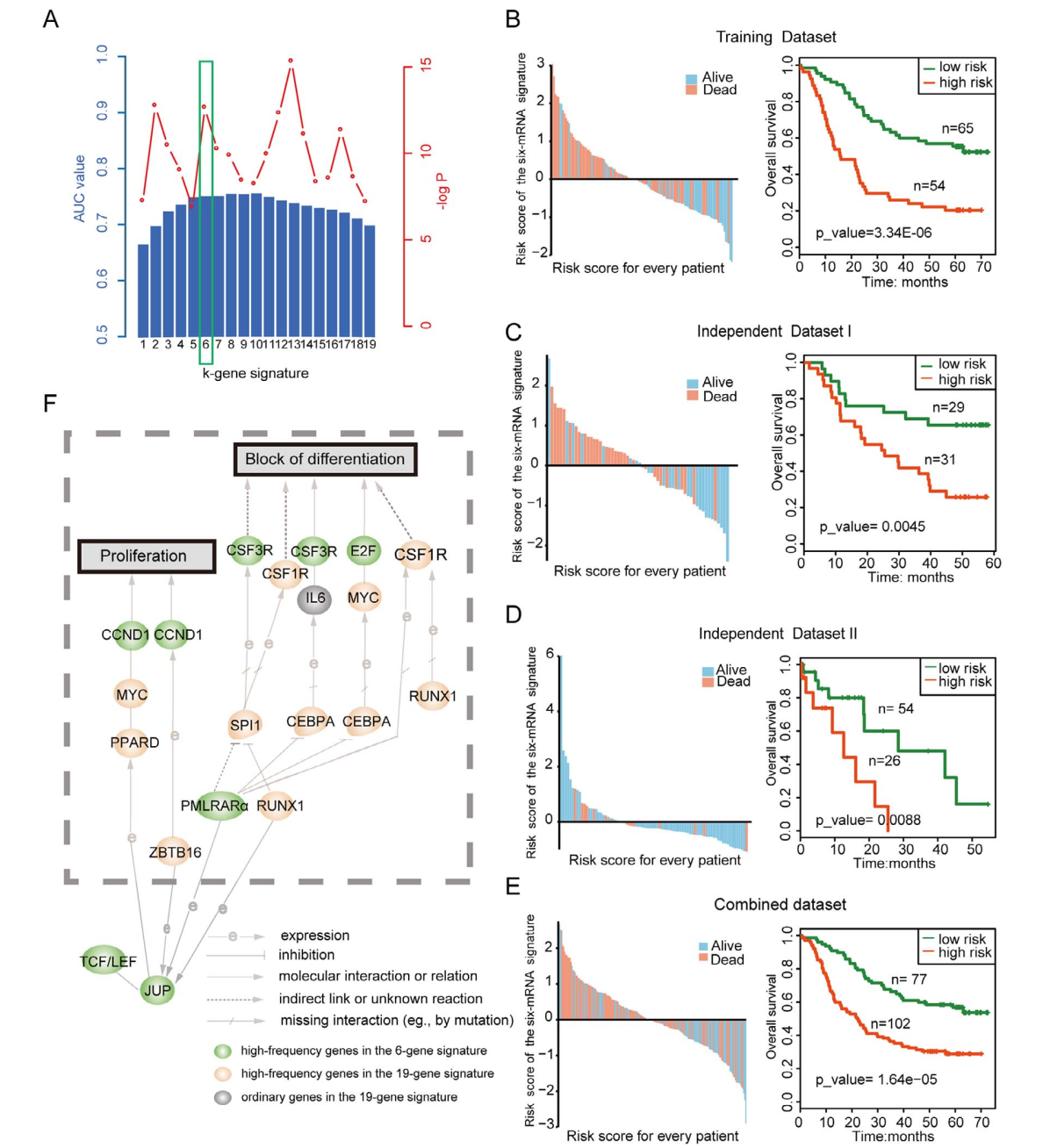 Fig 3. 6个基因构成的预后标志物模型。A) 挑选最佳基因组合;B-E) 6个基因分别在训练集、测试集1、测试集2以及训练集和测试集1的混合数据集的生存图;E) 子通路05200_24的功能可视化图。
Fig 3. 6个基因构成的预后标志物模型。A) 挑选最佳基因组合;B-E) 6个基因分别在训练集、测试集1、测试集2以及训练集和测试集1的混合数据集的生存图;E) 子通路05200_24的功能可视化图。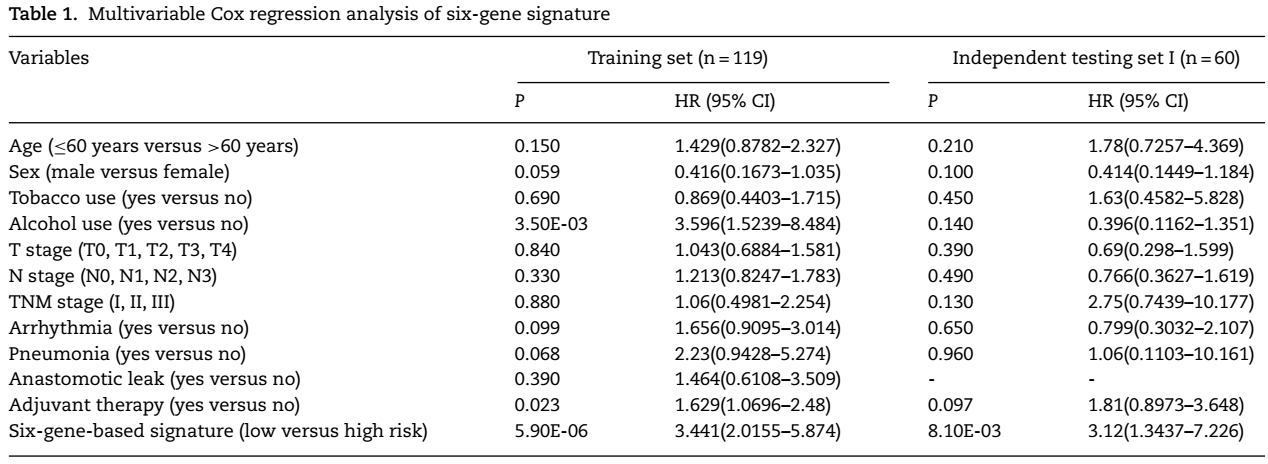
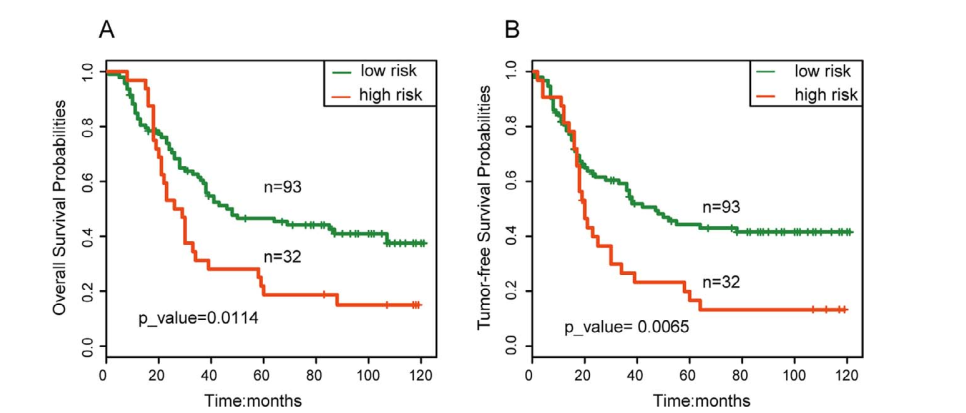 Fig 4. 6基因预后模型对训练集GSE121931高低风险组病人的生存曲线图。A) OS;B)TFS。
Fig 4. 6基因预后模型对训练集GSE121931高低风险组病人的生存曲线图。A) OS;B)TFS。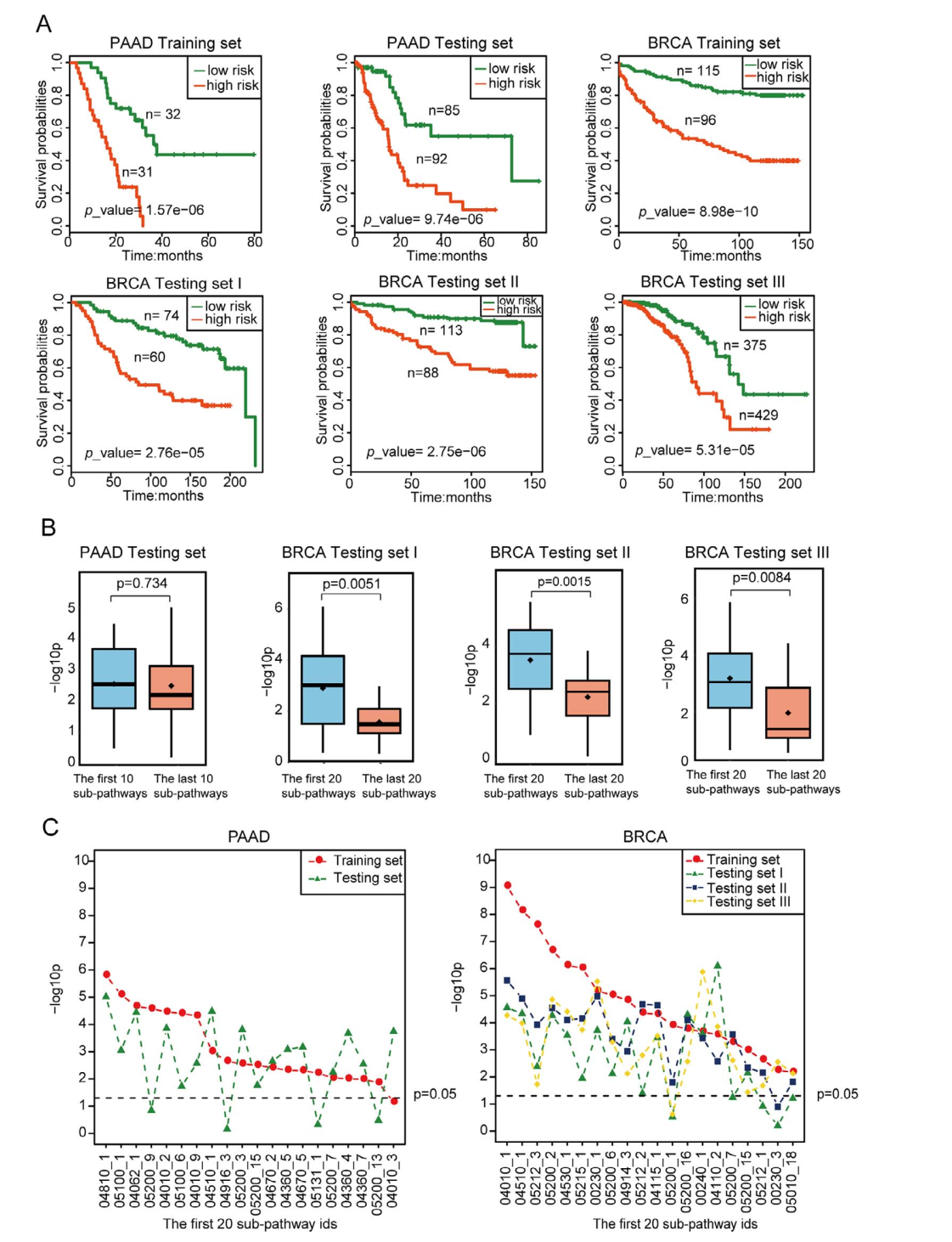 Fig 5. HiFreSP方法在PAAD以及BRCA疾病中的验证。
Fig 5. HiFreSP方法在PAAD以及BRCA疾病中的验证。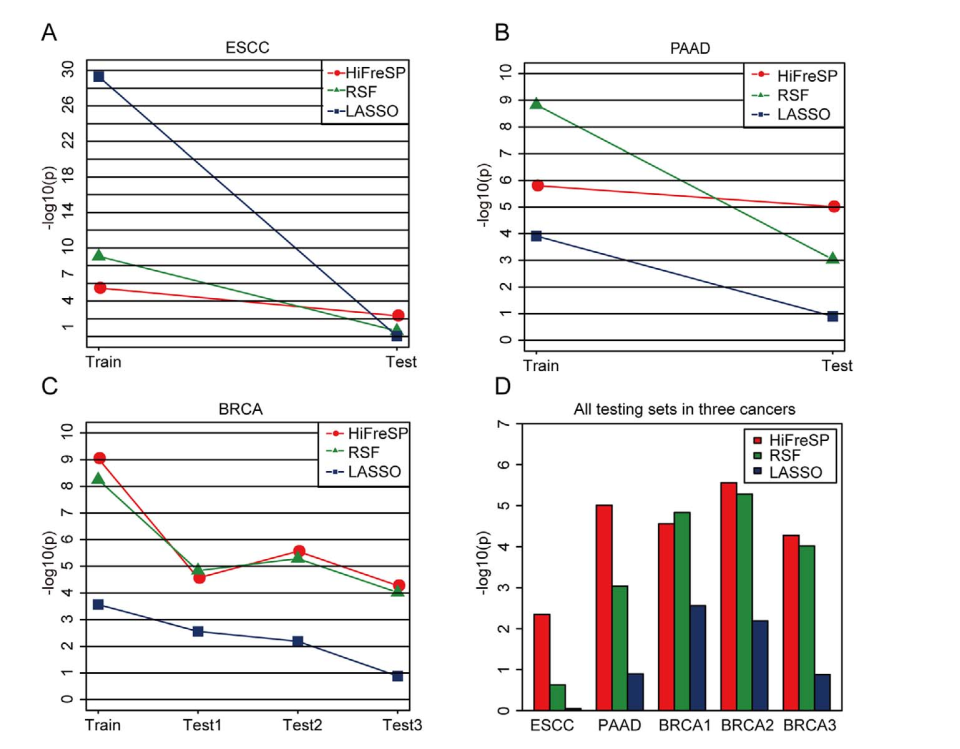
Fig 6. HiFreSP与LASSO和RSF识别预后标志物模型方法的比较。A) ESCC ;B) PAAD;C)BRCA;D) ESCC & PAAD & BRCA。
(6)在TCGA泛癌中对HiFreSP方法评估:将HiFreSP方法应用到TCGA数据库中的20种癌症中,对其识别出来的HFP进行排秩(如Fig 7),发现识别出来的通路大多和该疾病相关或者是癌症共有的特征通路,这个层面说明HiFreSP识别鲁棒的癌症相关子通路,这些通路在每种癌症中往往具有很好的预后预测能力。
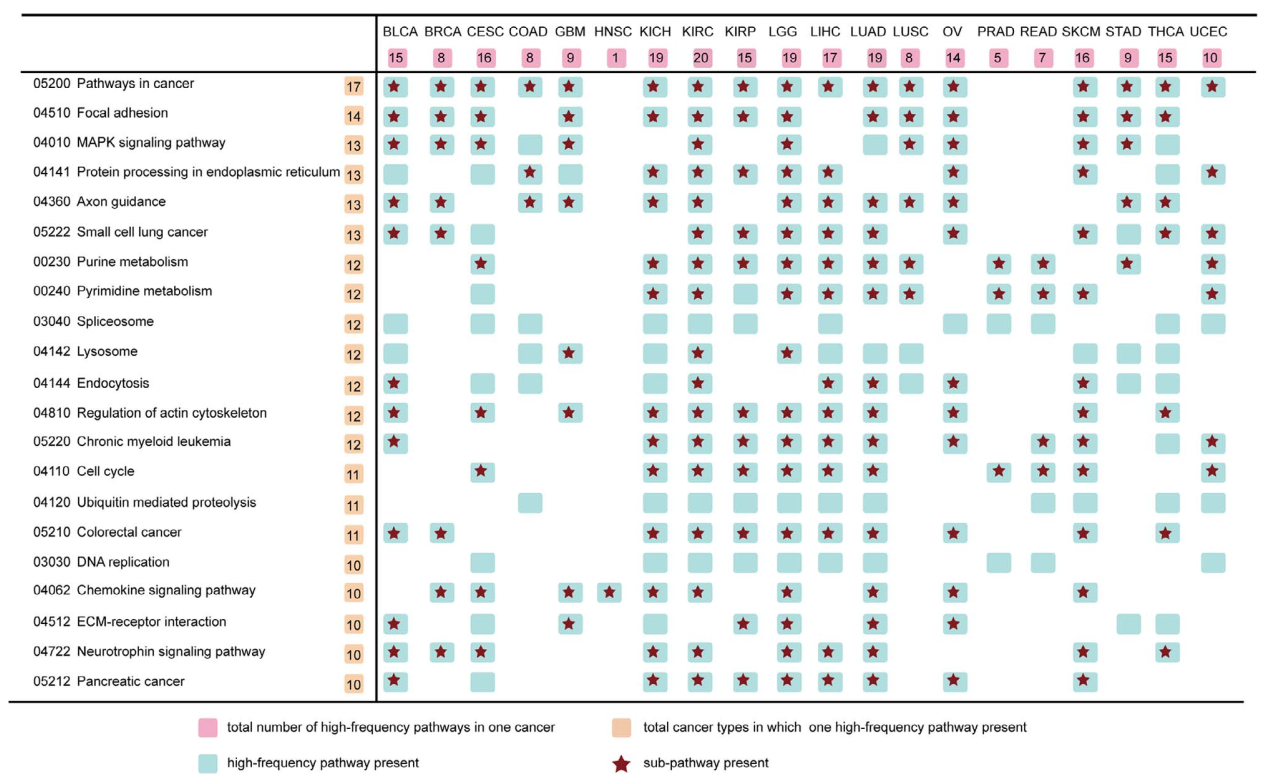 Fig 7. 在TCGA泛癌中对HiFreSP识别到的子通路。
Fig 7. 在TCGA泛癌中对HiFreSP识别到的子通路。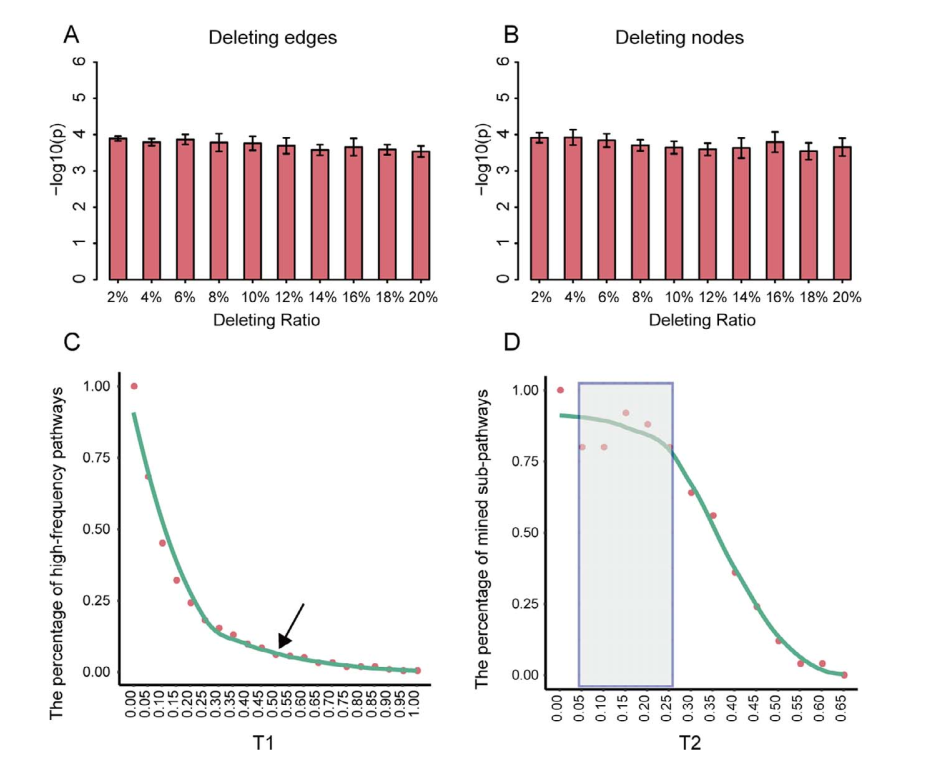 Fig 8. HiFreSP算法实现过程中相应参数随着边和点的缺失变化图
Fig 8. HiFreSP算法实现过程中相应参数随着边和点的缺失变化图
参考文献:
1. Cancer
Genome Atlas Research Network. Comprehensive
molecular characterization of clear cell renal cell carcinoma.
Nature 2013;499(7456):43–9.
2. Huang
S, Yee C, Ching T, et al. A novel model to combine
clinical and pathway-based transcriptomic information for
the prognosis prediction of breast cancer. PLoS Comput Biol
2014;10(9):e1003851.
3. Zhang C, Li C, Li J, et al. Identification of miRNA-mediated core gene module for glioma patient prediction by integrating high-throughput miRNA, mRNA expression and pathway structure. PLoS One 2014;9(5):e96908
4. Meng J, Li P, Zhang Q, et al. A four-long non-coding RNA signature in predicting breast cancer survival. J Exp Clin Cancer Res 2014;33(1):84.
 生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物
生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物
- 发表于 2019-08-26 13:34
- 阅读 ( 4125 )
- 分类:文献解读
