微生物多样性分析神器Qiime的centos 6.7的安装历程
QIIME (Quantitative Insights Into Microbial Ecology)是一个专门针对于微生物群落的分析pipline,可以进行OTU,以及多样性分析等等。拥有处理16s rRNA所需要的软件并呈现相应的处理结果。所谓分析pipline必是集成软件,QIIME也不例外。因此安装起来超级麻烦。
看教程不够直观,那就看视频吧! >>点击加载视频
QIIME (Quantitative Insights Into Microbial Ecology)是一个专门针对于微生物群落的分析pipline,可以进行OTU,以及多样性分析等等。拥有处理16s rRNA所需要的软件并呈现相应的处理结果。所谓分析pipline必是集成软件,QIIME也不例外。因此安装起来超级麻烦。
下面记录一下我的艰难历程:
首先根据官网安装教程进行安装,需要先安装miniconda 然后再使用命令安装
conda create -n qiime1 python=2.7 qiime matplotlib=1.4.3 mock nose -c bioconda
上述步骤非常顺利的安装完成。
随即按照官网测试命令跑一下走通了,兴高采烈的跑程序,建流程木有问题,除了usearch那玩意需要自己配置(看這里)之前,其他的都能够运行完,很easy。
但是,但是。。。。。。。作为处女座的一员总是感觉每次要用Qiime1之前需要输入这命令,感觉好不爽啊,如图:
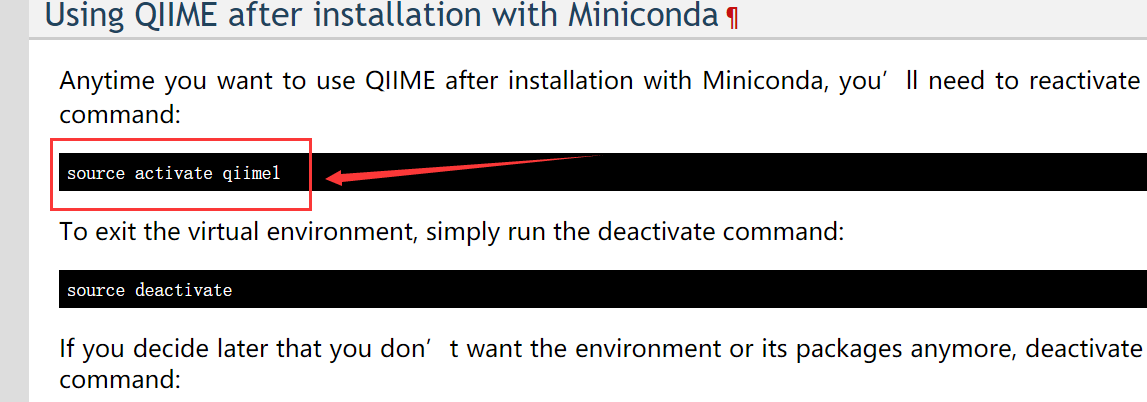
pip install qiime
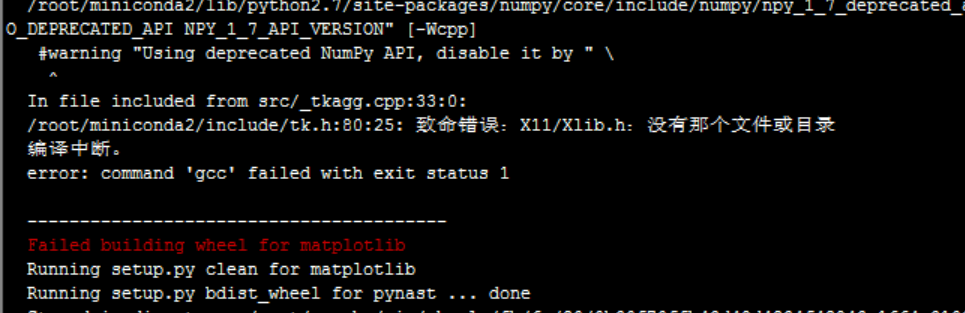
第一个过不去,问题如下
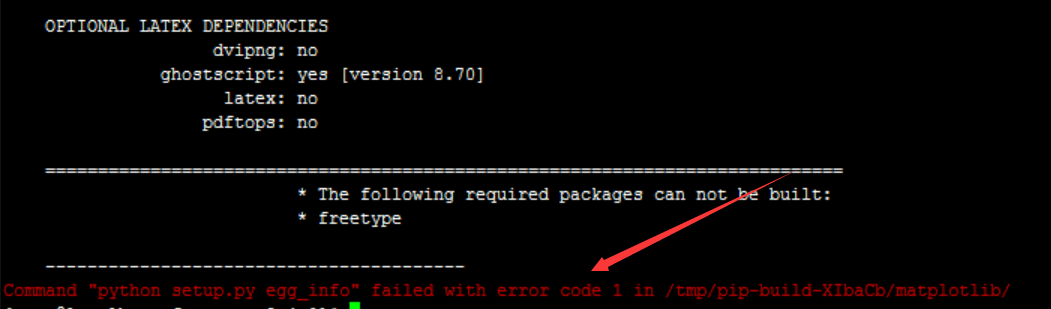
wget http://download.savannah.gnu.org/releases/freetype/freetype-2.4.0.tar.gz
tar -zxvf freetype-2.4.0.tar.gz
cd freetype-2.4.0
./configure
make && make install
make install时遇到问题:
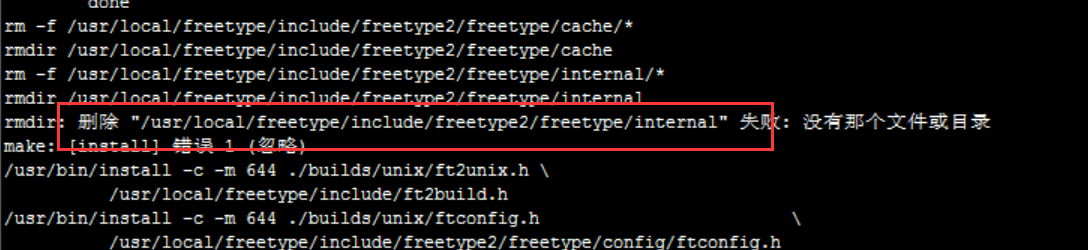
mkdir -p /usr/local/freetype/include/freetype2/freetype/internal
make install
继续 pip install qiime
又遇到问题:
仔细分析,应该是缺少libX11,运行:
yum install libX11-devel.i686
继续 pip install qiime
就這样成功了,迫不及待检测一下结果
- 发表于 2017-05-26 21:39
- 阅读 ( 7688 )
- 分类:软件工具
你可能感兴趣的文章
- 宏基因组扩增子最新分析流程QIIME2:官方中文帮助文档 7133 浏览
- 记录一下windows下安装python2.7包的艰苦经历 4246 浏览
相关问题
0 条评论
请先 登录 后评论