看代码,是y出了问题,你可以查看一下合并文件看是否出现了问题
您好,我是一个R语言的菜鸟,但这是我的毕设。我现在需要做乳腺癌miRNA的edgeR分析(无法使用小工具),看了您的代码,照着写的,可是遇到好多问题。想请教一下您!
源代码:
library("gplots")
library("limma")
library("edgeR")
foldChange=1
padj=0.01
setwd("F:\\大四下\\毕业课题设计\\miRNA-isoform数据集") #设置工作目录
rt<-read.table("F:\\大四下\\毕业课题设计\\miRNA-isoform数据集\\tcga_miRNA.txt",sep="\t",header=T,check.names=F) #改成自己的文件名
rt<-as.matrix(rt)
rownames(rt)=rt[,1]
exp<-rt[,2:ncol(rt)]
dimnames<-list(rownames(exp),colnames(exp))
data<-matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
data<-avereps(data) #重复的探针记录以均值替代
data<-data[rowMeans(data)>1,]
group=c(rep("Tumor",1077),rep("Normal",104)) #按照癌症和正常样品数目修改
#group<-c(rep("group2",nrow(dataupp)),rep("group3",nrow(datalow)))
design <- model.matrix(~group) #实验设计矩阵,以指示比对的方式
y <- DGEList(counts=data,group=group)#转化成R擅长处理的格式
y <- calcNormFactors(y) #标准化数据/归一化,创建标准化因子规范数据
y <- estimateCommonDisp(y) #先估计所有样品
y <- estimateTagwiseDisp(y) #再估计分组的
et <- exactTest(y,pair = c("Tumor","Normal")) #用exact Test计算p值
topTags(et) #通过p值或log-fold排序
ordered_tags <- topTags(et, n=100000)
错误:
> data<-matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
Error in matrix(as.numeric(as.matrix(exp)), nrow = nrow(exp), dimnames = dimnames) :
non-numeric matrix extent
In addition: Warning message:
In matrix(as.numeric(as.matrix(exp)), nrow = nrow(exp), dimnames = dimnames) :
NAs introduced by coercion
> data<-avereps(data) #重复的探针记录以均值替代
Warning messages:
1: In rowsum.default(x, ID, reorder = FALSE, na.rm = TRUE) :
missing values for 'group'
2: In rowsum.default(1L - is.na(x), ID, reorder = FALSE) :
missing values for 'group'
> data<-data[rowMeans(data)>1,]
然后我利用合并后的Merge_matrix.txt文件,出现的是这个错误
源代码:
library("gplots")
library("limma")
library("edgeR")
foldChange=1
padj=0.01
setwd("F:\\大四下\\毕业课题设计\\miRNA-isoform数据集") #设置工作目录
rt<-read.table("F:\\大四下\\毕业课题设计\\miRNA-isoform数据集\\Merge_matrix.txt",sep="\t",header=T,check.names=F) #改成自己的文件名
rt<-as.matrix(rt)
rownames(rt)=rt[,1]
exp<-rt[,2:ncol(rt)]
dimnames<-list(rownames(exp),colnames(exp))
data<-matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
data<-avereps(data) #重复的探针记录以均值替代
data<-data[rowMeans(data)>1,]
group=c(rep("Tumor",1077),rep("Normal",104)) #按照癌症和正常样品数目修改
#group<-c(rep("group2",nrow(dataupp)),rep("group3",nrow(datalow)))
design <- model.matrix(~group) #实验设计矩阵,以指示比对的方式
y <- DGEList(counts=data,group=group)#转化成R擅长处理的格式
y <- calcNormFactors(y) #标准化数据/归一化,创建标准化因子规范数据
y <- estimateCommonDisp(y) #先估计所有样品
y <- estimateTagwiseDisp(y) #再估计分组的
et <- exactTest(y,pair = c("Tumor","Normal")) #用exact Test计算p值
topTags(et) #通过p值或log-fold排序
ordered_tags <- topTags(et, n=100000)
错误:
> exp<-rt[,2:ncol(rt)]
> dimnames<-list(rownames(exp),colnames(exp))
> data<-matrix(as.numeric(as.matrix(exp)),nrow=nrow(exp),dimnames=dimnames)
Warning message:
In matrix(as.numeric(as.matrix(exp)), nrow = nrow(exp), dimnames = dimnames) :
NAs introduced by coercion
> data<-avereps(data) #重复的探针记录以均值替代
> data<-data[rowMeans(data)>1,]
>
>
> group=c(rep("Tumor",1077),rep("Normal",104)) #按照癌症和正常样品数目修改
> #group<-c(rep("group2",nrow(dataupp)),rep("group3",nrow(datalow)))
> design <- model.matrix(~group) #实验设计矩阵,以指示比对的方式
> y <- DGEList(counts=data,group=group)#转化成R擅长处理的格式
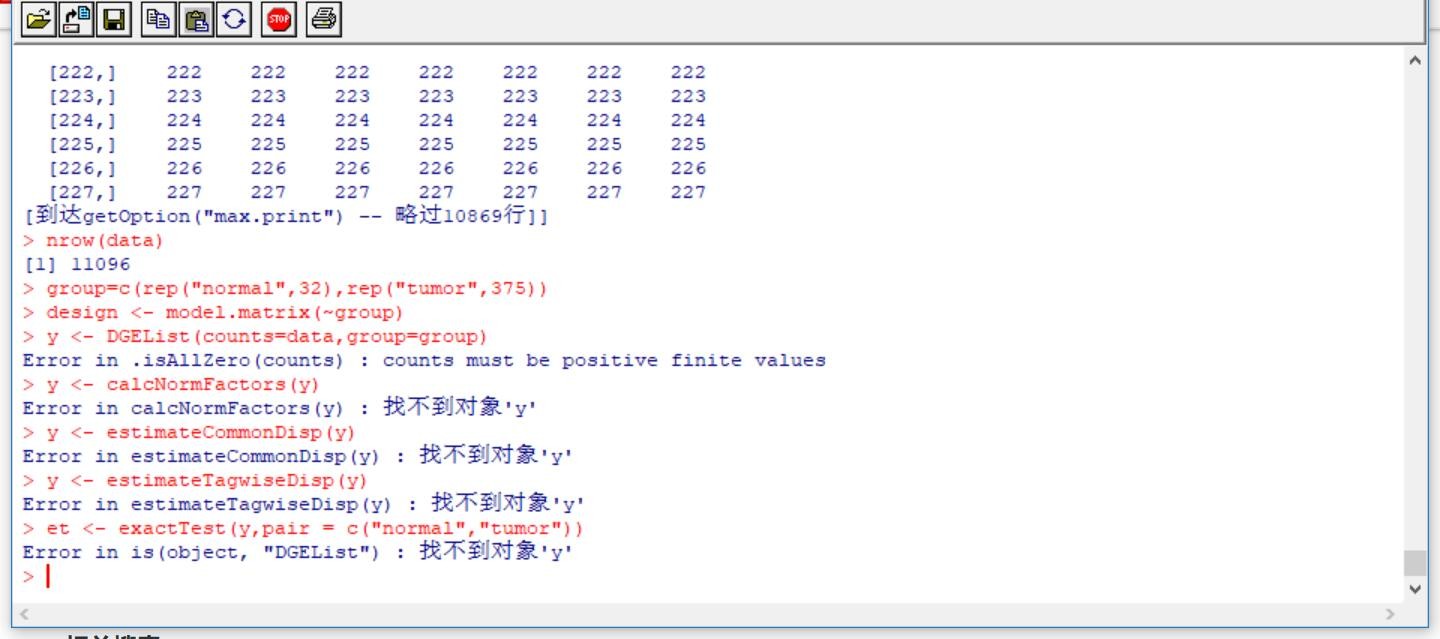
Error in .isAllZero(counts) : counts must be positive finite values
> y <- calcNormFactors(y) #标准化数据/归一化,创建标准化因子规范数据
Error in calcNormFactors(y) : object 'y' not found
> y <- estimateCommonDisp(y) #先估计所有样品
Error in estimateCommonDisp(y) : object 'y' not found
> y <- estimateTagwiseDisp(y) #再估计分组的
Error in estimateTagwiseDisp(y) : object 'y' not found
> et <- exactTest(y,pair = c("Tumor","Normal")) #用exact Test计算p值
Error in is(object, "DGEList") : object 'y' not found
> topTags(et) #通过p值或log-fold排序
Error in topTags(et) : object 'et' not found
> ordered_tags <- topTags(et, n=100000)
Error in topTags(et, n = 1e+05) : object 'et' not found
想请教请教您!拜托了
3 个回答
- 4 关注
- 0 收藏,16685 浏览
- duan 提出于 2018-03-23 17:23
 同问啊!!!!!!!!!!!!!!!!!!!!!!!@版主!!
同问啊!!!!!!!!!!!!!!!!!!!!!!!@版主!!