如何利用自己已发表的数据来再发一篇SCI
看教程不够直观,那就看视频吧! >>点击加载视频
话不多说,今天来安利一篇老少皆宜的数据挖掘的非编码RNA的“五分以上”文章,标题:Identification of a RNA-Seq based prognostic signature with five lncRNAs for lung squamous cell carcinoma,本文思路清晰,实现简单。

为了方便理解,就简单的将本文梳理一个流程图如下:
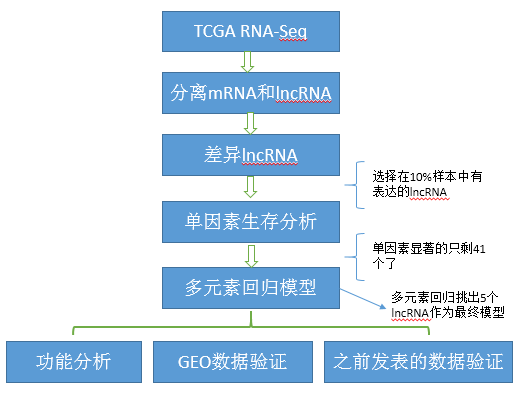
从流程图可以看出非常的简洁明了,是不是瞬间感觉自己也能做呢,那么下一步咱们一起来解读一下这篇文章的套路。
既然梳理了流程图那么咱们就直接看结果吧
1、首先文章共分离出了7589个lncRNA,事实上TCGA上的lncRNA比这个数多不少。
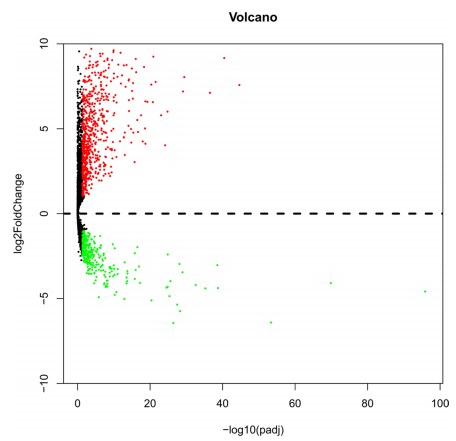
2、筛选差异表达的lncRNA,使用的R包是DEseq,共得到4225个差异的lncRNA,事实上lncRNA丰度都比较低,使用该包未必是最优的。
美美的画一张火山图,展示一下:
 3、开始对每一个差异的lncRNA做单因素生存分析,筛选出有预后差异的lncRNA,共得到41个lncRNA,不好展示直接用表格列出在补充材料中。
3、开始对每一个差异的lncRNA做单因素生存分析,筛选出有预后差异的lncRNA,共得到41个lncRNA,不好展示直接用表格列出在补充材料中。
4、将这41个lncRNA使用多因素生存回归分析,最终构建出5个lncRNA的生存模型,其实也是一条命令就完事的。

5、得到了五个lncRNA之后首先要做的是展示这五个lncRNA的各方面信息,比如在染色体位置,预后的显著性等等,让大家知道这五个lncRNA的基本情况
 6、既然构建了这五个lncRNA的预后模型,那么要看看不同风险分数(PI)下他们的一个表达和预后情况,为之后的样本分类做好准备
6、既然构建了这五个lncRNA的预后模型,那么要看看不同风险分数(PI)下他们的一个表达和预后情况,为之后的样本分类做好准备
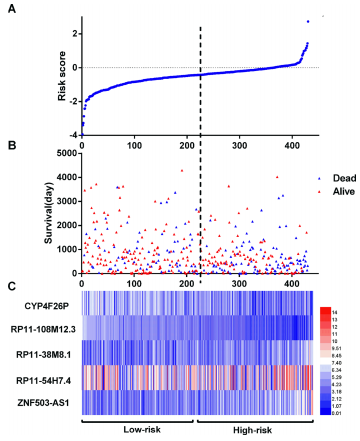 这个图呢可以直观的看出五个lncRNA构建的预后模型中不同样本的风险得分以及表达水平上的对应关系,从图中可以看出随着风险得分的增高样本的死亡时间有所加快(图B),而表达情况前两个lncRNA有下降趋势,后三个有上升趋势(不能做个zscore么。。。这好不明显。。。),这就说明三个问题:
这个图呢可以直观的看出五个lncRNA构建的预后模型中不同样本的风险得分以及表达水平上的对应关系,从图中可以看出随着风险得分的增高样本的死亡时间有所加快(图B),而表达情况前两个lncRNA有下降趋势,后三个有上升趋势(不能做个zscore么。。。这好不明显。。。),这就说明三个问题:
一、风险得分越高,预后越差
二、前两个lncRNA表达越低预后越差
三、后三个表达越高预后越差
那么这个图是怎么做的呢,其实很简单,三个图横轴都是样本,按照风险得分进行排序,第一个就是散点图,第二个也是(将死亡的标记成红色),第三个是热图,三个图组合一下就完事,实在不行一个一个画,画完之后用AI拼一下就行了
7、既然构建了这五个lncRNA的预后模型了,那么要对比一下这个模型与现有的病理学分类有啥区别,最终发现这个模型比他们的要好一些(这就是这篇文章的全部意义。。。)。
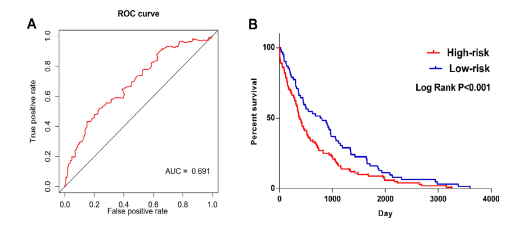
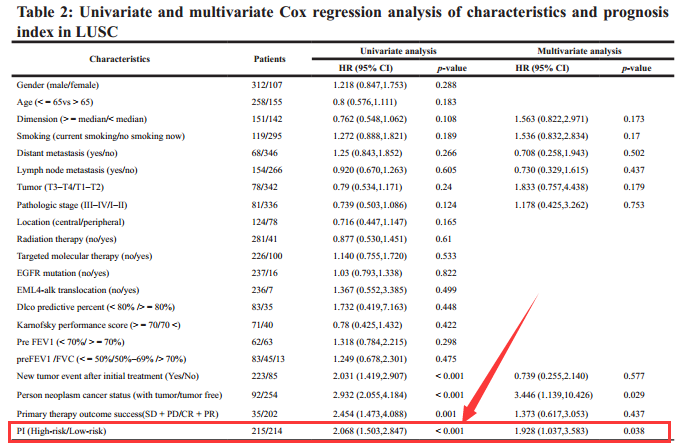 8、既然模型公式有了,那么选择一个好用的阈值来分类是必不可少的,这里使用ROC来评判模型的好坏顺便选择一个最优的阈值(A图中对应y轴-x轴最大那个点)
8、既然模型公式有了,那么选择一个好用的阈值来分类是必不可少的,这里使用ROC来评判模型的好坏顺便选择一个最优的阈值(A图中对应y轴-x轴最大那个点)
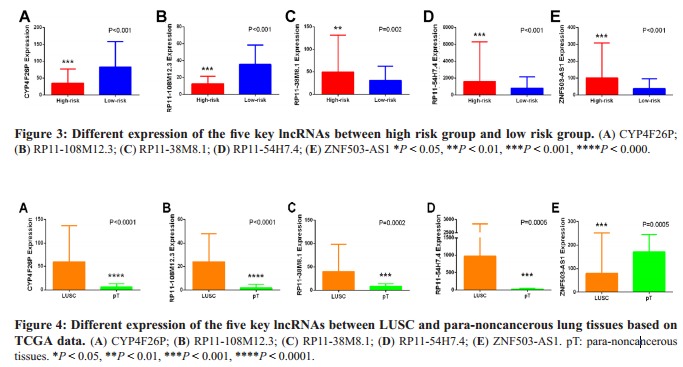
9、找到最优的阈值之后对样本分类然后对比一下五个lncRNA的表达情况,图一定要好看
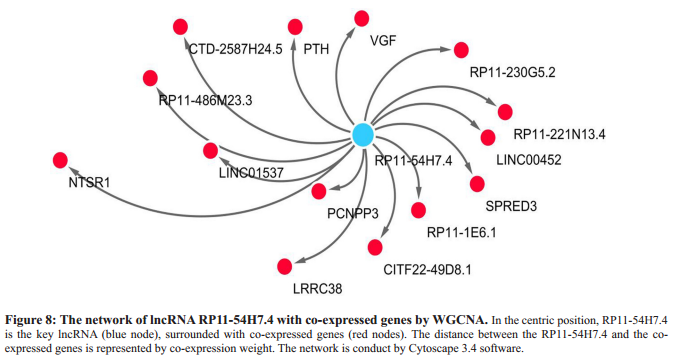
10、开始看看这五个基因的功能,使用WGCNA构建共表达网络,没看懂这一步为了说明什么,找到这五个lncRNA中最关键的lncRNA-RP11-54H7.4,我猜应该是,但是为什么不跟基因表达一起构建呢,这样更有利于说明这个lncRNA的功能喂。
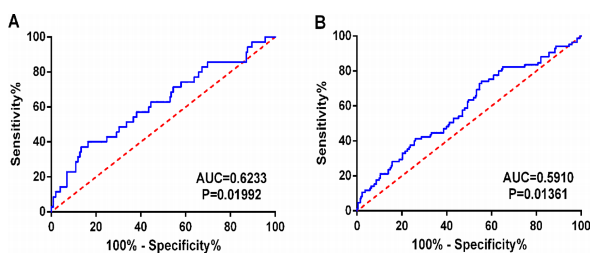
 11、找各种其他数据集验证比如找了好多套GEO的来验证一下预后,还结合之前课题组之前发的一篇文章中的lncRNA数据来验证了一下差异表达。
11、找各种其他数据集验证比如找了好多套GEO的来验证一下预后,还结合之前课题组之前发的一篇文章中的lncRNA数据来验证了一下差异表达。
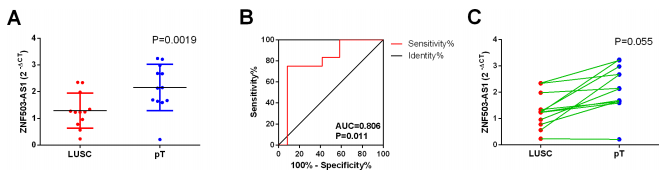
至此文章就结束了,虽然后面各种图乱入,但是无外乎就是本文前面的那个流程图,后面的主要亮点在于其他数据集也能 验证的出来这几个lncRNA确实差异表达,同时也跟预后有关。
- 发表于 2017-07-01 17:09
- 阅读 ( 11577 )
- 分类:文献解读
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9526 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10548 浏览
- TCGA数据库 7788 浏览
- TCGA下载工具超级版,在线秒下载 7565 浏览
- 在GEO数据库如何找带有预后信息的数据?一分钟搞定,适用于找任何的样本特征数据 20985 浏览
- 生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物 10091 浏览
