批量计算生存分析工具使用例子
以下我们使用GEO的一套数据来演示以下该工具使用
首先我们选择下载GSE25065数据集,下载之后使用GEO芯片数据转换器将数据提取出来,最终我们得到了这两个文件
打开SampleInfo.xls文件找到随访...
看教程不够直观,那就看视频吧! >>点击加载视频
工具下载链接:http://gap.shengxin.ren/tool/7/
以下我们使用GEO的一套数据来演示以下该工具使用
首先我们选择下载GSE25065数据集,下载之后使用GEO芯片数据转换器将数据提取出来,最终我们得到了这两个文件
 打开SampleInfo.xls文件找到随访信息列:
打开SampleInfo.xls文件找到随访信息列:
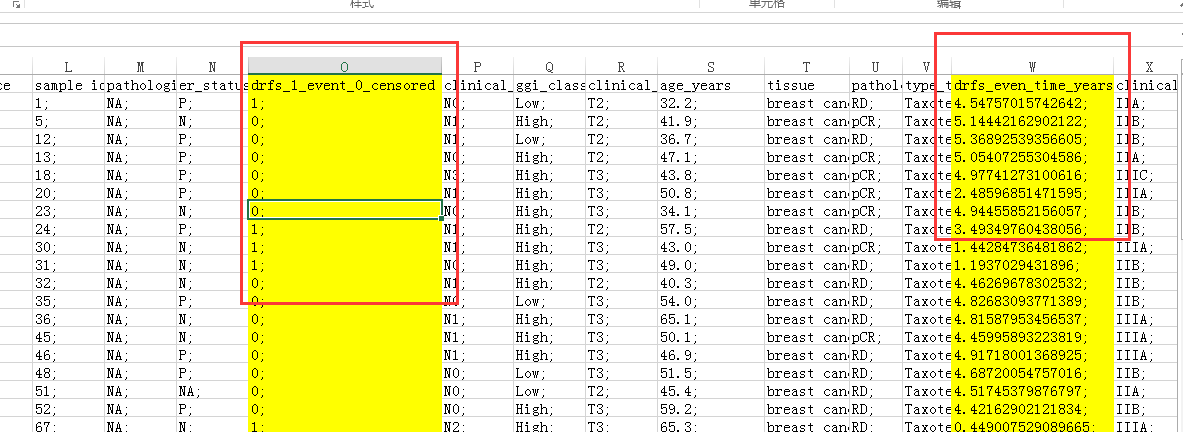
去除其中的分号,保存
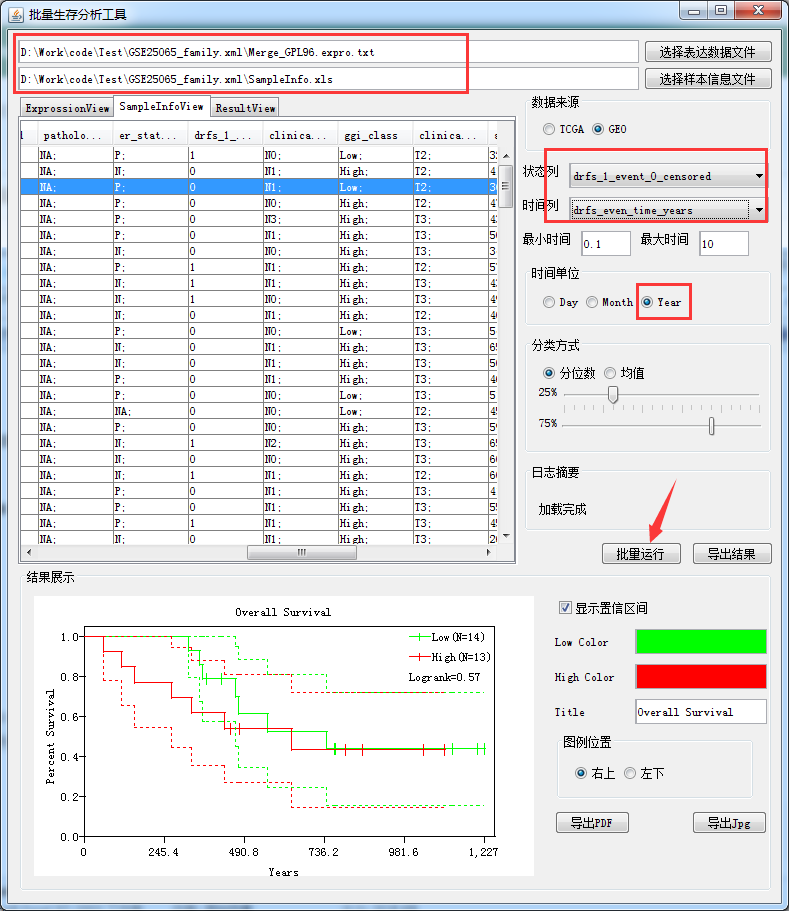
然后启动本工具,导入样本信息和表达谱数据,如图:
 最终得到结果:
最终得到结果:
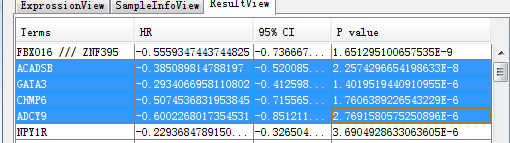
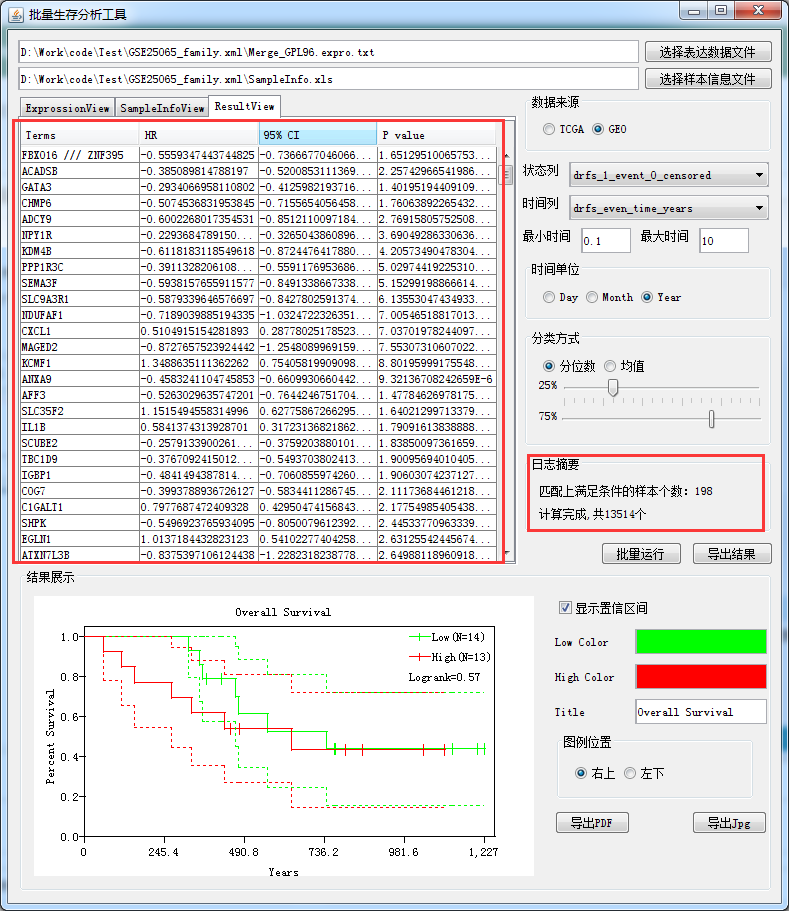 然后导出就OK了,结果中包括95%的置信区间,以及风险比,还有p值,使用方法是CoxRegression
然后导出就OK了,结果中包括95%的置信区间,以及风险比,还有p值,使用方法是CoxRegression
我们导出结果:
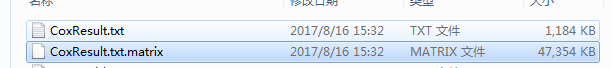 结果包含两个文件,第一个是每个基因的Cox结果,第二个是样本的表达谱和对应的临床随访信息整合的表格,可以自己导入到R里面去分析
结果包含两个文件,第一个是每个基因的Cox结果,第二个是样本的表达谱和对应的临床随访信息整合的表格,可以自己导入到R里面去分析
进一步的如果想观察每个基因的K-M曲线,该工具提供了K-M绘制曲线的功能,可以直观的看出分类效果,并提供导出pdf功能
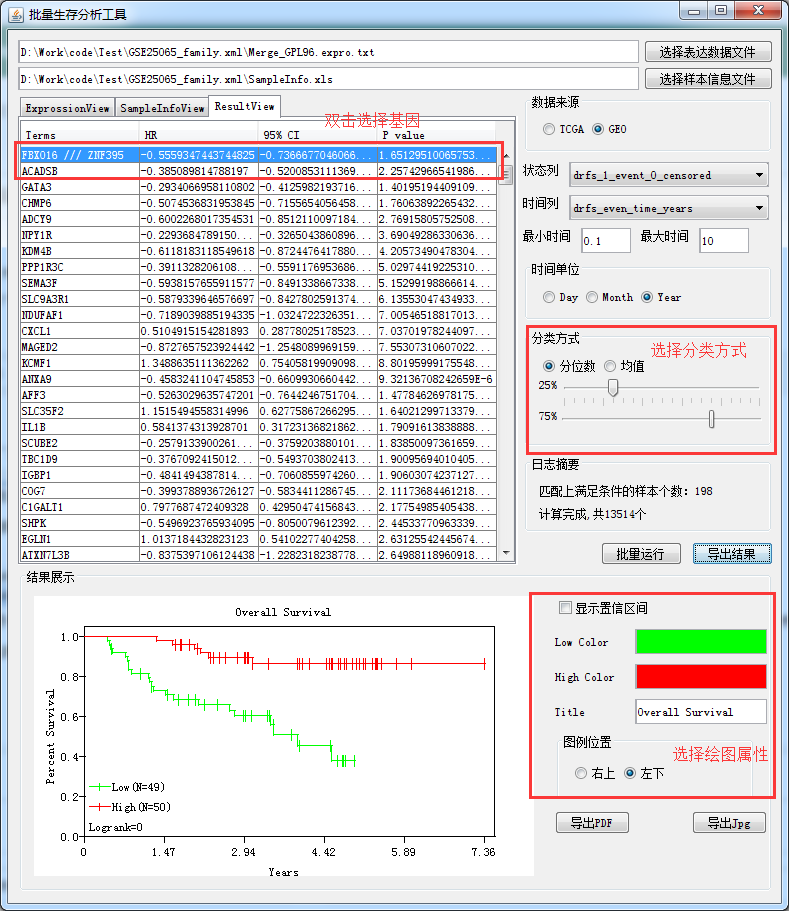
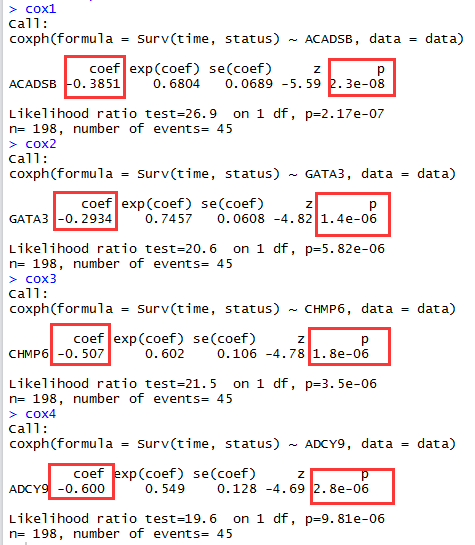
与R语言相比如下:
library(survival)
setwd("D:/Work/code/Test/GSE25065_family.xml")
data=read.csv('CoxResult.txt.matrix',sep = '\t',row.names = 1)
head(data)
time=data[,1]
status=data[,2]
cox1=coxph(Surv(time, status) ~ ACADSB,data)
cox2=coxph(Surv(time, status) ~ GATA3,data)
cox3=coxph(Surv(time, status) ~ CHMP6,data)
cox4=coxph(Surv(time, status) ~ ADCY9,data)
- 发表于 2017-08-16 15:39
- 阅读 ( 16012 )
- 分类:软件工具
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9662 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10614 浏览
- 多因素生存分析工具 5088 浏览
- 一键式的批量生存分析工具 4874 浏览
- KaplanMeier生存曲线绘制工具 11272 浏览
- TCGA数据库 7836 浏览
53 条评论
请先 登录 后评论
