植物MWAS研究—谷子产量与微生物组关联分析
看教程不够直观,那就看视频吧! >>点击加载视频
本文内容首发植物微生物组公众号,更多阅读欢迎访问植物微生物组公众号
MWAS简介
微生物组关联分析(Microbiome/Metagenome-wide association studies , MWAS)是指捕获多维尺度上的互作作用,从而提供捕获复杂作用关系的方法,该方法切实可行的预测微生物组和疾病状态的关系。就是不做任何假设的分析整个微生物组的结构变化,目的是鉴定与疾病、表型相关的微生物物种、基因或代谢物质等。全微生物组关联分析 (MWAS) 与全基因组关联分析 (GWAS) 有些类似,都是把某些复杂的特征(如物种、基因)与表型特征关联起来。
国内最早研究肠道菌群的学者赵立平教授于2013年首次在Nature Reviews Microbiology期刊上提出了微生物组关联分析 (Microbiome-wide association studies ,MWAS)的概念和研究策略。
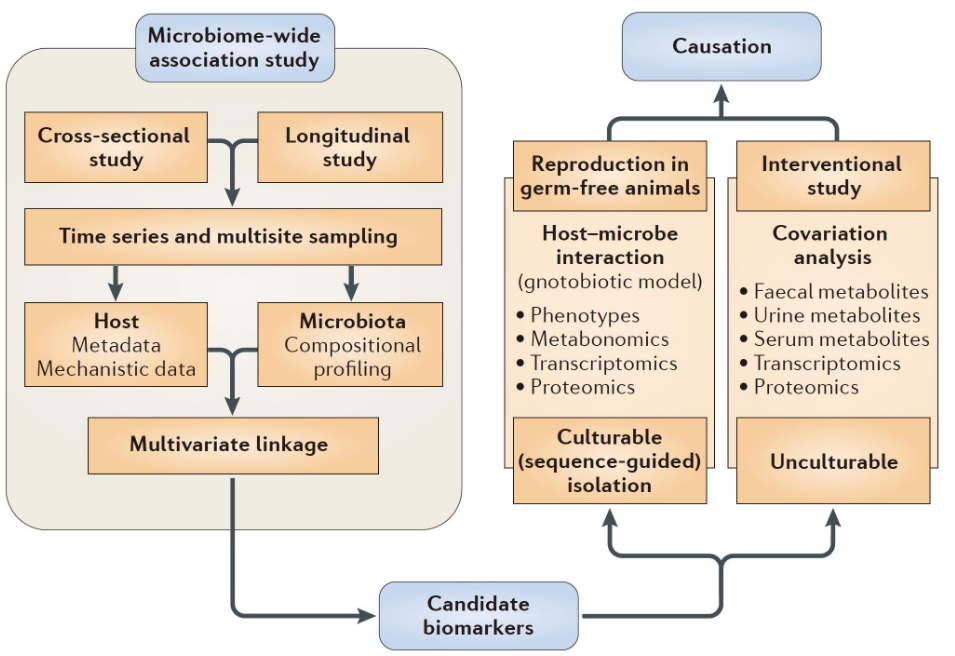
微生物组关联分析的研究策略(Figure 1 of Zhao LP, 2013, Reviews Microbiology)
这篇文章发布的4年后,此类研究如雨后春笋班的发表在各大顶级期刊上,方法也有了很大改进。最新的综述是2016年末王俊的 Nature Reviews Microbiology 和 Jack Gilbert的Nature两篇重镑综述,系统的概述了近三年的研究成功,而且也对现有研究方法和末来趋势进行了总结与展望,开展此领域研究必读。
之前的研究主要集中在人类疾病领域,那在农作物领域可以按这个套路搞吗?答案是当然可以,下面这篇文章就是在作物领域开展的MWAS研究。
2017年9月5日,GIGA SCIENCE杂志在线发表了华大基因-青岛 金桃团队关于我国特色作物-谷子微生物组关联分析的结果,中科院遗传发育所、佛罗里达大学、中科院南京土壤所等多家单位共同参与本研究。
本研究的发表,为揭示微生物组在产量中的应用指明的道路,同时将引领植物领域开展各物种的大规模微生物组关联分析。
研究背景
根表是宿主、土壤、微生物最错综复杂的互作环境。根系微生物(根际、根表、根内)主要来源于土壤,并受地理位置、营养状态和宿主基因型影响。微生物在植物产量、营养吸收和抗病中有重要作用。多个研究表明,宿主可以通过外排、根际沉积等方法选择根际有益微生物,同时改变土壤pH值、结构等。然而,根系微生物影响植物生长与产量中的作用仍然末知。因此,采用广泛的关联分析是鉴定农作物根系有益、有害微生物的有效手段。
谷子(也称小米、粟,英文Foxtail millet,拉丁名Setaria italica)由于具有耐旱和水分高效利用的特性,是干旱和半干旱地区重要的粮食作物。因此,理解谷子遗传和环境因素对产量的影响非常重要。目前深圳华大国家基因库已经有2540份谷子品种的表型和基因组数据。这些材料对于深入理解根系微生物如何影响谷子的生长、发育和产量至关重要。
摘要
背景:根系微生物组在植物产量、营养吸收和抗病中有重要作用。在作物和模式植物中,采用扩增子或宏基因组技术,已经对根系微生物的群落结构进行了广泛的研究。然而,根系微生物与宿主植物生长的功能是否相关仍然未知。本研究针对谷子根系微生物群体与产量性状进行关联分析。
发现:我们采用16S扩增子技术,对张家口和杨淩的2882份谷子样品的根表、根际土和土壤进行分析。鉴定了16109个OTUs,其中187个为根表的核心OTUs。Beta多样性分析显示,微生境(microhabitat)在微生物群落影响最大,其次是地理位置。大规模关联分析鉴定了与高产相关的潜在有益菌。此外,功能预测显示了谷子根表细菌群体有特异代谢通路。
结论:我们系统的描述了谷子根系细菌群落结构,并发现了根表核心细菌组分。结果表明宿主植物在根表富集特异的细菌和功能。这些潜在的有益菌将可能作为生物肥料应用于农业发展。
主是发现和结论揭示谷子根际微生物群落结构

图1. 谷子根和土壤细菌主要菌门。杨凌(YL)、张家口(ZJK)、非根际土(BS)、根际(RS)、根表(RP)
Fig.1 Dominant bacterial phyla detected in foxtail millet root compartments and bulk soils. YL: Yangling; ZJK: Zhangjiakou; BS: bulk soil; RS: rhizosphere; RP: rhizoplane.
微生境和地理位置对微生物群落影响较大

图2. 采用主坐标轴分析谷子根微生物组间Beta多样性。距离矩阵分别采用权重(a)和非权重Unifrac方法。结果表明微生境是组间最大差异;不同地理位置是第二大差异因素。
Fig.2 Principal coordinate analysis of foxtail millet root microbiota based on weighted (a) and unweighted (b) Unifrac matrices indicating that microhabitat is the largest separation factor (PCoA1) and geographic location is the second (PCoA2).YL: Yangling; ZJK: Zhangjiakou; BS: bulk soil; RS: rhizosphere; RP: rhizoplane.
鉴定不同地区根表共有和特异微生物

图3. 根表核心微生物组的样式。(a) 韦恩图展示两地点根表共有和特有OTUs。(b) 共有和特有OTUs的目水平物种注释分类统计。
Fig.3 Characterization of the core rhizoplane microbiota. (a) Venn diagram depicting the number of shared and specific OTUs of the core rhizoplane OTUs from both fields.(b)Taxonomic distribution of the core rhizoplane OTUs at the order level. The shared core rhizoplane OTUs from YL and ZJK were colored with brown, the specific core OTUs from YL and ZJK were colored with red and blue, respectively. YL: Yangling; ZJK: Zhangjiakou; BS: bulk soil; RS: rhizosphere; RP: rhizoplane.
根表微生物与健康状态相关

图4. 杨凌谷子的微生物中存在与健康状况相关的标志(marker) OTUs 。 (a) 采用随机森林模型对根表OTUs与产量进行回归分析。预测得到75个标志OTUs与产量相关。(b) 热图结合层级聚类展示标志OTUs的平均丰度。
Fig.4 Bacterial marker OTUs correlated with foxtail millet general health in Yangling. (a) A random forest model was applied to regress the rhizoplane bacterial OTUs against grain weight. Five repeats of 10-fold cross-validation in the training set (n=709) resulted in 75 marker OTUs to predict grain weight of foxtail millet. (b) Heatmap and hierarchical clustering of the mean of marker OTUs. relative abundance against grain weight. The samples were grouped according to grain weight (range from 1-30g), such as grain weight per plant less than 2 gram was classified into the first group, less than 3 gram for the second group and so on. The left heat map was plotted with training set samples and the right from the test set samples. OTUs negatively and positively correlated to the yield were marked with different color. Negative: red; positive: green.
鉴定根表与产量相关微生物网络

图5. 谷子根表产量相关标志OTUs的共丰度网络。左侧为负相关,右侧为正相关。边的颜色表示不同的相关系数。点的文字为科注释,颜色 为门注释。
Fig.5 Co-occurrence network of marker OTUs in foxtail millet rhizoplane microbiota. The enrichment direction of marker OTUs were tested by Spearman‟s rank correlation test (adjusted P<0.05). Negative and positive correlations with yield were plotted. Edges between OTUs were colored according to the Spearman‟s correlation coefficients(cc), cc >0.8 (purple), between 0.6 and 0.8 (light to dark blue), or cc <-0.4(red). OTUs annotated to families are colored according to phylum.
谷子根表、根表微生物富集的功能通路

图6. 根表或根际土中富集的KEEG通路。 (a) 根表和根际土相比,分别在张家口和杨淩地区富集的KEEG通路。红色为根表富集,蓝色为根际土富集,加号为富集程度较高。 (b) 根际、根表特异富集异生质生物降解途径。
Fig.6 KEGG pathways enriched in rhizoplane or rhizosphere soil samples. (a) The relative abundances of pathways were compared between rhizoplane to rhizosphere soil samples from Yangling and Zhangjiakou respectively. Pathways with a significant difference in reporter score (<-1.7, blue, enriched in rhizosphere soil; >1.7, red, enriched in rhizoplane) were retained. Reporter scores >2.3 or < -2.3 are shown in the map with +. (b) Compartment specific enrichment of Xenobiotics biodegradation pathways in rhizoplane and rhizosphere. The Reporter score of pathways more than 1.7 or less than -1.7 were plotted. YL: Yangling; ZJK: Zhangjiakou; BS: bulk soil; RS: rhizosphere; RP: rhizoplane.
方法核心OTU
在同一地点80%以上同类样品中出现OTU定义为Common OTUs。根际和根表差异采用Wilcoxon signed rank test,并用Benjamini and Hochberg (BH)校正 P-value。阈值 adjusted P < 0.01。
关联分析
关联表型和根表微生物。PERMANOVA采用vegan包的 adonis() 函数, permuted P value通过 999 permutations获得。mantel test采用ecodist包的 mantel() 鉴定微生物群体与表型相关。以上两种方法均采用BH方法校正,且 P value < 0.05作为显著的性状。所有表型性状相关采用 spearman‟s rank correlation,采用corrplot展示颜色梯度的相关系数。成块颜色的显著阈值为0.05。
randomForestSRC包对标准化的微生物群与产量回归,参数ntree=1,000 and nodesize=15 ( Subramanian et al [39]),1013中的709个用于建立预测模型,其它的用于检查适合度。其中839个OTUs高于30%的发生频率,与产量正相关或反相关。
功能预测采用PICRUSt
Reference
- 原文 https://doi.org/10.1093/gigascience/gix089
- 疾病表型与微生物组关联分析——让你的研究瞬间高大上 http://genome.cn/News/Achievements/354.html
- Zhao L. The gut microbiota and obesity: from correlation tocausality[J]. Nature Reviews Microbiology, 2013, 11(9): 639-647.
- Wang J, Jia H. Metagenome-wide association studies: fine-mining themicrobiome[J]. Nature Reviews Microbiology, 2016, 14(8): 508-522.
- Gilbert J A, Quinn R A, Debelius J, et al. Microbiome-wide association studies link dynamic microbial consortia to disease[J]. Nature, 2016, 535(7610): 94-103.
- 你所要知道的宏基因组关联分析知识都在这里 https://mp.weixin.qq.com/s?__biz=MjM5OTcyNjU2OQ==&mid=2651428022&idx=1&sn=257f09eef8af085d710e379a661d81c6&scene=0#wechat_redirect
- 王俊新发Nature综述 http://www.sohu.com/a/105739425_152537
- 发表于 2017-10-13 11:59
- 阅读 ( 6772 )
- 分类:其他组学
你可能感兴趣的文章
- 央视与遗传发育所合拍《走进科学-小菌株大作为》——枯草芽孢杆菌替代畜牧业抗生素添加 7690 浏览
- 16S+功能预测也能发Sciences:尸体降解过程中的微生物组 9177 浏览
- R画月亮阴晴圆缺:corrplot绘图相关系数矩阵 15881 浏览
- Cell:人体细菌到究竟有多少,再作报告必引此文 10499 浏览
- R语言添加p-value和显著性标记 17699 浏览
- ggalluvial:冲击图展示组间变化、时间序列和复杂多属性alluvial diagram 13474 浏览
