【DNA甲基化分析系列】450K甲基化谱分析,6篇讲义从零入门
看教程不够直观,那就看视频吧! >>点击加载视频
DNA甲基化(DNA methylation)是最早被研究的重要表观遗传修饰之一,基于芯片和下一代测序的高通量技术为DNA甲基化的广泛检测提供了技术平台(表 1-1)。Infinium HumanMethylation27 / 450 BeadChip芯片(以下简称为27K / 450K甲基化芯片)提供了全面的,经过专业选择的CpG位点覆盖的,高通量样本并行的和价格实惠的甲基化测定服务,使其成为表观基因组关联研究的理想解决方案。例如,癌症基因组图谱(TCGA)使用人类甲基化27 / 450 BeadChip芯片测定了涵盖34种癌症类型的超过12 000个样本中的DNA甲基化谱。与基于测序技术的甲基化检测手段相比,甲基化27 / 450 BeadChip芯片的低成本、高回报特性,使其得到了广泛的应用和推广,为群体甲基化模式、状态的研究提供了有力保障。
表 1-1 检测DNA甲基化的高通量检测技术
|
Assays |
Resolusion |
Whole Genome |
Target |
CGs |
% total CGs |
|
WGBS |
Single-base |
Yes |
All |
~ 28 million |
~100% |
|
RRBS |
Single-base |
No |
All |
~ 2-3 million |
~10.7% |
|
450K |
Single-base |
No |
All |
~ 0.5 million |
~1.8% |
|
27K |
Single-base |
No |
All |
~ 0.02 million |
~0.07% |
|
MeDIP-seq |
Region |
Yes |
Methylation |
/ |
/ |
|
MRE-seq |
Region |
Yes |
Unmethylation |
/ |
/ |
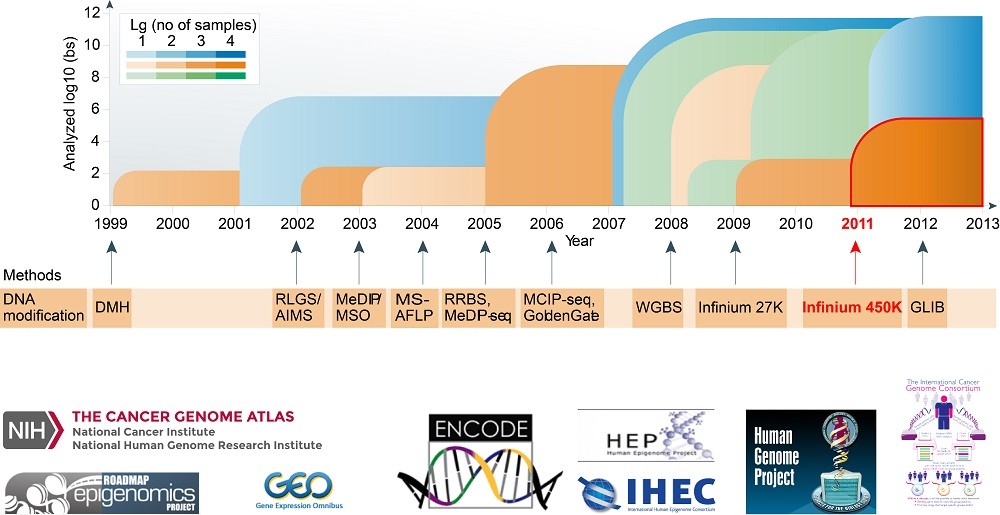
随着高通量生物技术(芯片、测序技术)的不断更新发展,高通量的DNA甲基化数据不断涌现,一些大型国际合作的生物大数据计划产生了Pb(petabyte)数量级的甲基化谱,如表1-2所示。由多个国家和地区的研究机构组成的“国际人类表观基因组同盟”(International Human Epigenome Consortium,简称IHEC)为了研究与人类健康和包括癌症在内的复杂疾病相关的细胞状态产出了超过1000个表观基因组的数据,其中由美国NIH资助的“表观组学线图计划” (Roadmap Epigenomics Mapping Consortium,简称Roadmap)产出了367个人类主要组织和细胞类型的DNA甲基化图谱,欧洲“血液表观基因组项目”(BLUEPRINT of Haematopoietic Epigenomes,简称BLUEPRINT)产出了与人类复杂疾病相关的82个不同血液细胞的DNA甲基化图谱。“DNA元件百科全书”计划(The Encyclopedia of DNA Elements,简称ENCODE)是继"人类基因组计划"后又一大型国际合作项目,来自世界各国32个研究机构对206个人类不同的细胞系和组织进行了DNA甲基化水平的测定。此外,“国际癌症基因组联盟”(The International Cancer Genome Consortium,简称ICGC)和美国癌症基因组图集(The Cancer Genome Atlas,简称TCGA)旨在从基因组、表观基因组和转录组等多维数据层面研究癌症的发生和发展,其中ICGC产出了涉及27种常见癌症的9000多个样本的DNA甲基化数据,TCGA产出了涉及34种癌症类型的10000多个样本的DNA甲基化数据,并且保留了癌症患者详细的临床数据资料,为生存分析提供了大量的数据资源。
表 1-2 人类表观基因组大数据计划中检测的样本数
|
项目 |
细胞/组织类型 |
样本数 |
|||||
|
27K |
450K |
WGBS |
RRBS |
MeDIP-seq |
MRE-seq |
||
|
TCGA |
34 |
2 720 |
10 181 |
27 |
|
|
|
|
ENCODE |
115 |
63 |
62 |
42 |
95 |
|
|
|
Roadmap |
132 |
|
|
140 |
116 |
45 |
45 |
|
ICGC |
27 |
|
9 246 |
71 |
|
|
|
|
BLUEPRINT |
82 |
|
6 |
146 |
|
|
|
在此,CEPI特推出针对450K甲基化芯片数据的分析做出了专题性实战策略,我们的目标是“包教包会,甲基化数据分析SO EASY”。
第一讲:芯片甲基化谱数据获取
第二讲:芯片甲基化谱数据标准化
第三讲:差异甲基化筛选
第四讲:甲基化动态性分析
第五讲:甲基化Markers功能富集分析
第六讲:提取生存相关的甲基化Markers
参考文献:
Laird, P.W., Principles and challenges of genomewide DNA methylation analysis. Nat Rev Genet, 2010. 11(3): p. 191-203.
Cancer Genome Atlas Research, N., et al., The Cancer Genome Atlas Pan-Cancer analysis project. Nat Genet, 2013. 45(10): p. 1113-20.
Cancer Genome Atlas Research, N., et al., Comprehensive Molecular Characterization of Papillary Renal-Cell Carcinoma. N Engl J Med, 2016. 374(2): p. 135-45.
Satterlee, J.S., D. Schubeler, and H.H. Ng, Tackling the epigenome: challenges and opportunities for collaboration. Nat Biotechnol, 2010. 28(10): p. 1039-44.
Roadmap Epigenomics, C., et al., Integrative analysis of 111 reference human epigenomes. Nature, 2015. 518(7539): p. 317-30.
Adams, D., et al., BLUEPRINT to decode the epigenetic signature written in blood. Nat Biotechnol, 2012. 30(3): p. 224-6.
Consortium, E.P., An integrated encyclopedia of DNA elements in the human genome. Nature, 2012. 489(7414): p. 57-74.
International Cancer Genome, C., et al., International network of cancer genome projects. Nature, 2010. 464(7291): p. 993-8.
- 发表于 2017-10-14 09:21
- 阅读 ( 16159 )
- 分类:方案研究
你可能感兴趣的文章
- 基因组学数据分析常用的在线资源 8313 浏览
相关问题
- CGTA 甲基化数据分析,用python方便吗,支持的工具有哪些啊? 1 回答
- 450芯片怎么下载 1 回答
- 不常见芯片平台GPL6604 2 回答
