编程模板-R语言脚本写作:最简单的统计与绘图,包安装、命令行参数解析、文件读取、表格和矢量图输出
看教程不够直观,那就看视频吧! >>点击加载视频
写在前面
个人认为:是否能熟悉使用Shell(项目流程搭建)+R(数据统计与可视化)+Perl/Python等(胶水语言,数据格式转换,软件间衔接)三门语言是一位合格生物信息工程师的标准。
之前分享过我个人的Shell语言和Perl语言脚本写作模板,今天再分享一下我的R语言模板,一次性解决困扰新手的众多问题,如包安装、命令行参数解析、文件读取、Anova组间统计和箱线图展示、表格和矢量图输出等。
希望对新手有所帮助!老司机尽情拍砖,指导我写出更好用、更规范的脚本。
模板主要内容
此模板主要以下分为5部分:
1. 程序功能描述和分析思路
每次写脚本,一定要写清楚程序的功能、实现的主要步骤,每个参数的详细说明和使用示例。如果英语描述不方便理解,建议中英文全写,方便自己和同行快速理解。千万要认识写清楚这些,现点养成好习惯,将来节省的时间会更多。
2. 依赖关系检查、安装和加载
R己有过万的可用包,很少需要我们开发新功能。每次需要加载需要的包,但没有会报错,直接安装又不会提醒导致重复安装浪费时间。我添加判断R包是否安装的代码,当R包缺失时会自己动从CRAN安装。对于CRAN没有的包,也提供了Bioconductor和Github安装代码,只需手动修改包名安装即可。
此步还包括加载了optparse包后,对命令行的解析,以及在屏幕上显示参数,让用户校验是否正确。
3. 读取输入文件
R用于作图,读入最多的是制表符分割的表格。我提供了使用rnorm生成的测试数据,并输入了测试文件,其实这种方式在程序测试、教程和实验分析对照中也是很常用的。
同时提供了read.table读取文件,或read.table(file.choose())弹窗选择输入文件两种方式。
4. 统计与绘图
这其实才是正文部分,用户根据自己的需求编写代码分析数据,完成工作。本模板以anova组间比较,和箱线图展示为例,方法大家测试工作过程。
表1. Anova分析测试数据结果
| GroupB-GroupA | 1.17 | 0.41 | 1.93 | 0.004 |
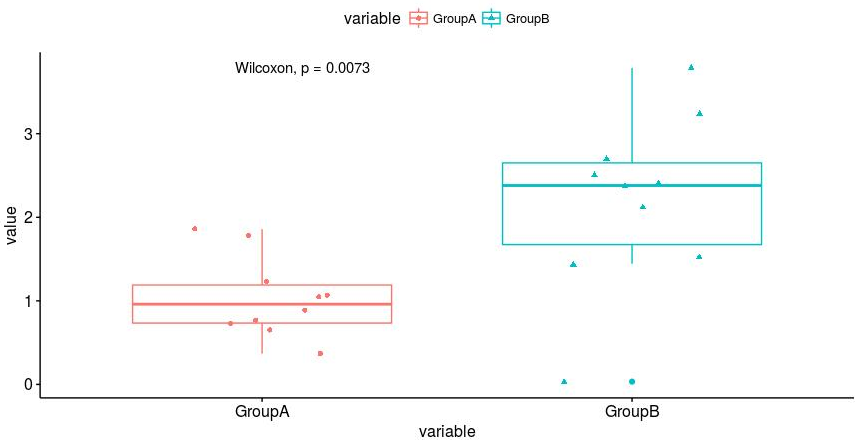
图1. 箱线图展示组间比较,且按组分配不同的颜色和形状(方便多组时区分),并添加统计p值。
5. 保存图表
分析的结果最重要的工作是保存图表,用于进一步分析和论文写作。本模板提供了输出表格的代码,并解决了存在行名输出时,文本文件在Excel中打开列名位置串一格的问题(R表输出时行/列名交叉点为空)。图片建议一律输出PDF格式,主要控制图片的长宽即可,不存辨率的问题,后期AI修改组合非常方便。杂志也更喜欢,因为他们接收后修改方便。
模板使用方法
保存文末的全部代码,推荐在Rstudio中保存文件名为template.r,可以逐行运行学习,体会常用功能。
命令行中使用
# 方法1. 直接使用Rscript执行 Rscript template.r # 调置输入数据文件,输出图表文件名前缀 Rscript template.r -i data_table.txt -o output # 方法2. 添加可执行权限和环境变量 chmod +x template.r # 链接至有权限写入的环境变量目录,如我的是~/bin/ ln `pwd`/template1.0.r ~/bin/ # 任何地方可以直接调用此脚本 template.r -i data_table.txt -o output
程序运行结果如下:
[1] "The input file is data_table.txt" [1] "The output file prefix is output" [1] "The output table is output.txt" [1] "The output figure is output.pdf"
文中把每个段落用if条件语句控制是否执行。方便读者开、关功能代码段。
用户还可根据自己的需求添加段落,再也不要为找常用语句的写法到处搜索了,有了模板,常用语句段落直接复制、修改,快速实现自己的工作!!!
模板全部代码及讲解
#!/usr/bin/env Rscript # 1. 程序功能描述和主要步骤 # 程序功能:Anova组间统计和箱线图展示 # Anova Script functions: Calculate pvalue of groups by aov and TukeyHSD # Main steps: # - Reads data table input.txt # - Calculate pvalue and save in output.txt # - Draw boxplot and save in output.pdf # 程序使用示例 # USAGE # Default # anova.r -i data_table.txt # -o otuput filename prefix for output directory name # 参数说明 # Options # -i/--input 输入数据表文件 input.txt # -o/--output 输出结果文件名前缀 output_prefix, 通常会有统计表txt和矢量图pdf options(warn = -1) # 2. 依赖关系检查、安装和加载 # See whether these packages exist on comp. If not, install. package_list <- c("optparse","reshape2","ggplot2","ggpubr") for(p in package_list){ if(!suppressWarnings(suppressMessages(require(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)))){ install.packages(p, repos="http://cran.r-project.org") suppressWarnings(suppressMessages(library(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE))) } } # 另两种常见R包安装方法 if (FALSE){ # Bioconductor安装 source("https://bioconductor.org/biocLite.R") biocLite(c("reshape2")) # Github安装 install.packages("devtools", repo="http://cran.us.r-project.org") library(devtools) install_github("kassambara/ggpubr") } # 清理工作环境 clean enviroment object rm(list=ls()) # 加载依赖关系 Load essential packages library(optparse) library(reshape2) library(ggplot2) library(ggpubr) # 解析命令行 if (TRUE){ option_list <- list( make_option(c("-i", "--input"), type="character", default="input.txt", help="Input table file to read [default %default]"), make_option(c("-o", "--output"), type="character", default="output", help="output directory or prefix [default %default]") ) opts <- parse_args(OptionParser(option_list=option_list)) # 显示输入输出确认是否正确 print(paste("The input file is ", opts$input, sep = "")) print(paste("The output file prefix is ", opts$output, sep = "")) } # 3. 读取输入文件 # 需要使用哪种方式,将其设置为TRUE,其它为FALSE即可 # 产生两组数据比较 if (TRUE){ # 产生两种各10个正态分布数值,分别以均值为1和2,标准差为均值的0.5倍,colnames修改组名称 set.seed(123) A = rnorm(10, mean = 1, sd = 0.5) B = rnorm(10, mean = 2, sd = 1) dat=as.data.frame(cbind(A,B)) colnames(dat)=c("GroupA","GroupB") # 保存数据文件方便下面演示 write.table(dat, file="opts$input", append = F, quote = F, sep="\t", eol = "\n", na = "NA", dec = ".", row.names = F, col.names = T) } # 从文件中读取 if (FALSE){ dat = read.table("opts$input", header=T, row.names = NULL, sep="\t") } # 弹出窗口选择文件 if (FALSE){ dat = read.table(file.choose(), header=T, row.names = NULL, sep="\t") } # 4. 统计与绘图 if (TRUE){ # 将宽表格将换为长表格(变量位于同列方便操作) dat_melt=melt(dat, id.vars = 0) # anova比较各组variable的观测值value anova <- aov(value ~ variable, data = dat_melt) # 计算Tukey显著性差异检验 Tukey_HSD <- TukeyHSD(anova, ordered = TRUE, conf.level = 0.95) # 提取比较结果 Tukey_HSD_table <- as.data.frame(Tukey_HSD$variable) # 展示组间差异:四列分别为均值差异,差异最小值,差异最大值和校正的P值 Tukey_HSD_table # 采用ggpubr包中的ggboxplot展示组间比较箱线图,并添加Wilcoxon整体差异P值 # 增加了jitter点,color和shape按分组variable变化,添加统计值 p <- ggboxplot(dat_melt, x="variable", y="value", color = "variable", add = "jitter", shape="variable") +stat_compare_means() p } # 5. 保存图表 if (TRUE){ # 保存一个制表符,解决存在行名时,列名无法对齐的问题 write.table("\t", file=paste(opts$output,".txt",sep=""),append = F, quote = F, eol = "", row.names = F, col.names = F) # 保存统计结果,有waring正常 write.table(Tukey_HSD_table, file=paste(opts$output,".txt",sep=""), append = T, quote = F, sep="\t", eol = "\n", na = "NA", dec = ".", row.names = T, col.names = T) print(paste("The output table is ", opts$output, ".txt", sep = "")) # 保存图片至文件,pdf方便AI修改成出版级图片 ggsave(file=paste(opts$output,".pdf",sep=""), p, width = 5, height = 3) print(paste("The output figure is ", opts$output, ".pdf", sep = "")) }
猜你喜欢
- 热文:1高分文章 2不可或缺的人 3图表规范
- 一文读懂:1微生物组 2寄生虫益处 3进化树
- 必备技能:1提问 2搜索 3Endnote
- 文献阅读 1热心肠 2SemanticScholar3geenmedical
- 扩增子分析:1图表解读 2分析流程 3统计绘图 4功能预测
- 科研经验:1云笔记 2云协作 3公众号
- 系列教程:1Biostar 2微生物组 3宏基因组
- 生物科普 1肠道细菌 2人体上的生命 3生命大跃进4细胞的暗战 5人体奥秘
- 发表于 2018-01-18 22:55
- 阅读 ( 9795 )
- 分类:其他组学
你可能感兴趣的文章
- 央视与遗传发育所合拍《走进科学-小菌株大作为》——枯草芽孢杆菌替代畜牧业抗生素添加 7650 浏览
- 16S+功能预测也能发Sciences:尸体降解过程中的微生物组 9100 浏览
- R画月亮阴晴圆缺:corrplot绘图相关系数矩阵 15823 浏览
- Cell:人体细菌到究竟有多少,再作报告必引此文 10355 浏览
- R语言添加p-value和显著性标记 17624 浏览
- ggalluvial:冲击图展示组间变化、时间序列和复杂多属性alluvial diagram 13388 浏览
