BRIG∣原核生物比较基因组圈图展示神器
看教程不够直观,那就看视频吧! >>点击加载视频
细菌基因组比较分析是时下很火热的研究方向,通过比较能够获得两个或多个基因组之间的共性及异性,在系统进化分析,物种分类鉴定,功能研究等领域中具有重要价值。 如何直观地展示基因组结构之间的相似性?BRIG软件教您轻松搞定。
BRIG是一款功能强大的分析及可视化软件,操作简单,界面友好,能够在一张图中展示近百个基因组比较结果。
接下来让我们来一场与BRIG圈图相约的美妙旅行。
BRIG是由Java 1.6编写的,使用BLAST(Basic Local Alignment Search Tool)进行基因组的比对分析并使用CGView进行图像的生成。
1、首先安装Java和BLAST/BLAST+,解压BRIG安装包,双击BRIG.jar,运行BRIG。
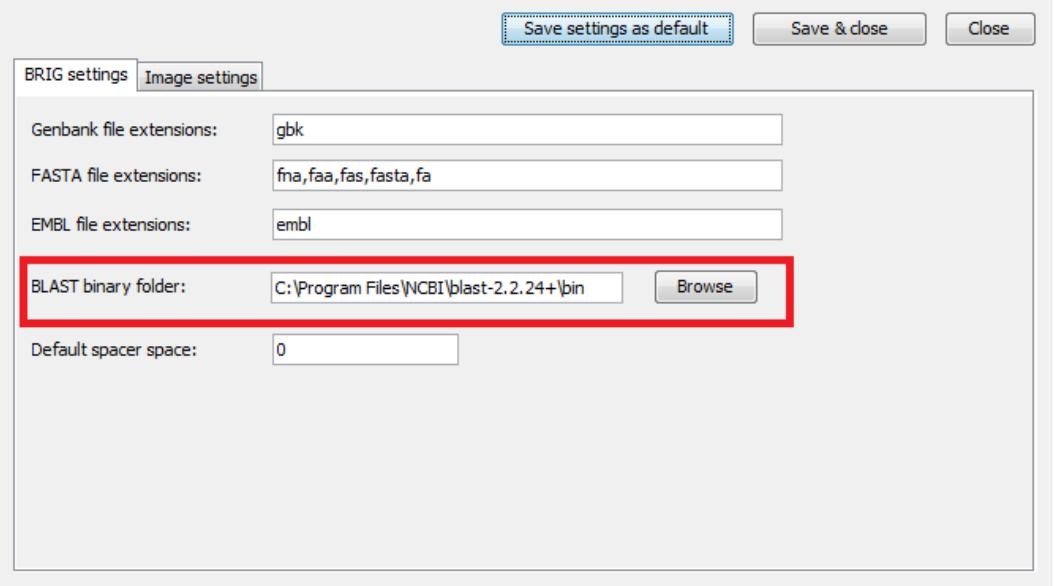
(1)通过Preferences >BRIG options,设置blast+软件路径,如下:
(2)载入示例文件
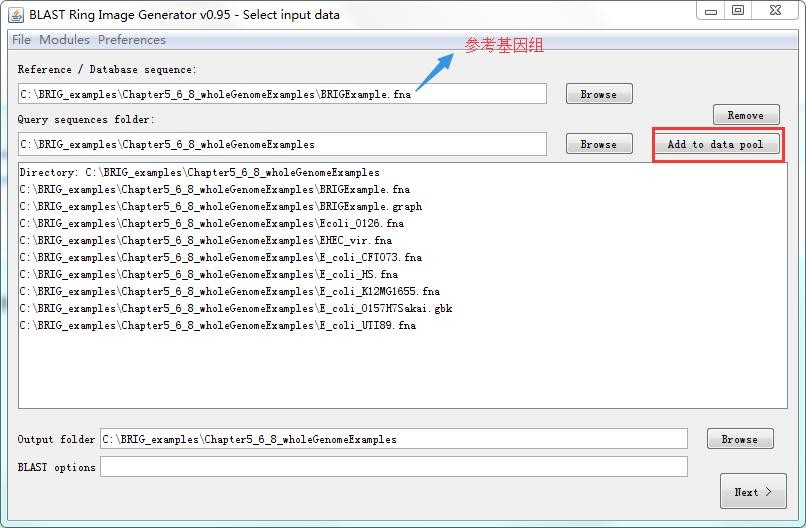
选择载入参考基因组,设置输入文件夹和输出文件夹,单击Add to data pool将输入文件夹中的序列添加到BRIG data pool,单击next。
(3)设置每一圈信息
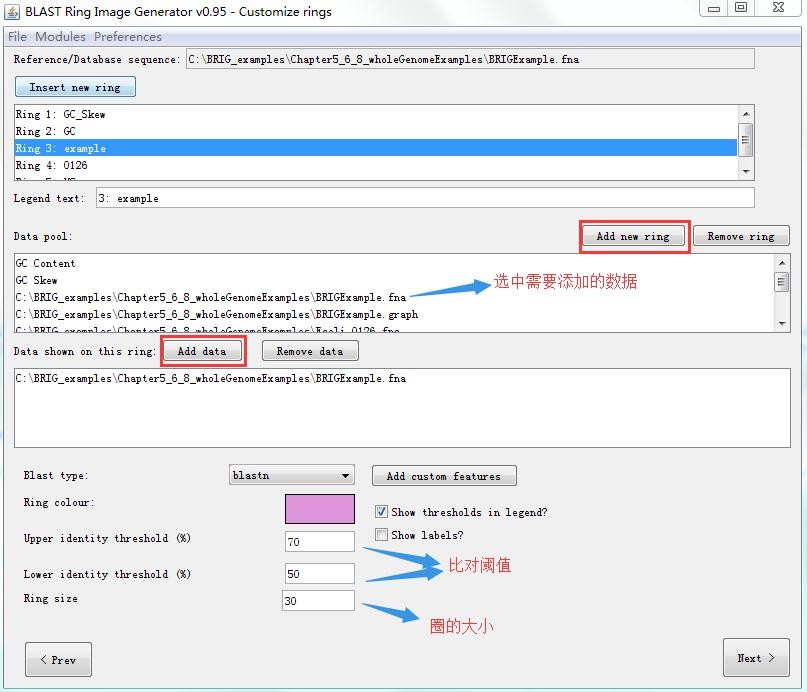
Add new ring添加圈,选中data pool中的数据,单击Add data,添加选中的序列。设置完成后单击next。一般设置如下:
Ring1 | GC_Skew |
Ring2 | GC content |
Ring3 | refgene.fna |
Ring4 | |
Ring5 | genome2.fna |
…… | …… |
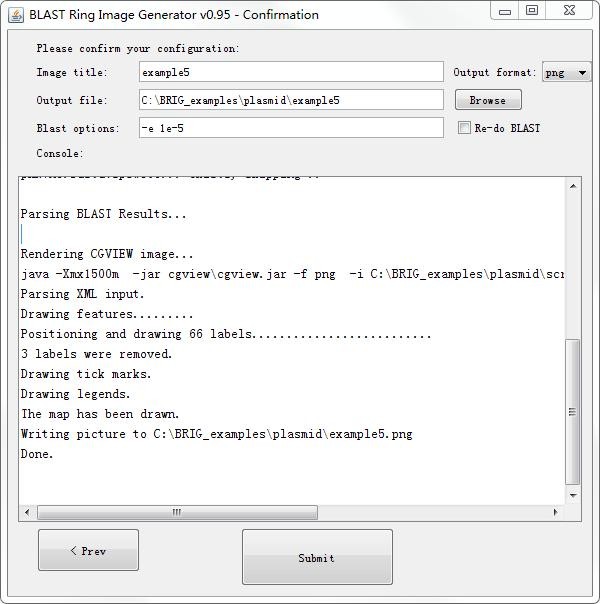
(4)提交任务
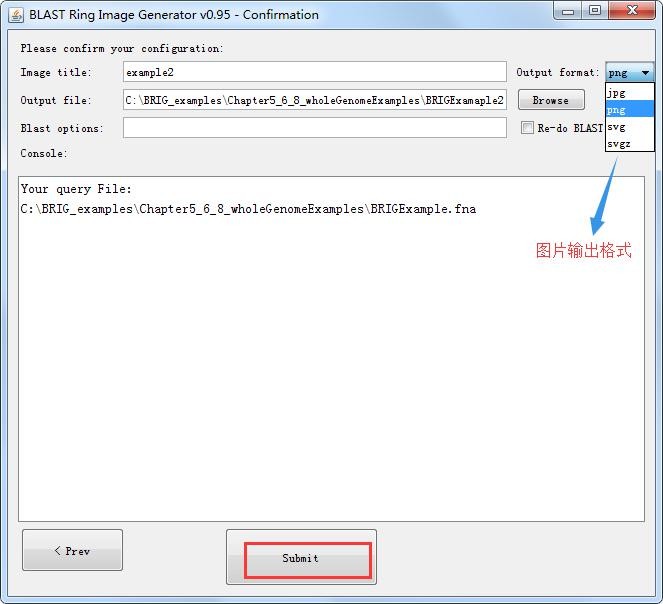
选择图片输出格式:jpg、png、svg、svgz,单击submit,提交比对任务并输出图片。
(5)最终输出结果
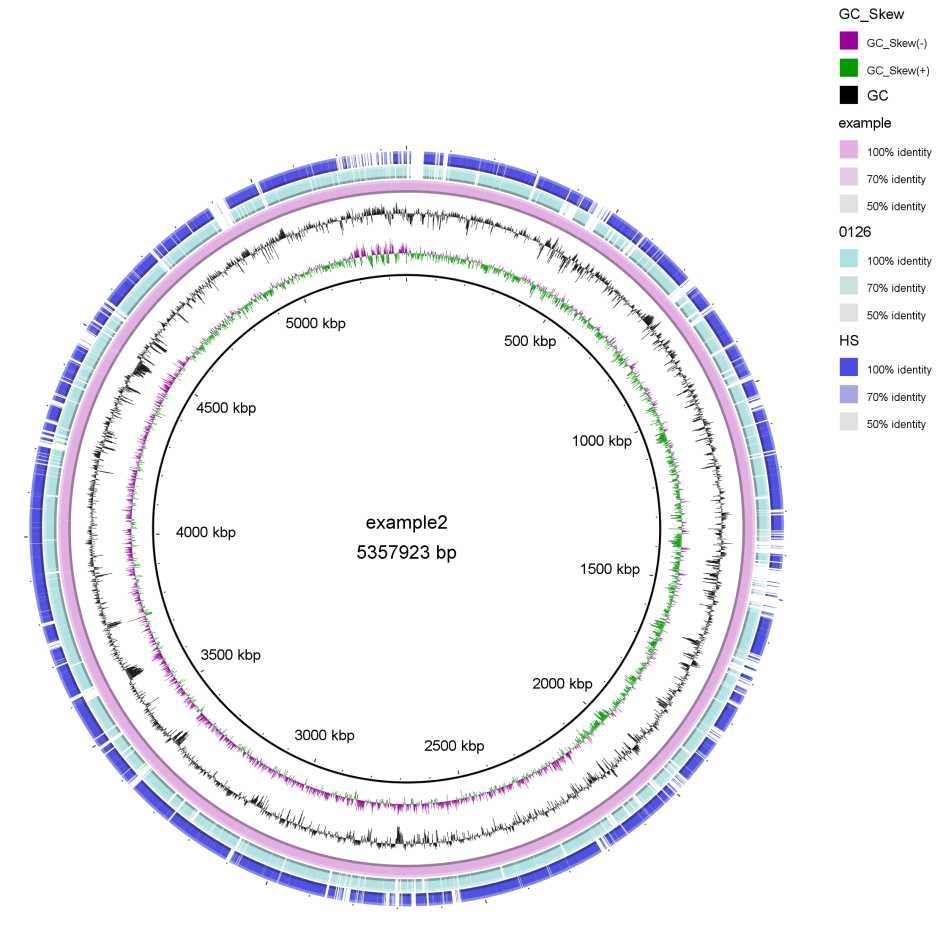
2、看着上面的图是不是感觉缺少点什么?是的,缺少注释信息。当序列添加完成后,通过Add custom feature按钮,进入基因注释信息编辑栏。
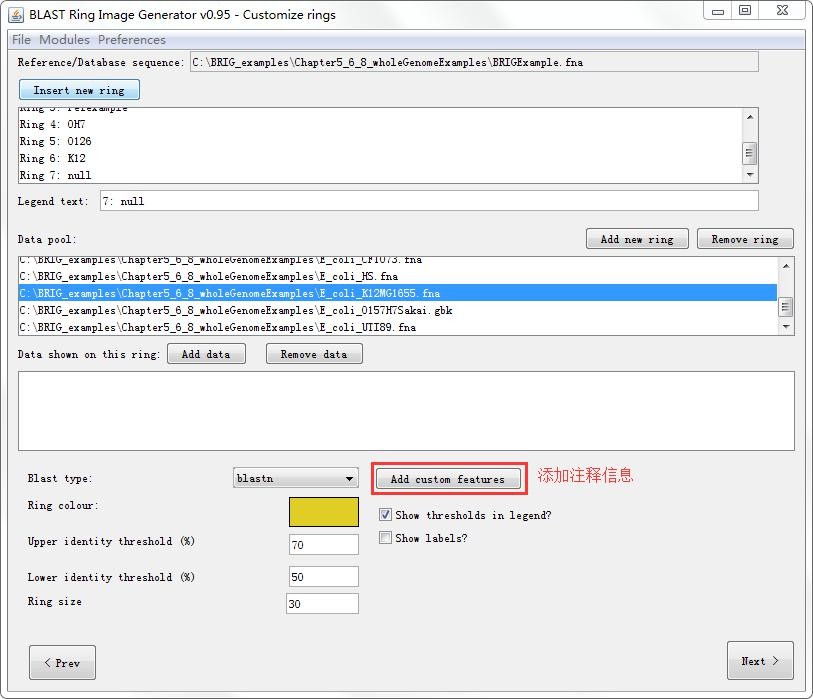
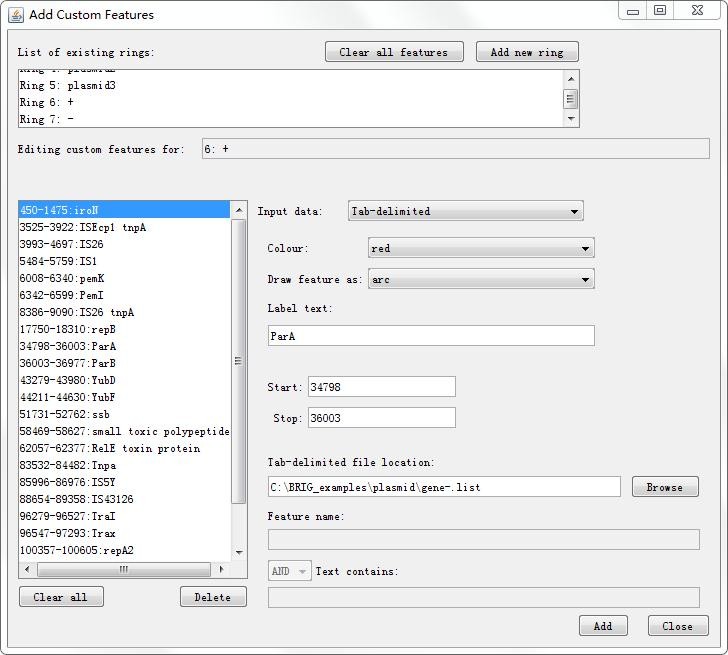
注释信息的添加有四种方式,单击Input data下拉栏显示:Single entry(即编辑单个基因)、Tab-delimited(导入xls文件)、Genbank(添加gbk文件)、Embl(添加embl文件)。
(1)通过xls文件导入自己感兴趣的基因。
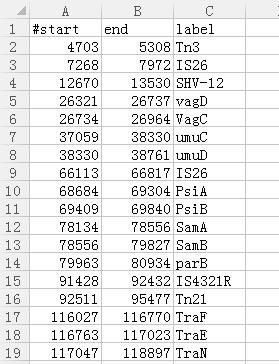
注释信息文件(list.xls)整理如下,共三列:strat、end、label,注意表头一定要添加#,基因起点(start)位置必须小于终止(end)位置。可将正链、负链基因整理成两个list,注释信息可添加成两圈。Input data选择“Tab-delimited”,上传list文件,同时可通过colour对不同基因进行颜色设置。
提交任务后,结果展示如下:
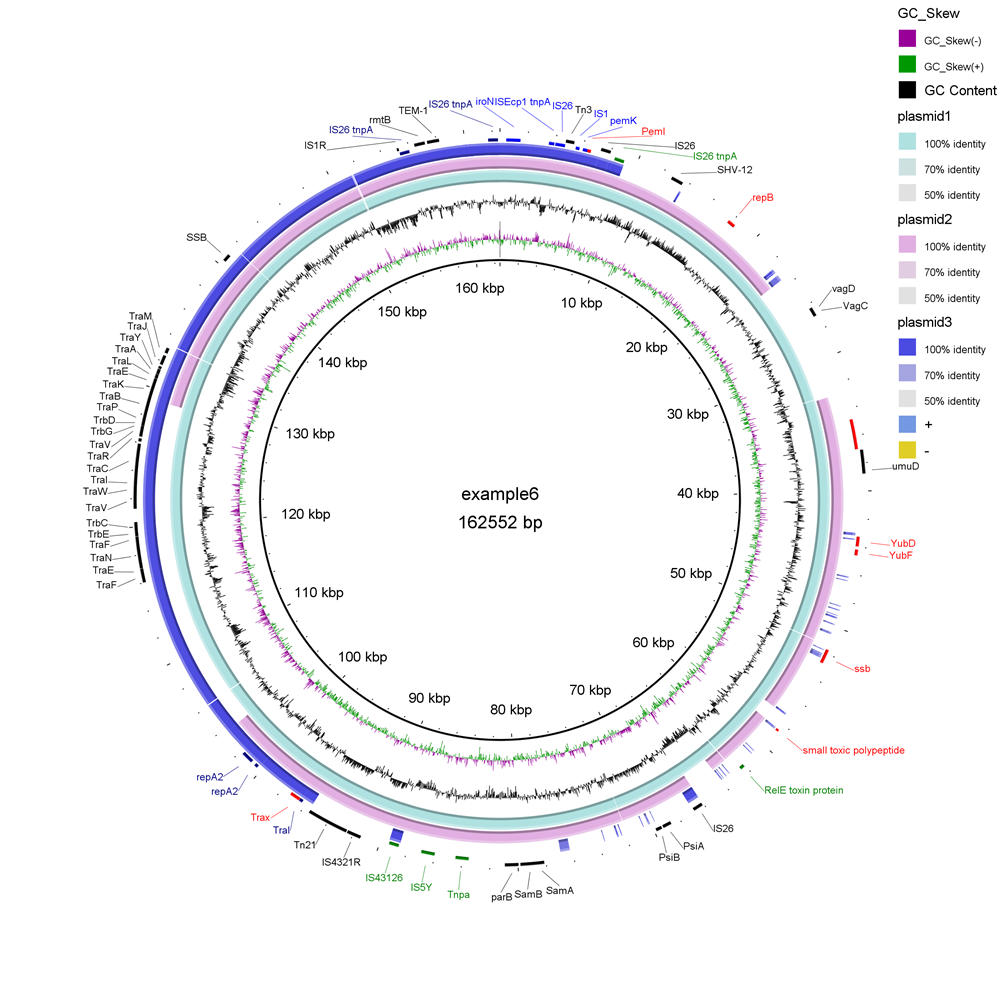
(2)通过gbk文件导入注释信息
Input data选择”Ganbank”,Feature name设置为”CDS”,通过Browse载入gbk文件。
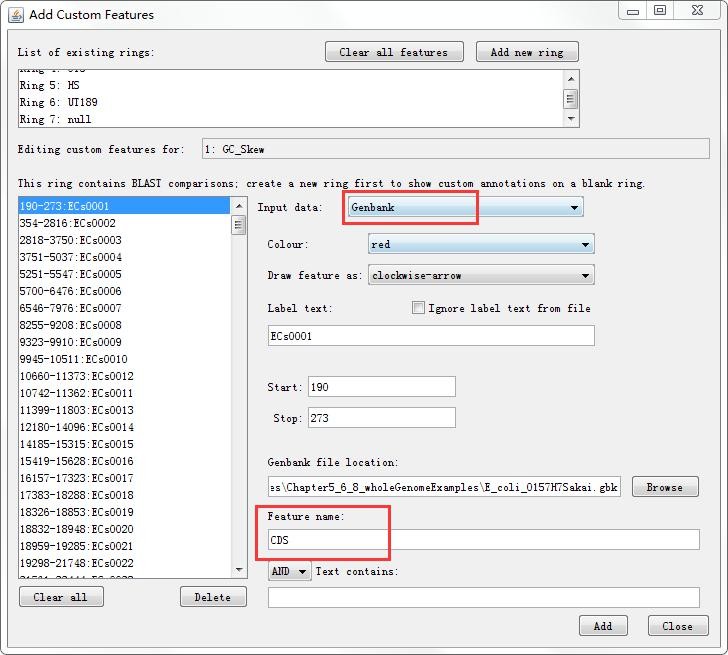
结果展示:
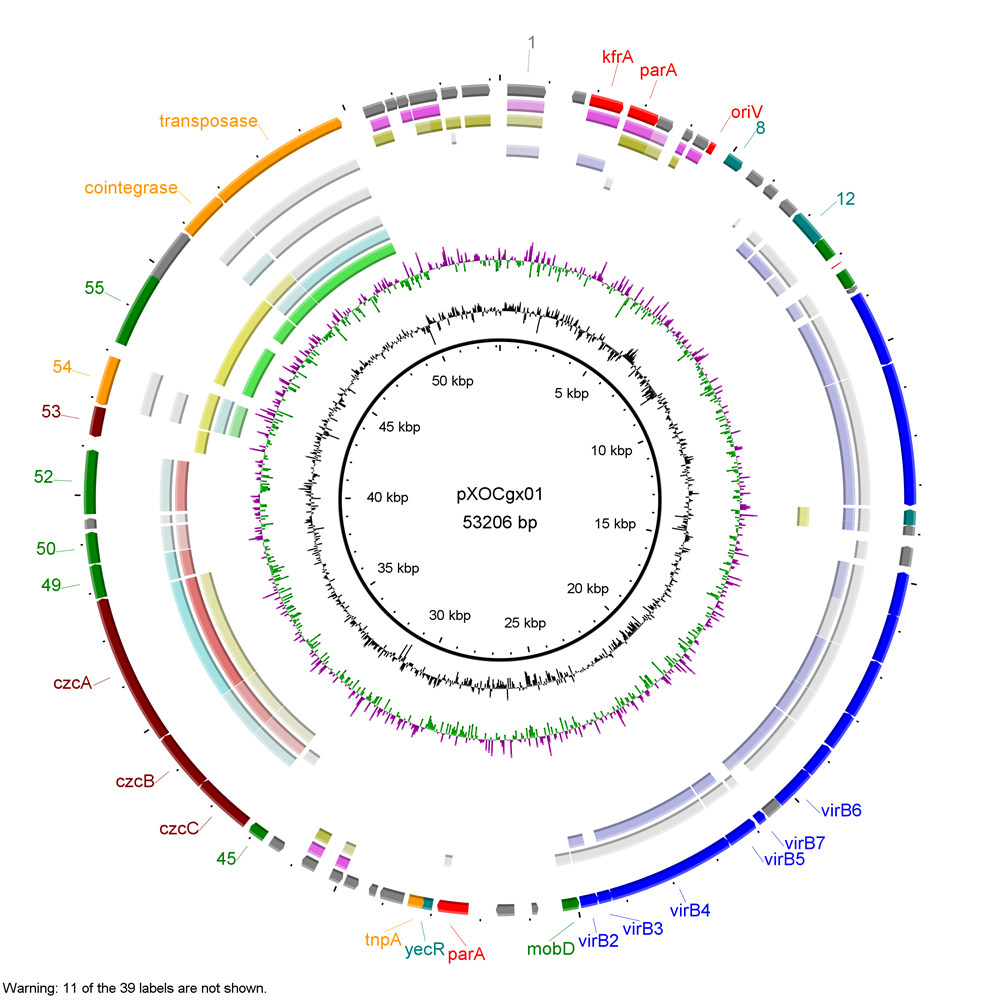
3、注释信息添加完成了,如果我们想知道这段序列的reads覆盖度该如何去做呢?别着急,BRIG中也可导入sam文件,从而显示序列的reads覆盖度信息。
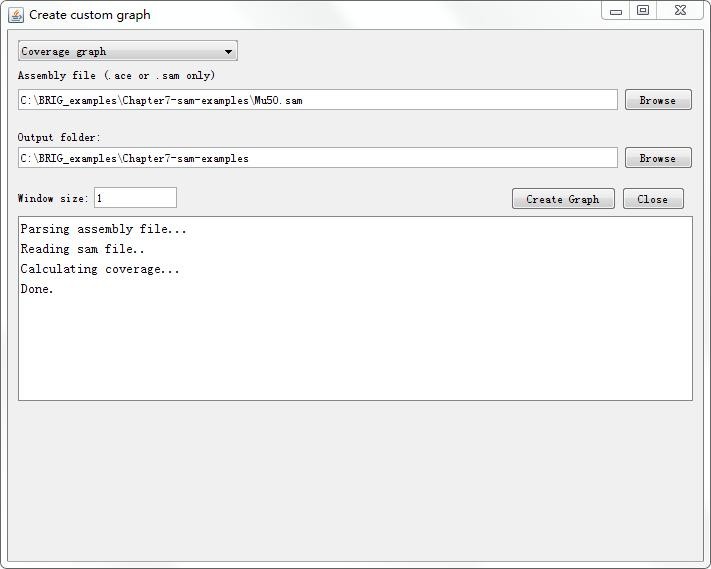
(1)创建覆盖度表格文件
通过Modules > Create graph files,通过下拉菜单选择Coverage graph(支持sam、ace格式文件),选择sam文件,输出文件目录,设置窗口大小为1,单击“Create Graph”完成创建。
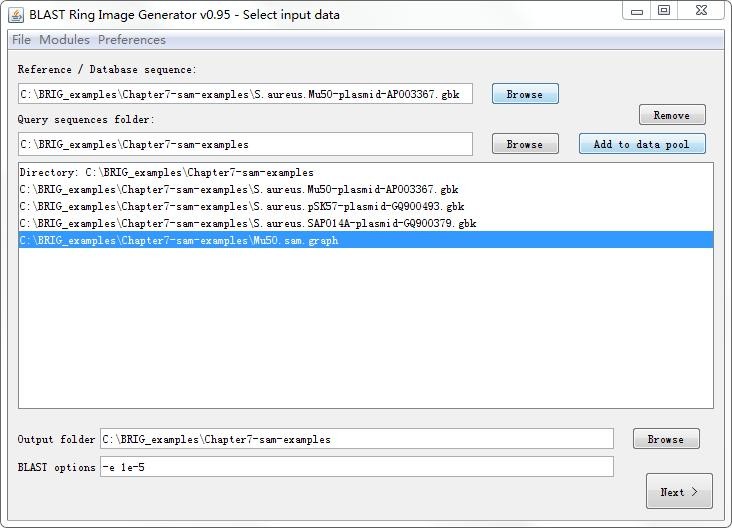
(2) 设置参考基因组
选择并设置号参考基因组,同时在data pool中显示我们创建成功的sam.graph文件,单击next。
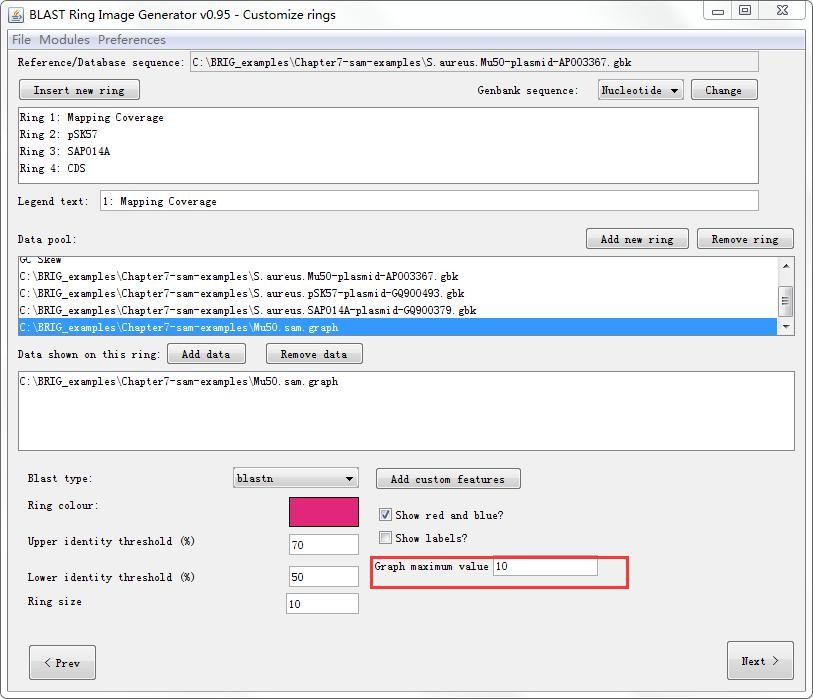
(4)添加比较基因组圈图信息
创建4个ring,分别命名为Mapping Coverage、pSK57、SAP014A、CDS。ring1添加数据Mu50.sam.graph,graph maximum value设置为10;ring2、ring3分别添加pSK57、SAP014A数据,设置完成后点击next并提交。
结果展示:

这项神技能,各位小伙伴们get到了吗?扫码了解更多哦!

参考文献:
Beatson SA, Ben ZNL, Petty NK, et al. BLAST Ring Image Generator (BRIG): simple prokaryote genome comparisons[J]. Bmc Genomics, 2011, 12(1):402.
- 发表于 2018-01-26 14:00
- 阅读 ( 21382 )
- 分类:基因组学
你可能感兴趣的文章
相关问题
- 如何进行HGT的分析 0 回答
