2篇五分数据分析文章教你怎么正确的利用CeRNA假说
看教程不够直观,那就看视频吧! >>点击加载视频
在pubmed上利用TCGA的数据及CeRNA假说整合分析所发表的文章已经快五十篇了,他们从不同程度上来诠释CeRNA假说以及应用场景,我们都知道ceRNA全称为competing endogenous RNA,是一种能够竞争结合RNA的作用元件,通常lncRNA和circRNA会竞争结合miRNA从而抑制miRNA对mRNA的作用。
简单来说这里就涉及到三种分子:miRNA\lncRNA\mRNA;恰好TCGA上这些数据非常多,并且也有完善的预后信息,那么用这三种数据+CeRNA假说+预后+三十多种肿瘤,我们可以讲的故事就太多了。
比如这篇三分多的:Pseudogenes transcribed in breast invasive carcinoma show subtype-specific expression and ceRNA potential;从假基因(TCGA中一种特殊mRNA)的角度去挖掘乳腺癌亚型特异的CeRNA机制
又比如这篇四分多的:MicroRNA-mediated networks underlie immune response regulation in papillary thyroid carcinoma;从免疫应答的角度结合TCGA的数据构建了miRNA介导的CeRNA网络,揭示甲状腺癌症免疫应答相关的生物学调控过程
又比如最近这篇四分多的:Comprehensive analysis of competitive endogenous RNAs network associated with head and neck squamous cell carcinoma;基于TCGA的头颈癌的RNA-Seq数据构建CeRNA网络,结合临床数据分析网络中预后相关的分子,挖掘潜在的lncRNA标志物。
以上都是基于CeRNA假说利用TCGA的数据构建的CeRNA网络,从不同的角度来诠释一些生物学现象,总结推理无非是建立某个肿瘤的CeRNA网络,从网络中寻找有意义的分子(与表型产生关联),进而找到有意义的生物学过程或调控过程或标志物。
那么这两篇文章五分多的文章就更有意思了,他们给了我们构建CeRNA的思路和工具
1、GDCRNATools: an R/Bioconductor package for integrative analysis of lncRNA, miRNA and mRNA data in GDC,提供了一个便捷的R包可以让你几条命令就能构建出自己的CeRNA网络来,这个R包主要是根据CeRNA的假说,根据TCGA的表达谱计算miRNA-mRNA-lncRNA直接的相关性从而建立起表达相关的CeRNA网络,便于你后续的分析和挖掘。
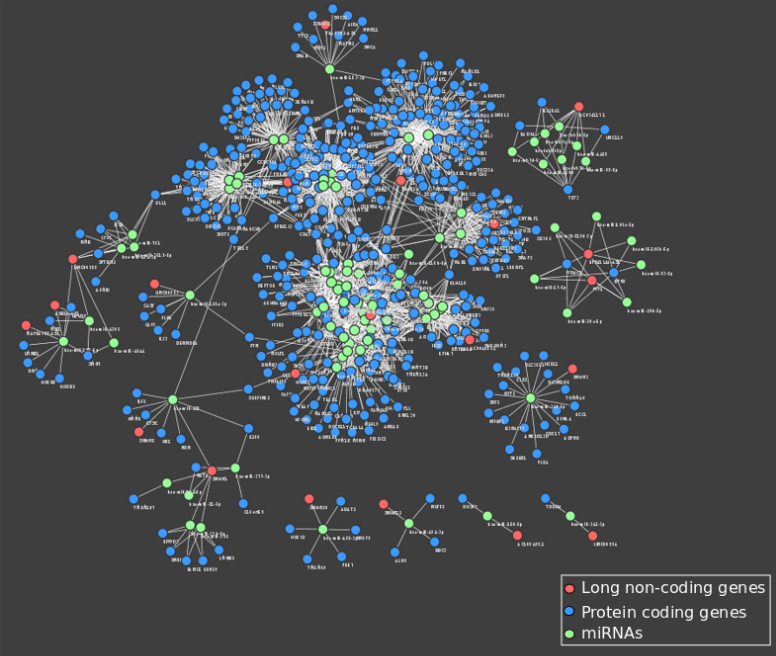
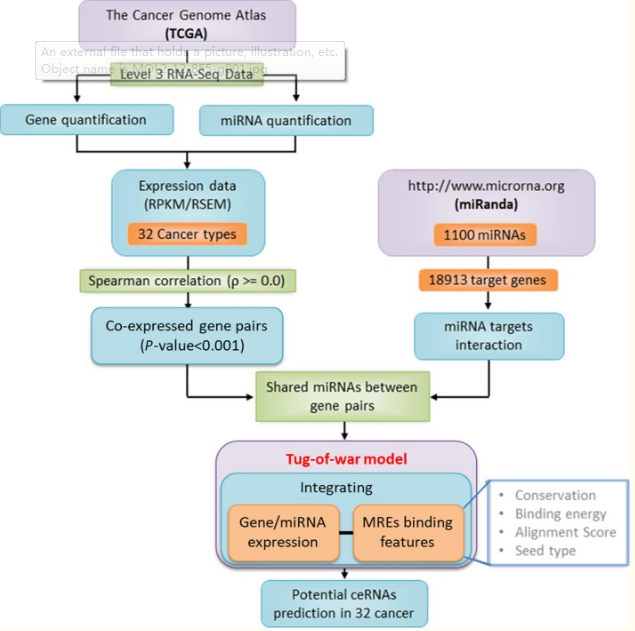
2、miRNA‐mediated ‘tug‐of‐war’ model reveals ceRNA propensity of genes in cancers,相对于第一种方式这篇文章提供了一个新的方法来预测CeRNA;我们在实际的研究(灌水)中可以结合这两种来做到大方向一致小课题不断。
当然思路在手,如何快速实现是个问题,数据分析文章牛人一个月一篇,没有门道的半年还在纠结差异分析怎么做,我们在社区回答了近千个数据分析的小白问题,开发了二十多个简易工具只为你能够更快的掌握数据挖掘的套路,文章不断,基金不断;这周末我们在浙大旁边有个高强度的生信训练营,旨在让科研工作者更快的入门生信分析,学会寻找更多的数据分析思路,让数据分析条条大路通罗马。
我们的生信训练营从最基础的何为RNA-Seq数据开始,辅以实际数据分析文章案例,两天半时间让你从思路获取、选题、构思、实现、写作、回复审稿意见六个方面得到充分的训练;然后在线上继续一年的视频学习(社区中一年内当前的和未来的所有视频);
从我们的生信训练营中你将学会诸如绘图、R语言入门、富集分析、CeRNA、WGCNA、网络分析、火山图、热图、数据下载、处理及标准化等等基本的生信技能。
注意:小班授课,训练营第2期还剩五个名额!!!!!
课程安排如下:

时间地点:

联系方式:

- 发表于 2018-08-17 14:38
- 阅读 ( 10012 )
- 分类:软件工具
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9698 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10631 浏览
- TCGA数据库 7851 浏览
- 如何解决生信新手分析时常常遇到的数据问题 7751 浏览
- TCGA下载工具超级版,在线秒下载 7666 浏览
- 图形化开放式生信分析云平台产品开发 - 7 分析报告的模板定制与自动生成 3628 浏览
