文献解读(7)肺ADC中的空间甲基化异质性
看教程不够直观,那就看视频吧! >>点击加载视频


肺腺癌(ADC)是肺癌中最普遍的亚型,占所有病例的46%以上。在分子层面上,ADC的特征是具有复杂的基因组格局。最近的研究集中在深入了解基因组和表观基因组改变的后果以及肿瘤异质性在治疗反应和治疗抵抗中的作用。ADC组织病理学生长模式的分析显示广泛的肿瘤内异质性(ITH)。值得注意的是,尽管在许多情况下发现了多种组织学,但主要的组织形态学生长模式(鳞状,腺泡,乳头状,微乳头状,实体状)具有很高的预后性和预测性。在分子水平上,研究发现驱动程序突变等位基因频率与组织学生长模式之间具有高度相关性。在表观基因组水平上,第一项研究报道了肺ADC中DNA甲基化模式的高肿瘤内异质性。但是,到目前为止,尚未进行ADC中表观基因组学和形态学ITH的比较空间分析。
那么今天小编为大家带来这篇文章作者系统地评估了来自七个主要ADC的122个肿瘤的形态生长模式,以评估组织学亚型的空间分布。

(1)患者和样本:作者根据2015年WHO肺癌分类标准(表1),对7位II至IV期ADC患者的肿瘤(n = 27)和非肿瘤(n = 7)组织标本进行了评估。对其中四名患者的六个淋巴结转移进行了类似的评估。病理学家将最大直径的肿瘤切片分为5 x 5 mm的区域并在连续的H&E染色切片上确定每个节段的主要组织学生长模式(定义为百分比最高的模式)。在这七名患者中,四名携带KRAS基因突变,三名携带TP53(其中两个是双重突变)。
(2)DNA提取和定量:将来自每个片段的六个连续的未染色的10 µm组织切片去石蜡并用蛋白酶K消化。使用LEV DNA纯化试剂盒进行DNA提取。使用Qubit HS DNA分析确定DNA浓度。
(3)Digital PCR:使用Quantstudio 3D Digital PCR系统利用20 ng模板DNA分析了患者4淋巴结中激活的KRAS G12S突变的存在,并验证了TaqMan SNP基因分型分析。
(4)全局甲基化谱:应用Infinium MethylationEPIC BeadChip 来评估> 866,000 CpG位点的全基因组DNA甲基化水平。在DKFZ基因组学和蛋白质组学核心设施中,对来自恶性和正常肺FFPE组织样本的500ng基因组DNA进行亚硫酸氢盐转化并进行甲基化分析。
(5)DNA甲基化数据分析:使用“minfi”R包处理原始甲基化数据。正常样本的甲基化定义为七个匹配的非肿瘤性肺组织样品的平均甲基化。
(6)基因组注释:通过标准的Illumina Infinium甲基化EPIC BeadChip标准进行CpG注释。
(7)无监督分层聚类:使用肿瘤内甲基化差异最大的10,000个探针的β值,进行甲基化水平的无监督分层聚类。聚类采用欧氏距离和平均连锁作为聚类方法。
(8)拷贝数分析:使用R包“conumee”从甲基化和未甲基化的探针信号强度值得出拷贝数分布图。
(9)系统发生树重建:根据肿瘤内甲基化变异的距离矩阵来推断系统发生的关系。使用具有最大肿瘤内甲基化差异的10,000个探针的Beta值来计算DNA甲基化欧几里德距离矩阵。最后使用R包“ape”来构建系统发生树。
(10)差异甲基化分析:使用线性模型来评估肿瘤和肺肿瘤组织中的甲基化差异。使用PANTHER分类系统对差异甲基化基因进行了GO和KEGG分析。
(11)相关性分析:应用Pearson相关系数来估计基于甲基化的距离。

(1)ADC的组织学和表观基因组分析:为了对组织病理学肿瘤内异质性进行系统表征,作者将7位ADC患者的中央肿瘤切片(表1)解剖为122个肿瘤和34个5 x 5 mm的非肿瘤部分。
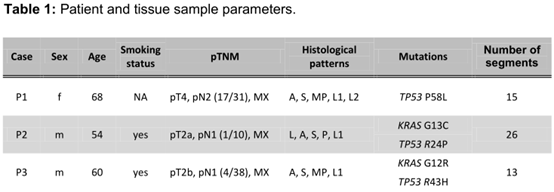
 表1:患者和组织样本参数
表1:患者和组织样本参数
靠近转录起始位点(TSSs)的DNA甲基化影响基因活性。为了确定TSS活性,作者将active 和 passive的TSS的甲基化水平与正常肺组织的H3 K4Me3标记进行了比较。结果显示具有K4Me3标记的潜在passive TSS在其TSS周围的1kb区域显示甲基化水平降低,而没有K4Me3标记的位点显示甲基化水平较高(图1 A)。并且与passive TSS相比,活性(active)位点总体上甲基化水平较低(图1 B):具有K4Me3标记的active TSS处的CpGs显示平均甲基化水平为0.05至0.1,而没有激活组蛋白标记的passive TSS显示平均甲基化水平> 0.5。接下来作者比较了非肿瘤组织的平均甲基化水平与患者2的两个腺泡,鳞状,乳头状和实心部分的甲基化。揭示了肿瘤内和组织内的异质性(图1C)。
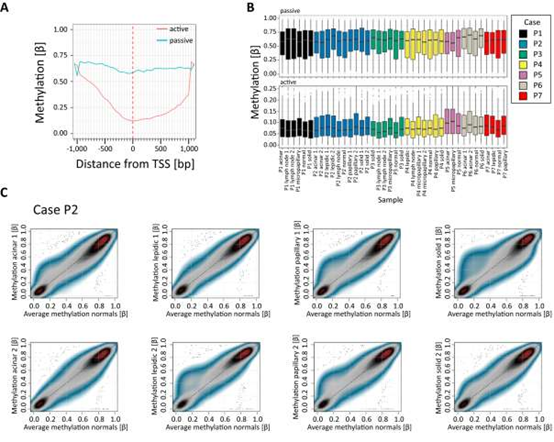 图1:DNA甲基化图谱的整体变化
图1:DNA甲基化图谱的整体变化
(2)DNA甲基化分析揭示了广泛的肿瘤内异质性:在这一部分作者对不同节段和淋巴结转移之间的DNA甲基化模式进行单独评估,发现DNA甲基化具有广泛的肿瘤内异质性,并通过无监督分层聚类来显示结果(图2A)。此外,对患者所有节段的分析进一步说明了同一肿瘤各节段之间的强烈变异性(图2 B)。
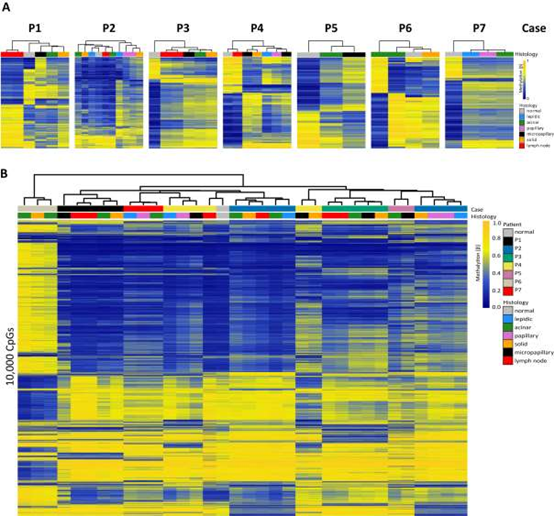 图2:甲基化模式的无监督分层聚类
图2:甲基化模式的无监督分层聚类
为了将异质性程度放在更广泛的背景下,作者对来自TCGA肺腺癌的33个肿瘤区段的启动子区域和369例患者(366个肿瘤和38个正常样品)的相应位点进行了12601个位点的无监督分层聚类。结果显示这些样本没有形成单独的群集,并且个体内的区段彼此之间的相似性仍然不与来自不同个体的区段相似(图3)。
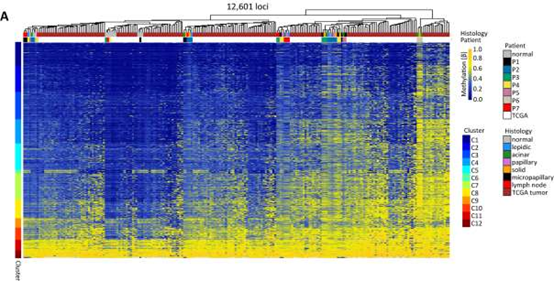 图3:来自TCGA肺ADC组的7例和369例(366个肿瘤样本,38个正常样本)样本的所有区段的启动子相关位点(n = 12,601)的甲基化模式的层次聚类。
图3:来自TCGA肺ADC组的7例和369例(366个肿瘤样本,38个正常样本)样本的所有区段的启动子相关位点(n = 12,601)的甲基化模式的层次聚类。
(3)拷贝数变异的异质性:为了更深入地了解拷贝数变异(CNV)的空间分布,作者基于EPIC阵列数据确定了每个区段的CNV分布图。结果在33个肿瘤区段中的32个中鉴定了具有体细胞拷贝数增减的CNV(图4A)。总体而言,CNV图谱在肿瘤内部比在肿瘤之间更相似。仅患者4的淋巴结转移未显示出具有与匹配的非肿瘤组织相似的特征的CNV。接下来作者基于TCGA数据进一步确定了在肺ADC和潜在的肿瘤驱动CNVs中被鉴定为反复扩增或缺失的基因的拷贝数状态。这项分析表明尽管所有片段均携带至少一个重复扩增或缺失的基因的CNV,却发现了克隆和亚克隆拷贝数得失的片段之间的空间异质性(图4 B)。
 图4:按染色体分组的每个染色体(列)和患者/组织学的体细胞拷贝数变化图
图4:按染色体分组的每个染色体(列)和患者/组织学的体细胞拷贝数变化图
(4)基于CNV和DNA甲基化数据的生长模式的克隆进化:为了深入了解ADC的克隆进化并探索淋巴结转移的组织学起源,作者根据DNA甲基化变化和CNV得出的距离矩阵对40个多区域片段进行了系统发育重建。结果作者在表观遗传水平上观察到ADC的分支进化,在原发肿瘤的生长模式之间具有广泛的克隆多样性(图5 A)。甚至同一增长模式的各个部分也显示出分支演化,表明同一模式在不同区域或不同时间点独立出现(图4 B)。为了进一步探索不同肿瘤区域之间的亚克隆关系不受选择性限制或生态位适应的影响,作者对远离调控元件的基因组位点(n = 83,756)进行了相关分析。相关性证实了肿瘤内的亚克隆关系和广泛变化(图5B)。
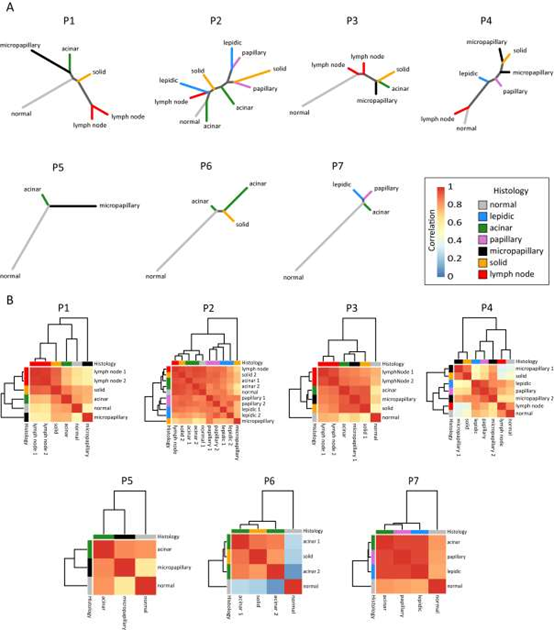 图5:基于DNA甲基化变化的片段之间的系统发生关系以及不同肿瘤区域之间的相关性分析
图5:基于DNA甲基化变化的片段之间的系统发生关系以及不同肿瘤区域之间的相关性分析

为了重建组织形态学模式的克隆进化,本研究对27个原发性肿瘤区域,7个匹配的正常组织和7个ADC病例的6个淋巴结转移进行了整体DNA甲基化分析。结果表明,表观基因组特征的广泛变化有助于原发性ADC和淋巴结转移的分子和表型异质性。
文献解读(1)MiRNA表达模式与肺腺癌的肿瘤突变负担相关?
文献解读(2)通过膀胱癌肿瘤突变负荷和免疫治疗的关系来进行多组学分析
文献解读(3)what?铁代谢的分子治疗可能对癌症的治疗有帮助?
文献解读(4)m6A修饰可能和AML疾病的发生发展相关?TP53突变特征与m6A调控基因发生基因组学改变存在一定的关系?m6A对生存有影响?m6A修饰发生基因组学上可以改变相关的临床特征?
文献解读(5)对于那些存在整体甲基化水平缺失的病人,使用免疫治疗药物对于这一类病人的生存时间竟然会有显著地改善
文献解读(6)胎盘转录组与孕妇孕前体重指数和新生儿出生体重的共表达分析
- 发表于 2019-11-25 13:41
- 阅读 ( 4753 )
- 分类:文献解读
