2020年5+生信文章什么样?
看教程不够直观,那就看视频吧! >>点击加载视频
话不多说,直奔主题。(看发表时间,很新鲜)

这杂志大家也不陌生,都是老朋友了,生信友好型杂志。
看看文章都干了什么:
1.故事背景
作者系统性的开发了一个跨越不同转录组平台和病人队列的胰腺癌预后模型;
通过质控和数据标准化,确定使用2个RNA-seq数据和7个微阵列数据(array)来识别胰腺癌患者中普遍存在的异常基因;
采用加权基因共表达网络(WGCNA)分析,探讨基因表达模式与临床特征之间的关系;
采用lasso和cox回归建立预后风险模型;
用过ROC曲线下面积来检验模型的预测能力;
最终确定了一个四基因signature的胰腺癌预后有biomarker
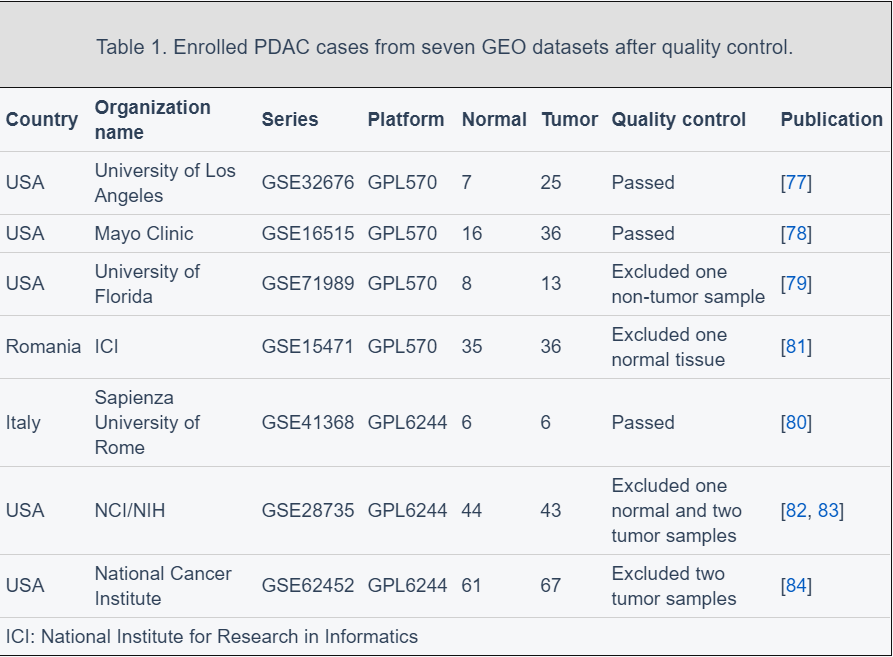
table 1.数据展示
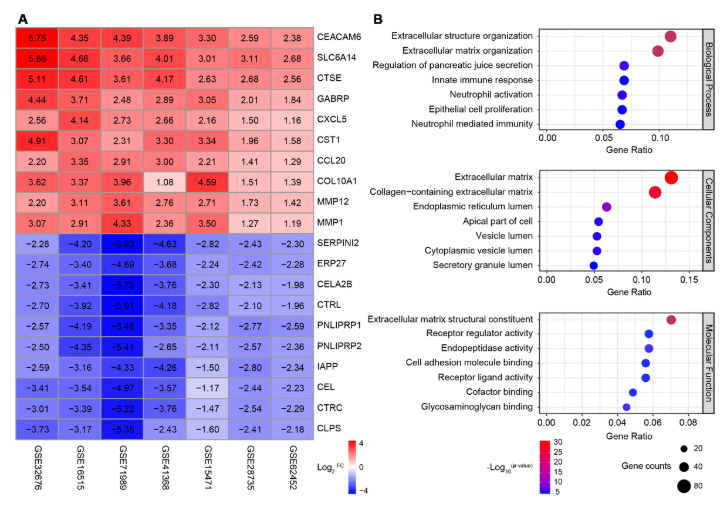
figure 1.差异基因分析和GO富集结果展示——红色和蓝色框分别表示上调和下调的基因,颜色饱和度与基因水平相关;
对所有DEG进行分析。每个类别(BP,CC和MF)中七个最丰富的GO术语列在左轴旁边。圆圈的大小表示富集基因的数量,颜色与相应的log 10 p 值相关;
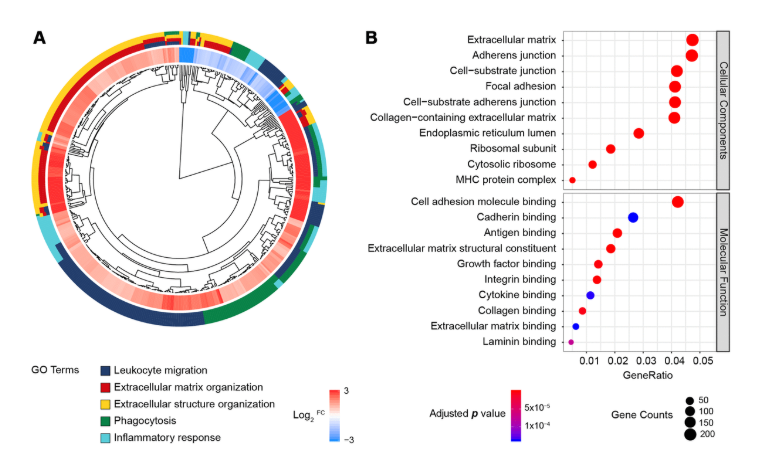
figure 2.对RNA-seq数据进行差异基因分析和GO富集分析——内部树状图表示基因表达图谱的层次聚类,外部圆代表每个DEG的log 2 FC,
其颜色对应于基因水平,最外面的圆代表分配给该基因的GO BP术语;
图B表示10个最丰富的CC和MF术语。圆圈的大小表示富集基因的数量,其颜色对应于调整后的 p值
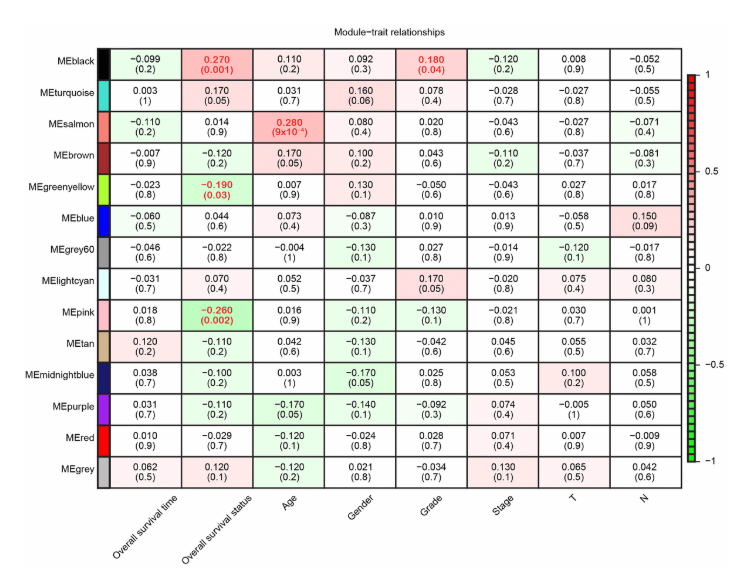
figure 3.模块特征关系——每行代表一个颜色编码的模块特征基因,每列代表临床特征,
每个单元格代表相应模块特征的皮尔逊相关系数(上位数)和 p值(在括号中)。每个单元的颜色表示相关程度。
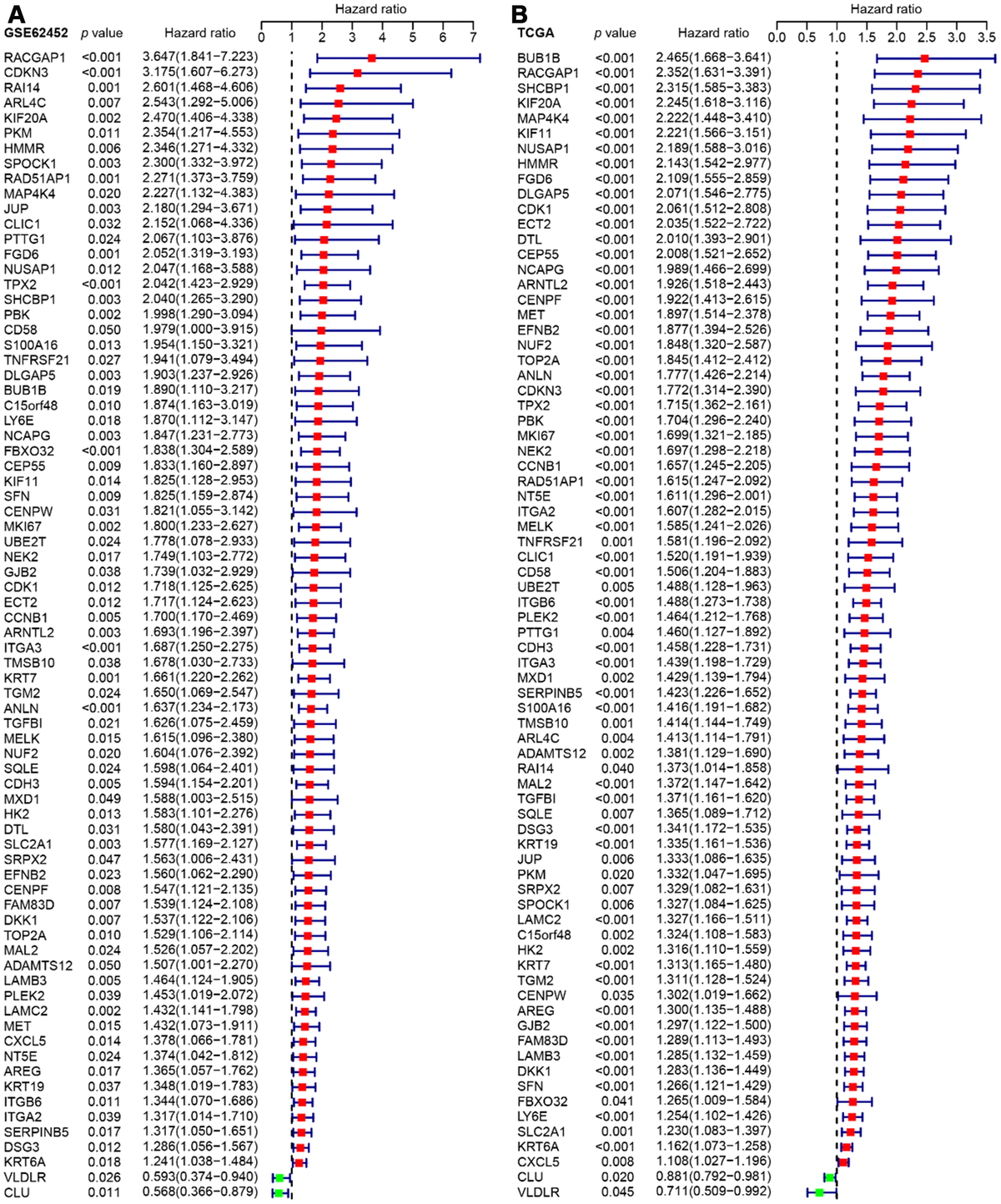
figure 4.通过GSE62452和TCGA的单变量Cox分析确定的76个预后DEG的HR的森林图。
前三列分别显示基因名称, p值以及HR和95%CI。在森林图中,保护关联显示为绿色,危险因素显示为红色。
(虽然涉及到的基因非常多,但这个图其实有更好的展示形式,好好画一画可以非常美观)
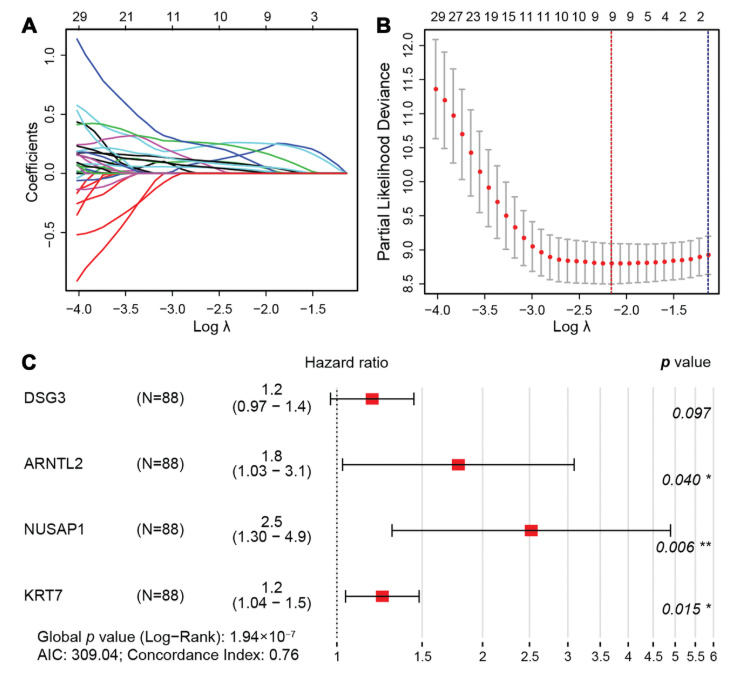
figure 5. LASSO回归模型
( A)76个预后DEG的LASSO系数曲线。每条曲线代表一个系数,x轴代表正则化罚分参数。随着λ的变化,系数变为非零值将进入LASSO回归模型。
( B)交叉验证以选择最佳调整参数(λ)。红色垂直虚线穿过最优对数λ,它对应于多元Cox建模的最小值。两条虚线代表与最小值的一个标准偏差。
( C)根据来自TCGA的训练队列的多变量Cox回归分析,四个基因的HR和95%CI。
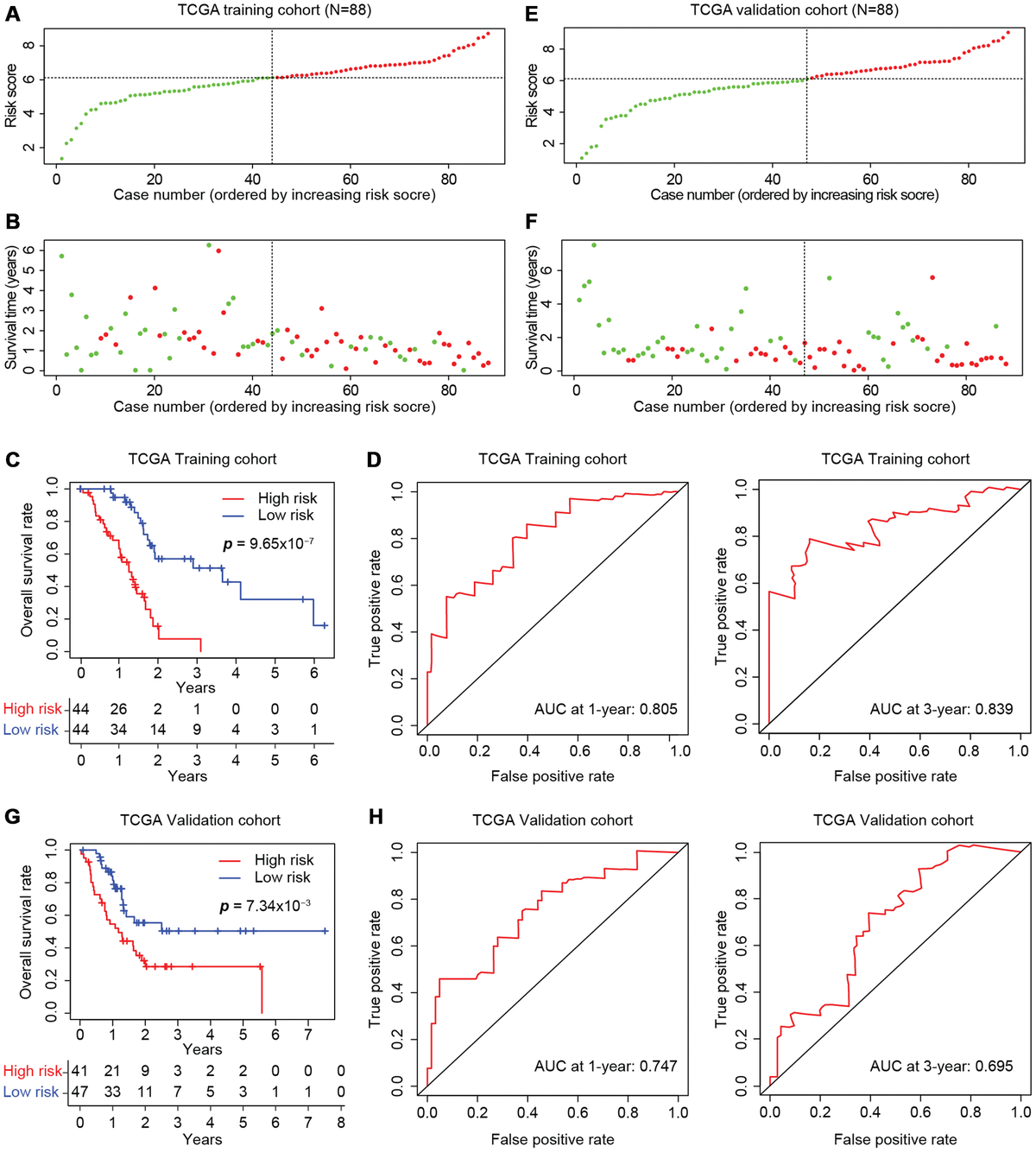
figure 6.TCGA数据的预后打分
图A和E:训练集和验证集的风险分布(水平虚线表示用于对患者进行分层的风险评分的截止水平,而垂直虚线则根据低风险(绿色)或高风险(红色)来区分患者。)
图B和F:训练集和验证集中总体生存和死亡分布
图C和G:训练集和验证集中不同风险患者的生存曲线
图H:时间相关的ROC曲线,用于预测TCGA 中训练集合验证集的一年和三年生存期
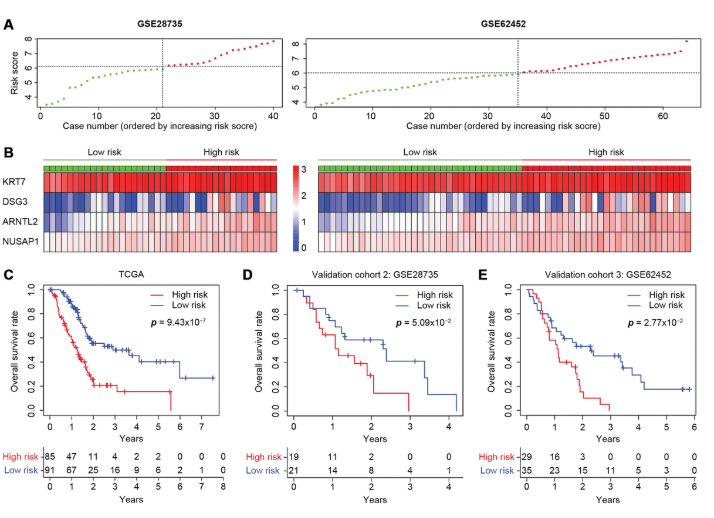
figure 7.在两个独立的微阵列数据集中对四基因模型的验证
( A)GSE28735和GSE62452队列中的风险评分分布。
( B)热图显示高风险和低风险组中四个基因的水平。如键所示,每种情况的颜色对应于基因水平的log 2 FC。
( C – E)TCGA和GSE28735和GSE62452队列中高危或低危患者的Kaplan-Meier生存图,底部显示了在特定时间点剩余的患者数量。
我们来讲一下这篇文章的优点:
l 首先这篇文章在背景部分没有花大量篇幅介绍胰腺癌如何如何,在我看来,这种文字都是用来充数的
l 因为你是生信文章,不是最新前沿治疗进展,对不?生信文章就好好把你用到的方法介绍好。
l 其次文章的图表描述非常清晰易懂,很多人都害怕把一个事情描述的清楚,或者没有能力用简单的语言描述清楚。显然作者团队没有这种担忧,他们把图表结论用简单的语言描述的很清楚,以至于我都不用怎么总结。
l 本研究涉及到了多个维度的数据(其实也就2个),建立了一个4基因的预后风险模型,并且通过了TCGA和GEO双重数据集的验证,鲁棒性很好。
l 主流的生信分析方法都有涉及到了,并且并不是凑数的,分析的结果与文章核心结论都有联系。
结束语:
我知道很多人看不起AGING,但AGING现在实打实的5.515分,今年基本还是5分水平

对于我们没有做到的事情,还是要有一些敬畏之心,先做到再说。
另外纯生信没有不行,可能是你的生信不行....
如果需要合作,请联系

- 发表于 2020-03-30 11:33
- 阅读 ( 10529 )
- 分类:文献解读
你可能感兴趣的文章
- 文章分享(五)这篇文章竟然能为癌症的治疗提供新的研究思路? 5514 浏览
