文章分享(五)这篇文章竟然能为癌症的治疗提供新的研究思路?
看教程不够直观,那就看视频吧! >>点击加载视频
端粒(Telomere)是存在于真核细胞线状染色体末端的一小段DNA-蛋白质复合体,它与端粒结合蛋白一起构成了特殊的"帽子"结构[1]。端粒有在保护染色体末端中起重要作用并防止它们被识别为DNA损伤部位[2],端粒的变短可能导致细胞衰老和死亡[3-5],维持端粒的长度可以让细胞绕过危险并能无限增殖[6]。肿瘤细胞可以通过端粒维持机制使其具有无限增殖的能力,如:端粒酶反转录酶(TERT)的激活,TERT除了具有维持端粒长度的功能,TERT还能激活与细胞生存相关的通路。但当前对于端粒酶基因在癌症中的表达模式没有深入的捕捉和刻画。在此,为大家介绍一篇发在Molecular Cancer上的关于预后相关低氧免疫分类器的文献【Pan-cancer analysis identifies telomeraseassociated signatures and cancer subtypes 2019.6.10 IF:10.679】


为实现在癌症类型和起源器官中更进一步的分析疾病之间的共性以及差异,在2012年10月26日至27日,TCGA在加利福尼亚州圣克鲁斯举行的会议上启动了泛癌(Pan-Cancer)计划。Pan-Cancer的目标是跨肿瘤类型以及跨平台收集连贯一致的TCGA数据集,然后分析和解释这些数据。Pan-Cancer提出的两个月时间内,整理了12种癌症的在基因组、蛋白质、表观以及转录组6种不同平台的数据(如Fig 1A所示)。通过泛癌分析可以发现不同肿瘤类型中的基因组和细胞改变之间的相似性和差异。
因此,在这项研究中,Luo等人基于TCGA中8种癌症(BRCA, KIRC, KIRP, LGG, LIHC, LUAD, SARC和THCA8)的mRNA表达、miRNA表达、体细胞突变以及甲基化数据,通过泛癌的多组学数据对TERT高(TERThigh)和TERT低样本组(TERTlow)进行系统的分析,去找到那些在疾病中特异以及共同的影响TERT的关键基因,并系统而深入地分析这些基因的功能。通过Luo等人的研究,他们深入刻画了在癌症中端粒酶相关的分子特征,为癌症的治疗提供新的研究思路(如Fig 1B所示)。
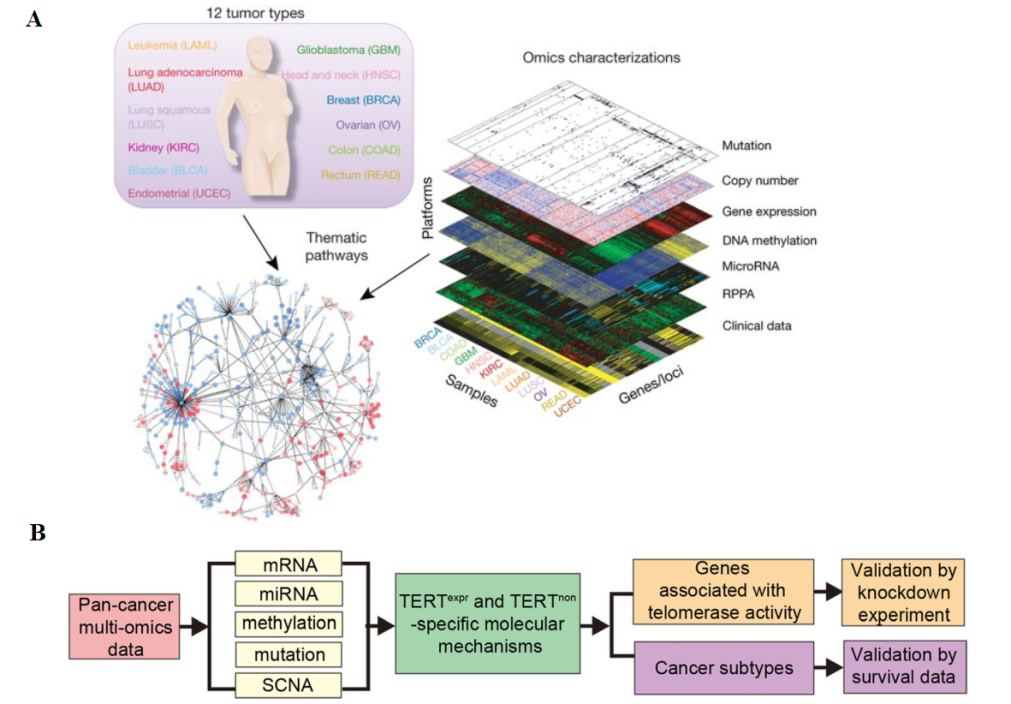 Fig 1. 研究思路
Fig 1. 研究思路

(1)TERThigh和TERTlow数据:通过Barthel等人[7]发表的工作,得提取估计端粒酶活
性的基因列表。
(2)TCGA多组学数据的获取:从cBioPortal数据库获取TCGA数据库中BRCA, KIRC,
KIRP, LGG, LIHC, LUAD, SARC和THCA8种癌症的mRNA表达数据、临床数据、甲基化数据(HM450);Firehose数据库获取miRNA表达谱数据、MAF以及SCNA数据。
(3)数据的处理:对于从cBioPortal数据库得到的癌症mRNA表达数据,进行log2转换,
并只保留那些在TERThigh和TERTlow组中平均表达值都大于1的基因进行后续分析。
(4)倾向性评分匹配权重算法(Propensity score matching weight algorithm,PSM):该算
法由Liang Li[8]提出,用来寻找TERThigh和TERTlow相关的特征。
(5)miRNA靶基因数据:从miRtarBase, miRanda和miRDB数据库获取miRNA与mRNA
互作数据,并进一步通过R包“WGCNA”识别miRNA-mRNA共表达模块。
(6)基因功能分析:通过R包“clusterProfiler”中的EnrichGO函数实现功能富集,参数
为ont = “MF”, pvalueCutoff = 0.01, qvalueCutoff = 0.05,通过“Benjamini-Hochberg”算法实现对p值的校正。其中,选择对那些在TERThigh组病人中特异突变和扩增的基因进行通路的富集分析。
(7)药物靶标数据:从Drugbank数据库获取FDA许可药物及其靶标数据,通过将TERT相关标志物映射到药靶上,从而识别潜在影响TERT功能的药物。
(8)疾病亚型的识别:基于“global markers”和“cancer-specific markers”,对样本划分成两类,并进一步通过R包“randomForest”去构建分类器识别疾病亚型。

(1)识别标志物的分布:基于TCGA数据库中BRCA, KIRC, KIRP, LGG, LIHC, LUAD, SARC和THCA8的体细胞突变、拷贝数改变、mRNA表达、DNA甲基化以及miRNA表达这5个分子层面的组学数据,通过PSM算法在各癌症中识别的标志物的分布如Fig 2所示。
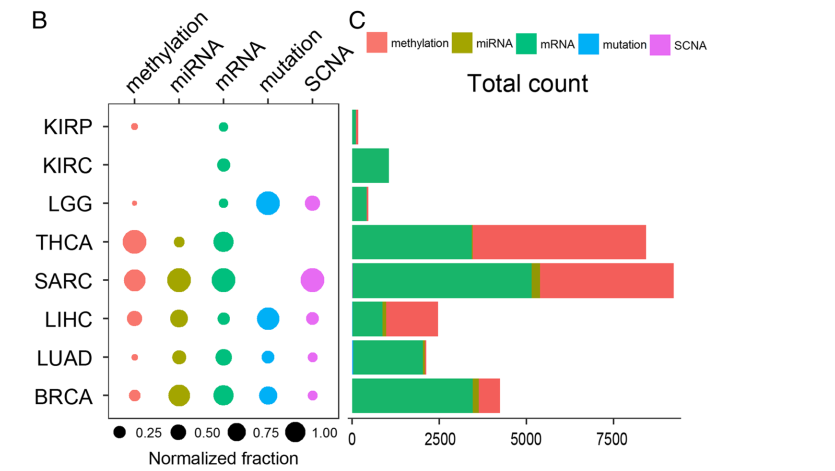 Fig 2. 各组学标志物在不同癌症中的分布情况。
Fig 2. 各组学标志物在不同癌症中的分布情况。
(2)TERThigh和TERTlow中特异表达的基因功能富集分析:基于甲基化数据和mRNA表达谱数据,识别在TERThigh和TERTlow中特异表达的基因,并进一步通过R包“clusterProfiler”分别去展示在TERThigh和TERTlow两组中分别特异表达基因的功能,发现TERThigh和TERTlow两组病人中特异的基因所影响的功能确实存在差异,如图Fig 3所示。
 Fig 3.基因功能富集分析
Fig 3.基因功能富集分析
A)TERThigh组中特异基因的参与的功能展示;B)TERTlow组中特异基因的参与的功能展示。
(3)对模块中基因进行分析:基于上述8中癌症的mRNA表达谱数据,通过WGCNA算法在每个癌症中识别出相应的功能模块,在BRCA,KIRC,KIRP,LGG,LIHC,LUAD,SARC和THCA中分别得到38,63,76,70,52,101,47和 58共表达模块,并对这些模块中的基因进行功能富集分析,发现这些基因参与的功能可以大致分为四类,即细胞分裂、RNA剪切、细胞外基质以及凋亡/血管生成,这些结果进一步说明TERT和细胞周期以及RNA的功能相关。
(4)对模块中hub基因进行研究:对上述模块中的基因,计算其连通度,对连通度较大的EXO1以及TPX2进行敲除实验,发现基因的敲除使得端粒的活性和细胞的生存能力降低,如Fig 4所示。
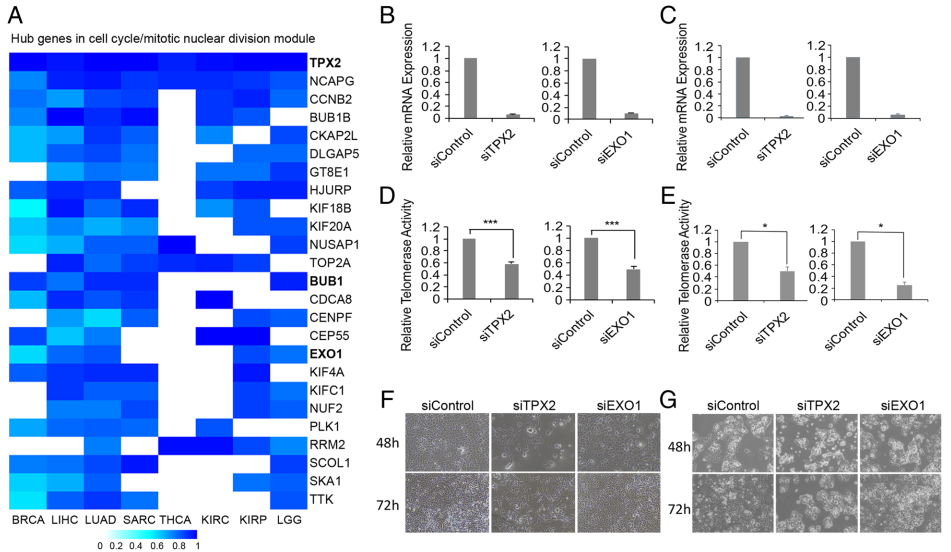 Fig 4. Hub基因对端粒酶的影响。
Fig 4. Hub基因对端粒酶的影响。
A)各癌症中模块基因连通度展示;B-D)EXO1以及TPX2敲除后端粒酶活性以及细胞生长力的展示。
(5)TERThigh组特异的miRNA簇机制分析:miRNA作为一种重要的转录后调控因子,在Luo等人的研究中,发现在TERThigh和TERTlow组中特异的miRNA和mRNA的表达呈现负相关(如Fig 5A),之前的研究表明miR-19b和TERT的表达相关,而miR-19b是从miR-17-92中转录来的,因此对miR-17-92簇在TERThigh和TERTlow组差异表达(如Fig 5B),miR-17-92簇调控网络如Fig 5D所示。
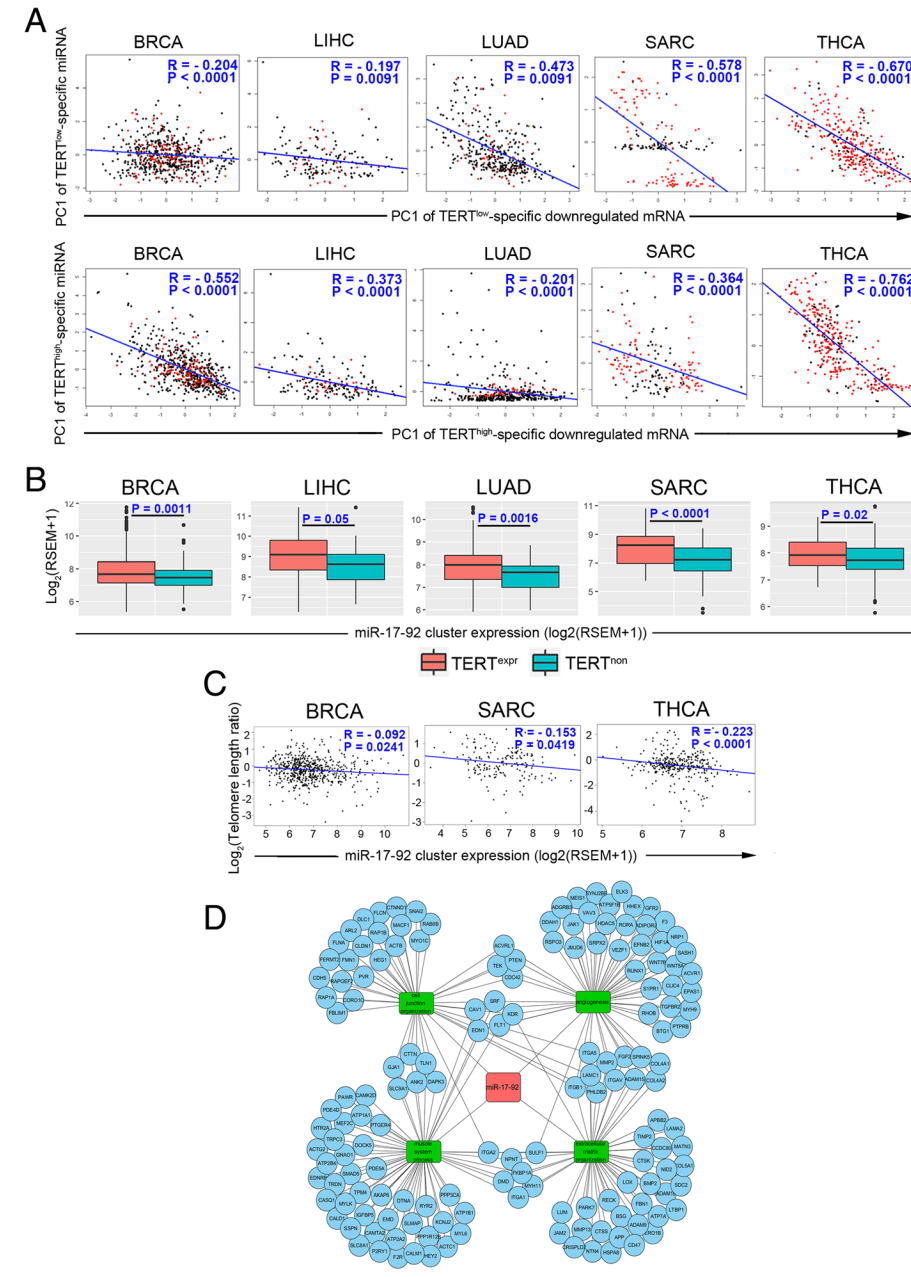 Fig 5. 癌症中影响TERT的miRNA簇分析
Fig 5. 癌症中影响TERT的miRNA簇分析
(6)TERT突变特征刻画:结合体细胞突变数据、拷贝数数据和mRNA表达数据,识别和TERT相关的标志物(如Fig 6所示),进一步结合药物靶标数据,从TERT相关标志物中作为药物靶点的标志物,并刻画药物和TERT相关标志物的关系(如Fig 7所示)。
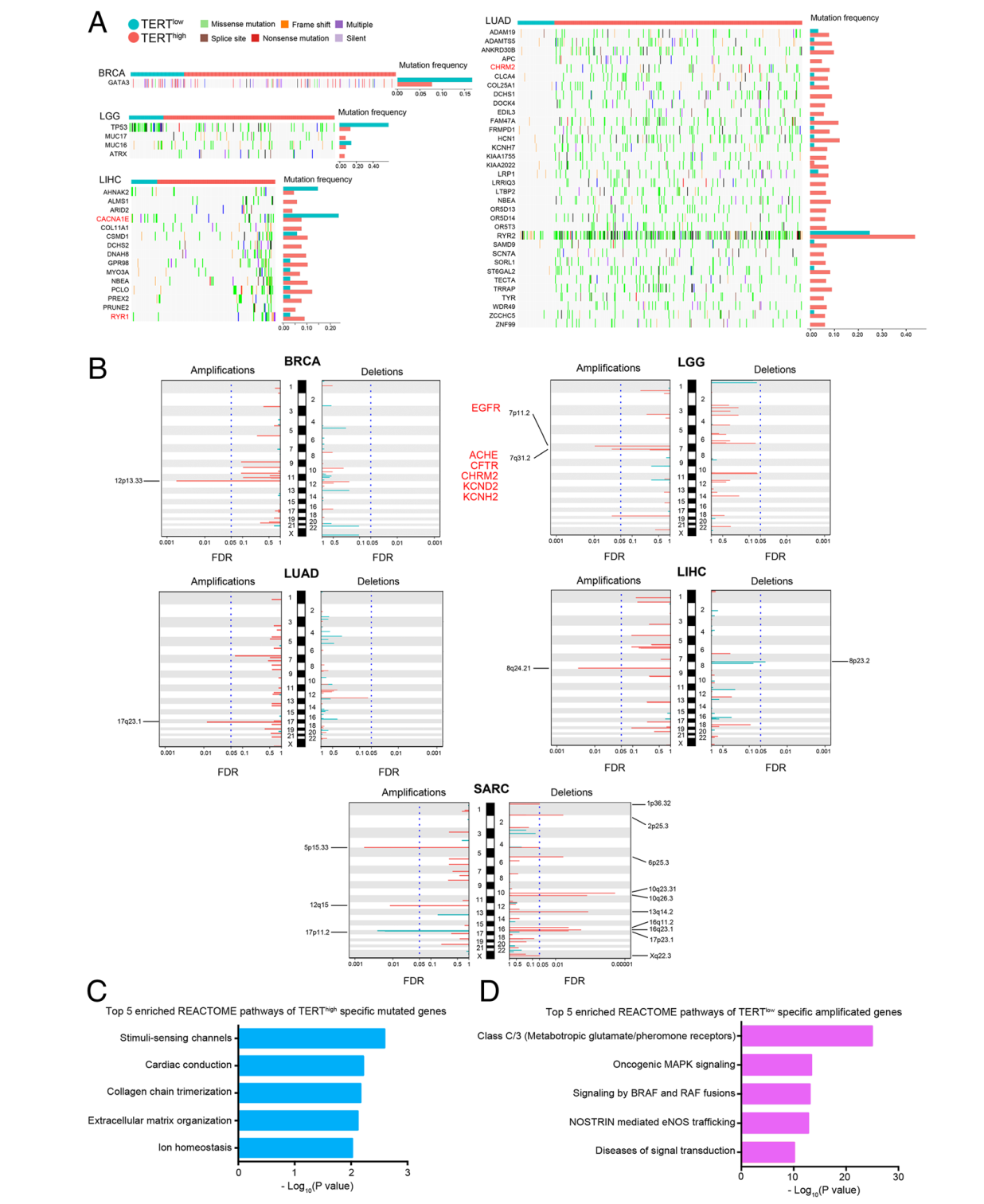 Fig 6. TERThigh和TERTlow组特异改变特征刻画。
Fig 6. TERThigh和TERTlow组特异改变特征刻画。
A)突变特征;B)拷贝数特征;C)Top 5突变基因功能展示。其中,红色的基因是FDA药物作用靶标。
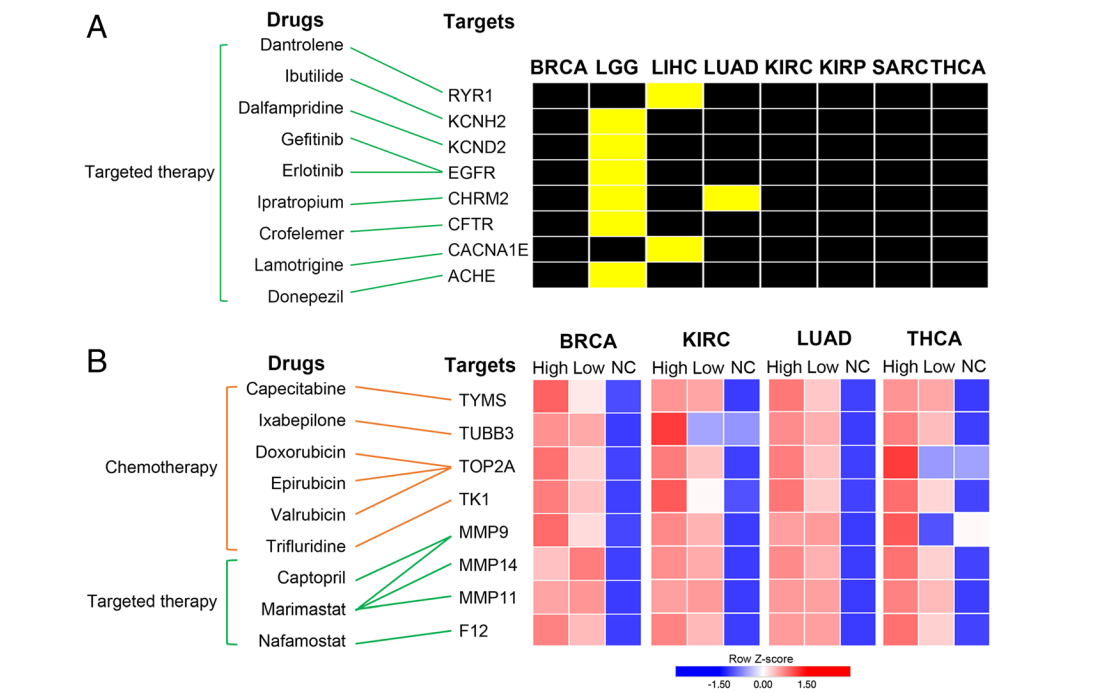 Fig 7. TERThigh和TERTlow组特异marker与药物对应关系
Fig 7. TERThigh和TERTlow组特异marker与药物对应关系
(7)癌症亚型的划分:整合在WGCNA识别的模块中参与细胞分裂相关的功能中模块中的所有基因与在8种癌症中识别到的与TERT相关的top前20基因,通过“randomForest”分成两类,即:“RFcluster1”和“RFcluster2”,对这两组病人的TERT活性进行分析,同时,利用该分类器在8组癌症病人中划分相应的亚型,并刻画两组病人亚型在8种癌症中的生存情况(如Fig 8所示)。
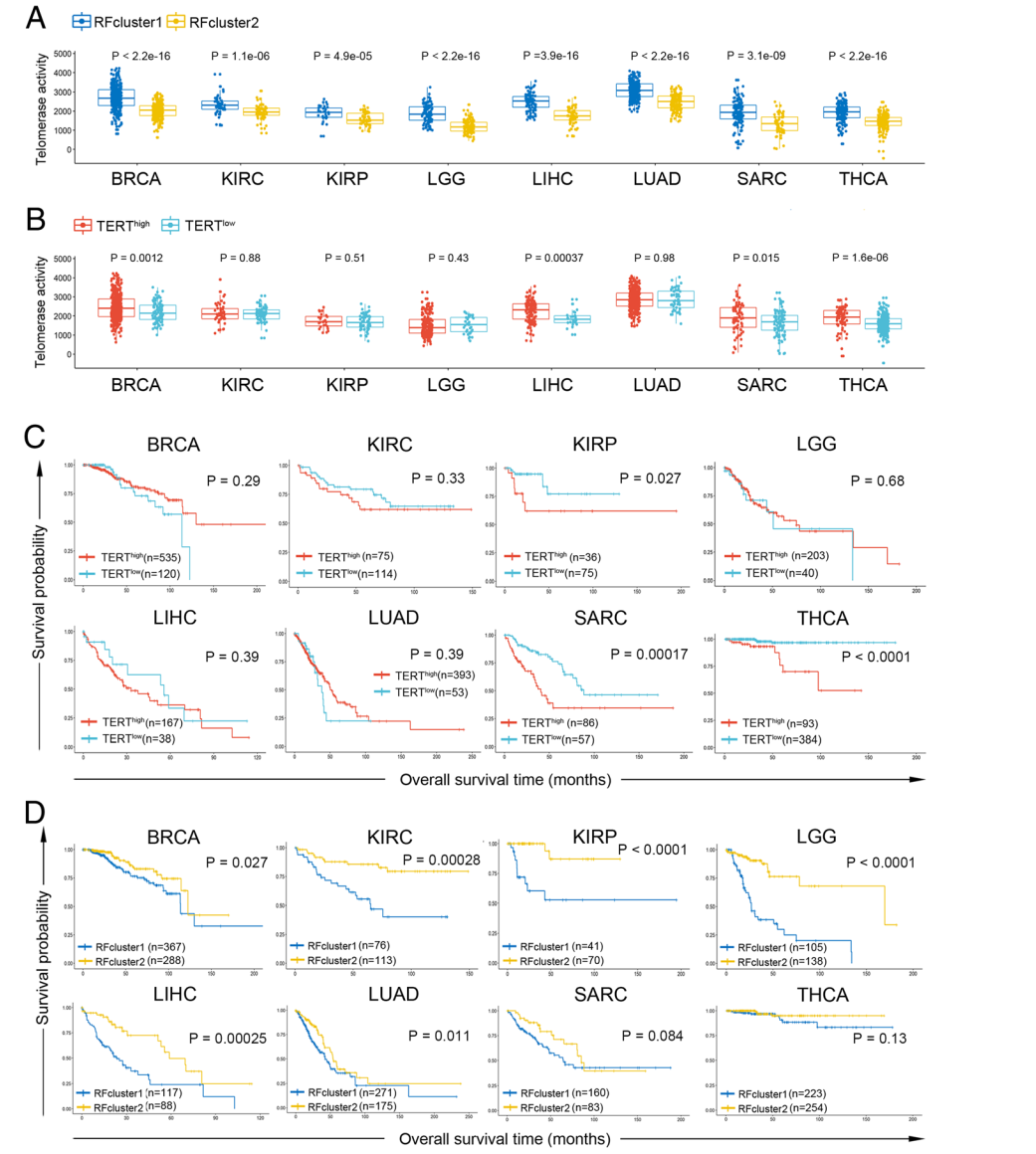 Fig 8. 癌症亚型的刻画
Fig 8. 癌症亚型的刻画
参考文献:
1. Osullivan R J, Karlseder J. Telomeres: protecting chromosomes against genome instability[J]. Nature Reviews Molecular Cell Biology, 2010, 11(3): 171-181.
2. Neumann A A, Reddel R R. Telomere maintenance and cancer ? look, no telomerase[J]. Nature Reviews Cancer, 2002, 2(11): 879-884.
3. Harley C B, Futcher A B, Greider C W, et al. Telomeres shorten during ageing of human fibroblasts[J]. Nature, 1990, 345(6274): 458-460.
4. Harley C B, Vaziri H, Counter C M, et al. The telomere hypothesis of cellular aging.[J]. Experimental Gerontology, 1992, 27(4): 375-382.
5. De Lange T. How Telomeres Solve the End-Protection Problem[J]. Science, 2009, 326(5955): 948-952.
6. Neidle S, Parkinson G N. Telomere maintenance as a target for anticancer drug discovery[J]. Nature Reviews Drug Discovery, 2002, 1(5): 383-393.
7. Barthel F P, Wei W, Tang M, et al. Systematic analysis of telomere length and somatic alterations in 31 cancer types[J]. Nature Genetics, 2017, 49(3): 349-357.
8. Li L, Greene T. A Weighting Analogue to Pair Matching in Propensity Score Analysis[J]. The International Journal of Biostatistics, 2013, 9(2): 215-234.

文章分享(三)iCCA的亚型与基因组、表观组、预后以及细胞来源的差异的关系
文章分享(四)CT基因的表达与癌症的亚型、分子机制、调控以及预后之间的关系
- 发表于 2019-07-25 15:13
- 阅读 ( 5507 )
- 分类:文献解读
你可能感兴趣的文章
- 文献解读(1)癌症干性,肿瘤内异质性和跨癌症的免疫反应 11527 浏览
- 2020年5+生信文章什么样? 10521 浏览
