你的这个截图可以看出 人家是用的配对 T检验,如果你和他用的是同一套数据的话,可能使用limma就筛不出来,大概率问题在于foldchange普遍比较小,你试试 使用1.5倍,或者1.3倍筛选

首先下载原始数据,做了merge,没有教程中的normalized数据,是gprocesssignal,请问是什么意思?
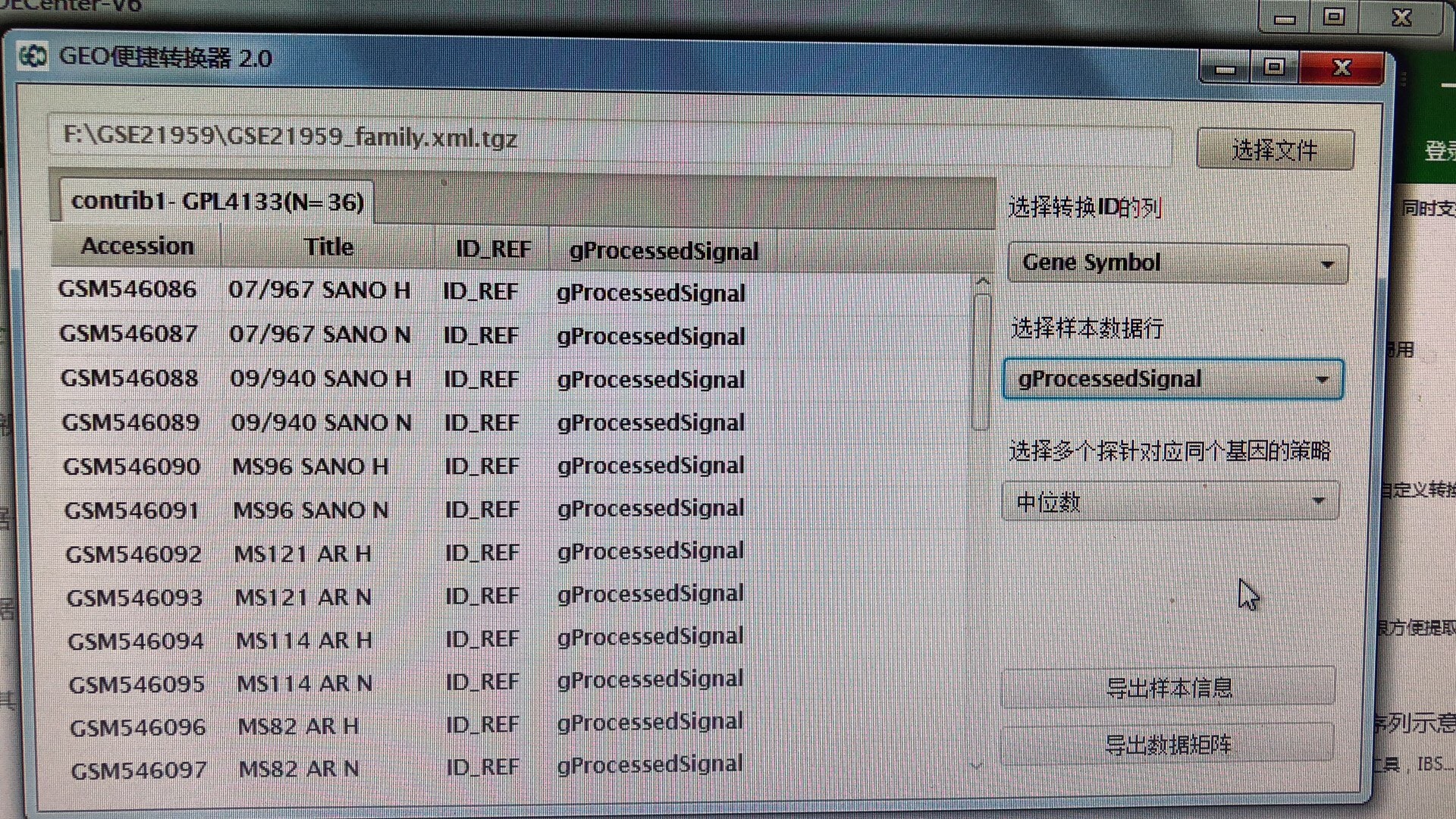 也没有unigene symbol,只有gene symbol,merge完数据,进行分组,做limma、T以及原文用的配对T,在原文条件下也没有获得差异基因。
也没有unigene symbol,只有gene symbol,merge完数据,进行分组,做limma、T以及原文用的配对T,在原文条件下也没有获得差异基因。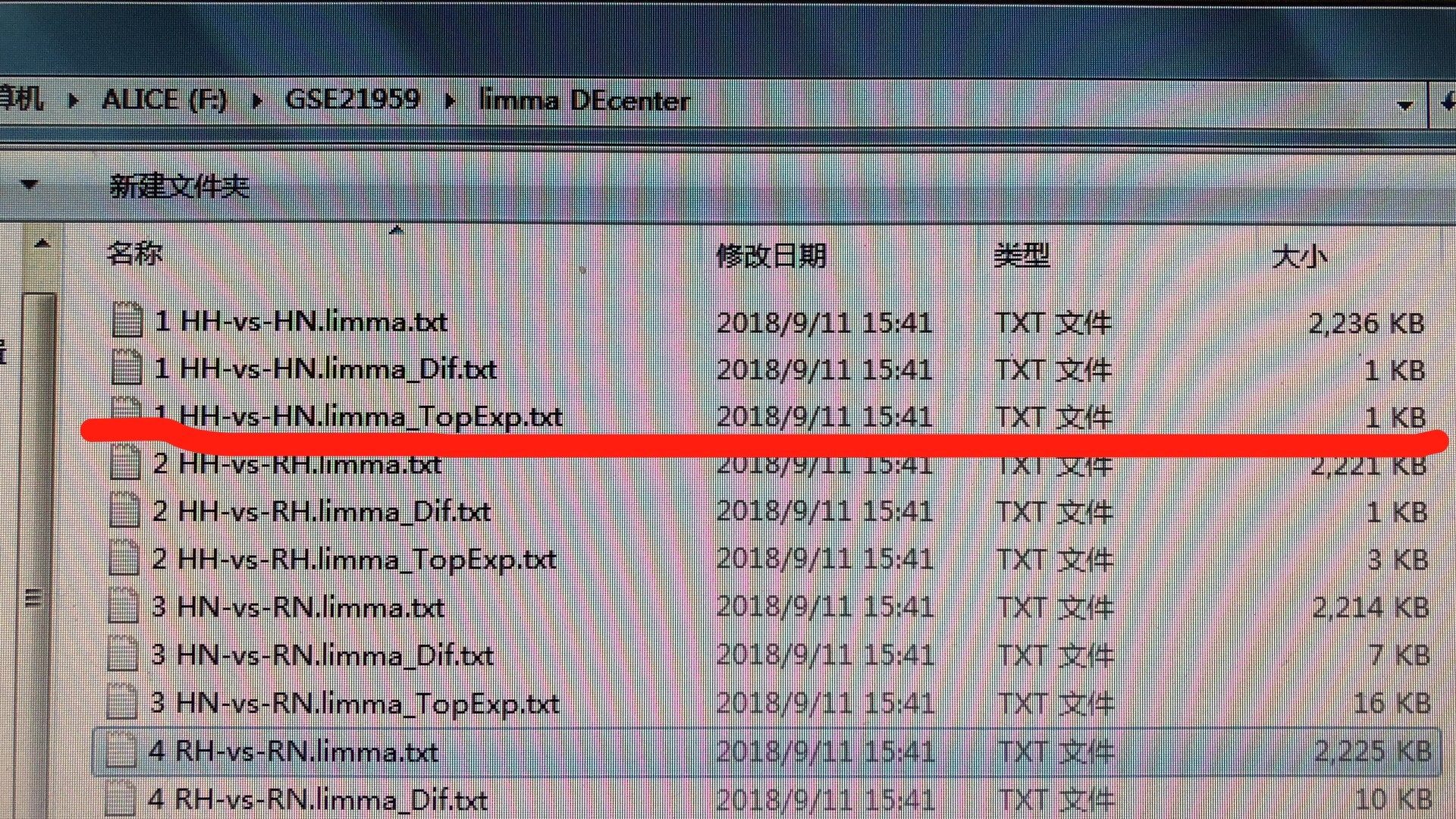
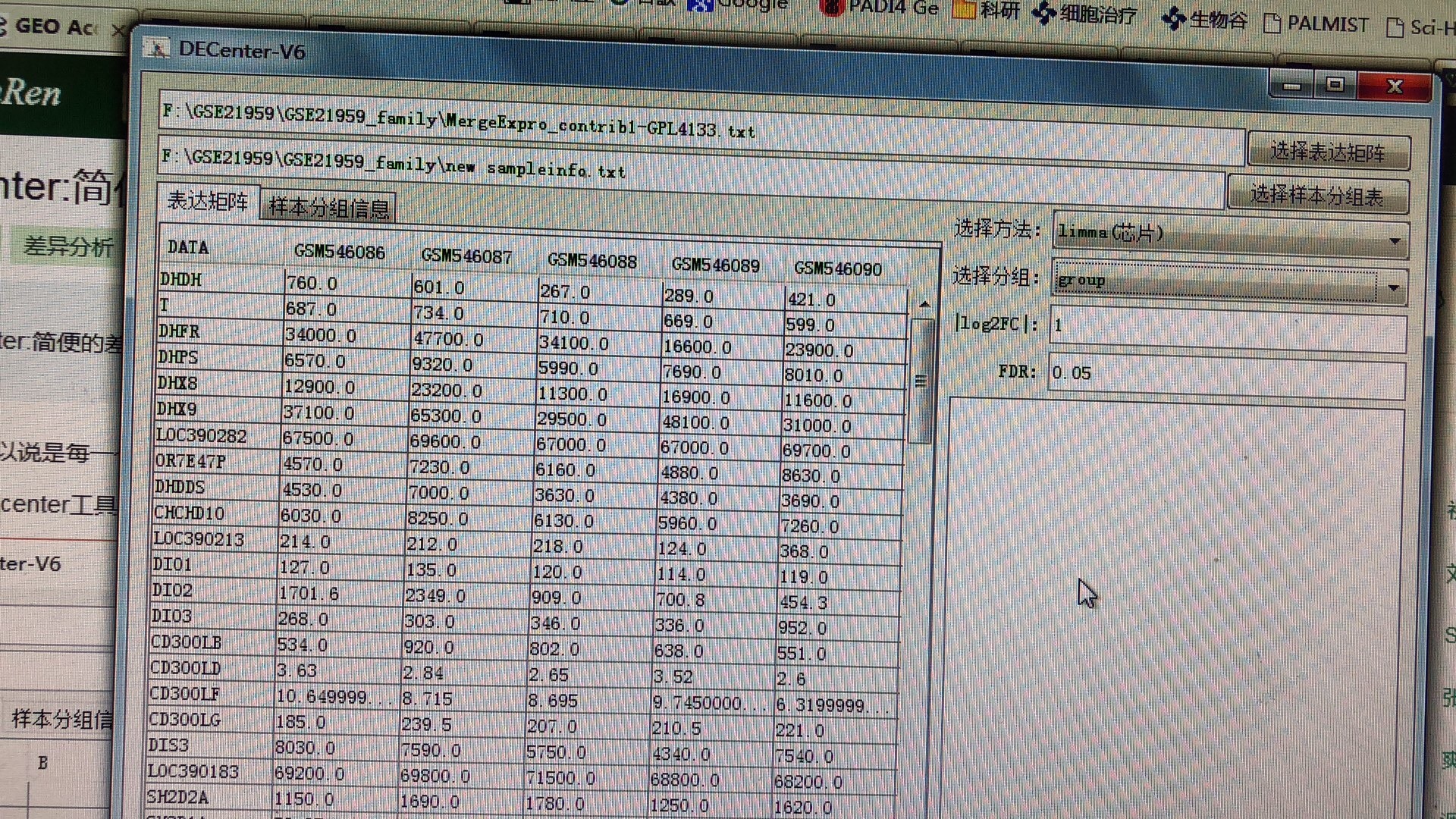
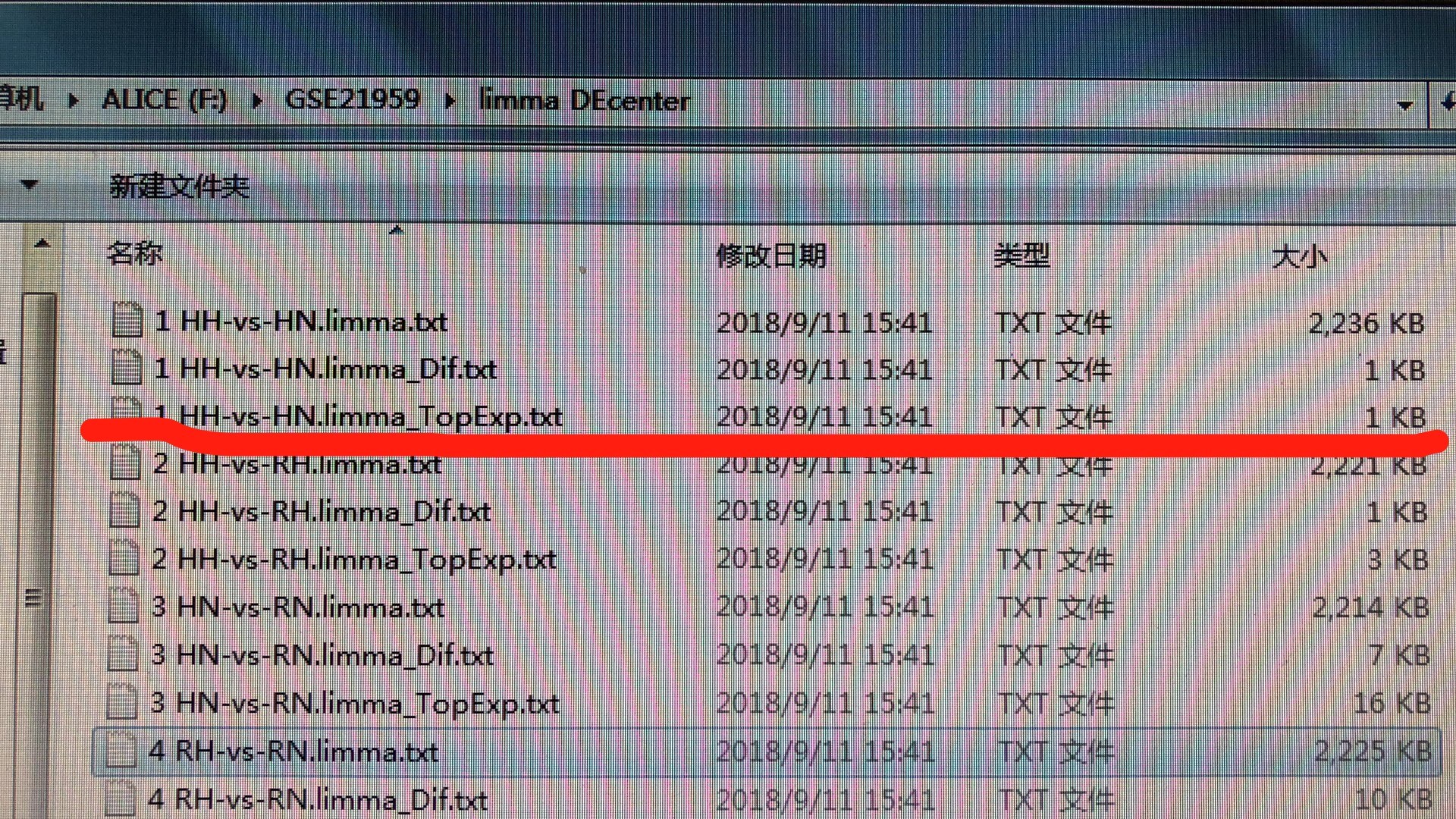
我知道是我设置条件太苛刻了,但是FDR的放宽可以放宽到多少呢?困惑。。。
