没有转化的呢,都是原始的,这里你可以下载.nocnv的
- 1 关注
- 0 收藏,5996 浏览
- Bubbles 提出于 2018-10-10 09:17
相似问题
- 请问为什么生信人没法下载TCGA数据? 1 回答
- TCGA下载临床数据后合并数据后怎么转换到Excel表格 1 回答
- 请问在TCGA数据库中的RNA-seq中下载的mRNA基因表达谱数据是芯片数据还是测序数据,谢谢 1 回答
- 现在的桑格盒子下载TCGA临床信息后,如何合并为矩阵文件? 1 回答
- 老师您好,v1.0.9版本提示本地无法连接TCGA数据库 1 回答
- 从GEO数据库下载下来的甲基化芯片要什么处理呢?下载来的芯片,通过R语言处理后得到了甲基化差异区域,通过注释获取了相关基因。但是我不明白什么这些区域哪些是低表达还是高表达的,对于基因的影响,希望大佬们能帮忙一下,好几天了很绝望啊 3 回答
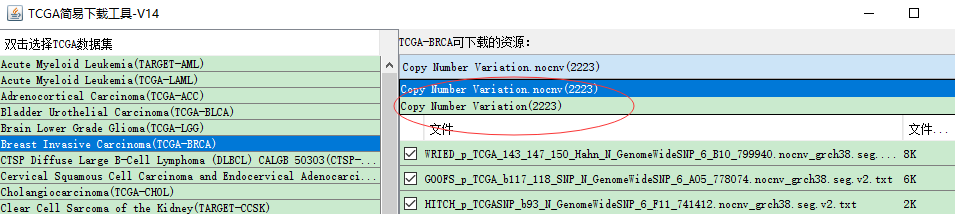 做差异分析应该选择下载图中哪种类型的数据?
做差异分析应该选择下载图中哪种类型的数据?