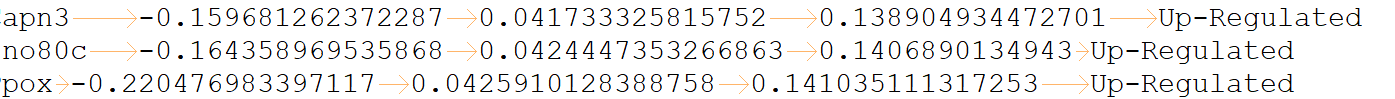这是DECenter中的代码:
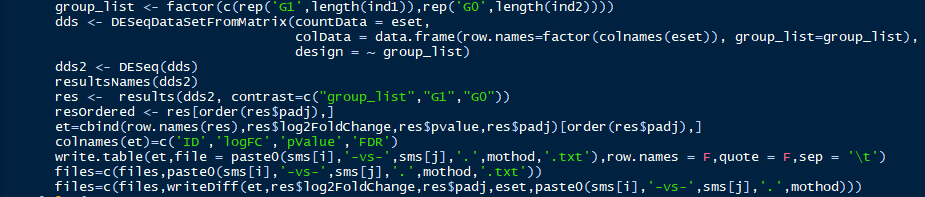 没有做预处理,用户给什么运行什么
没有做预处理,用户给什么运行什么
我用DECenter中的DESeq2 做差异分析,和我自己在R 中用DESeq2做差异分析得到的结果有比较大的出入,请问小程序中的DESeq2有做pre-filtering吗?
library(rlang)
library(DESeq2)
cts=as.matrix(read.csv("gene_count_matrix.csv", row.names=1, sep="\t"))
coldata=read.csv( "anno.csv" ,row.names=1)
dds <-DESeqDataSetFromMatrix(countData = cts,colData = coldata, design =~condition)
dds$condition <- factor(dds$condition, levels = c("A","B","C","D"))
dds=DESeq(dds)