文献解读(4)识别与m6A相关的mRNA Signature 来预测胰腺癌患者的预后情况
看教程不够直观,那就看视频吧! >>点击加载视频
点击"生信学霸"添加关注,我们一起进步
The m6A-Related mRNA Signature Predicts the Prognosis of Pancreatic Cancer Patients.
识别与m6A相关的mRNA Signature 来预测胰腺癌患者的预后情况
发表期刊:Mol Ther Oncolytics
发表日期:2020 Apr 29
影响因子:5.71
DOI: 10.1016/j.omto.2020.04.011
01—
研究背景
6-甲基腺嘌呤(N6-methyladenosine即m6A)具有重要的转录修饰作用,可以控制癌症的自我更新和细胞代谢途径。m6A是由一个甲基转移酶复合物编码的,它包含三个同源基因,即Writers,Erasers和Readers。mRNA修饰在基因表达过程中对转录后的调控至关重要,m6A可以修饰调节mRNA代谢的不同阶段,包括折叠,翻译,编译等过程,因此可以调节许多生物过程。大量研究表明,m6A能够控制癌症自我更新和细胞代谢过程,是哺乳动物mRNA中存在的最多的转录后修饰之一,m6A修饰与肿瘤发生、发展有关,容易引起乳腺癌,肺癌,成胶质细胞瘤等各种癌症。
胰腺癌(PAAD)是很强的致死性癌症,尽管最近已经取得了很大的进展,但是患者5年生存率仍然很低。尽管通过手术治疗能够很好的改善患者的生存,但是由于胰腺癌早期的症状不明显,使大多数PAAD患者错过了肿瘤切除的机会。此外,通常以TNM分期系统为指导的辅助治疗(如化疗和放疗),也不能有效改善患者预后。因此,作者通过公共数据库如TCGA和ICGC中患者的临床数据和基因表达数据,根据m6A调节因子对不同癌症亚型和临床分期患者的调控能力不同,使用LASSO-cox回归建立了一个gene signature,能预测和改善PAAD患者的预后情况。
02—
材料和方法
1. 数据来源
训练集:TCGA数据库PAAD转录组数据和体细胞突变数据以及相应的临床表型信息
验证集:ICGC数据库来自澳大利亚PAAD转录组数据(对数据进行normalized和log2转化),来自cBioPortal体细胞突变数据以及相应的临床表型信息
2. 筛选m6A相关的差异基因
将TCGA训练集分为两组,m6A突变组和无突变组,使用R包limma在两组之间筛选差异基因(DEGs),阈值p<0.05,FC>2,使用R包pheatmap绘制热图。
3. DEGs富集分析和计算免疫细胞评分
使用在线网站Metascape计算GO富集分析与KEGG富集分析,使用CIBERSORT算法计算PAAD每个样本中免疫细胞评分,并且使用R包vioplot可视化相应的结果。
4. 构建预后模型
使用单变量COX回归分析从DEGs中筛选与预后显著(p<0.01)的基因,并通过lasso-cox回归建立基因预后模型。使用R包survival绘制K-M曲线,并通过 log-rank检验计算不同组之间的生存差异,绘制K-M曲线。使用R包time ROC计算模型的ROC,最后通过单变量和多变量Cox回归模型检验风险评分的独立预后能力。
03—
主要结果
1. m6A导致TCGA-PAAD患者体内基因突变
作者首先统计了TCGA-PAAD患者体细胞突变数据中三类(即Writers,Erasers和Readers)m6A的突变类型患者的比例,其中Writer中基因VIRMA突变频率最高, 其次是基因HAKAI(4%)、基因THDF3 (3.4%)、基因IGF2BP1(3.4%)和ALKBH5(2.7%)(图1A;表1)。
表1 统计PAAD患者中m6A调控因子不同突变模型(n=149)
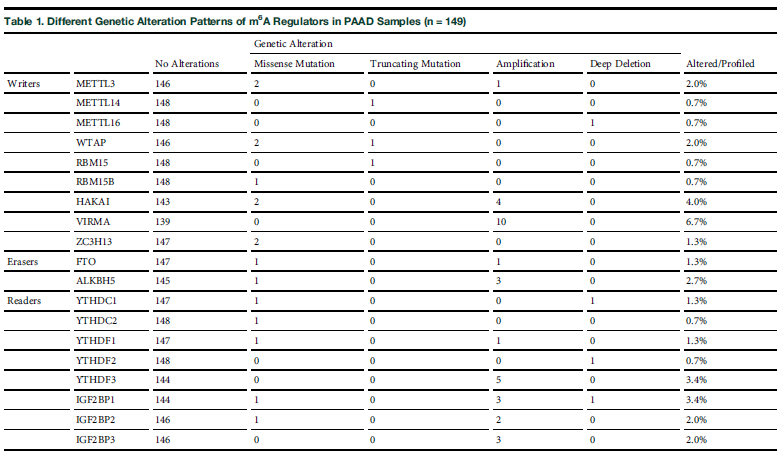
由m6A引起的CNVs中,主要以插入和缺失的方式改变基因结构,剩余的75.17%的PAAD患者没有受到m6A调节因子的影响而改变基因结结构(图1C)。最后,作者在TCGA训练集中根据m6A是否发生突变,将患者分为两组即突变组和无突变组,并且对两组患者进行OS生存分析和DFS无病生存率分析,结果显示两组患者之间的OS生存结果存在显著差异(p<0.05),DFS无病生存率没有差异(图1D-E)。
2. 分析m6A导致基因突变与无突变患者两组中临床变量的差异
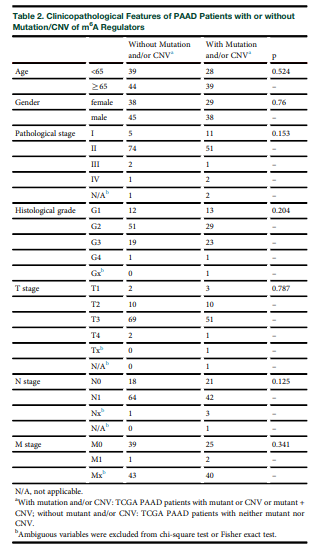
作者将PAAD患者分为基因突变型患者和无突变型患者,使用训练集携带的临床信息,分析两组患者中临床病理之间的关系,结果表明在年龄,性别,病理分期,组织学分级或TNM分期两组患者之间没有差异(表2)
表2 基因突变与无突变患者两组中临床变量统计表
3. DEGs差异分析与免疫评分分析
使用limma包在训练集基因突变组和无突变组两组之间共找到196个DEGs(图2A),进一步对这些基因进行GO与KEGG功能富集,结果显示这些基因参与了胰腺的分泌,运输等功能(图2B-D)。最后,作者还分析了不同状态下的患者的免疫评分(2E),结果表明在uncommitted macrophages(M0)、 alternatively activated macrophages (M2)和cluster of differentiation (CD)4+ memory resting T cells 在无突变组中免疫评分要显著高于突变组,而其他免疫细胞评分在两组中无差异。
4. 在训练集中构建预后gene signature
将TCGA所有的样本根据是否由m6A引起的基因突变,分为突变组和无突变组,两组在年龄,性别,病理分期,组织学分级或TNM分期等临床信息上无显着差异(表2)。作者首先在训练集中使用单因素cox比例回归分析,结果有21个DEGs与预后显著相关。然后使用LASSO回归通过对lambda(λ)值进行log2变换(图3A)和10倍交叉验证(图3B)选择最佳的阈值,得到16个风险基因对PAAD患者的生存情况进行预测,这些基因中有6个基因表现为上调,10个基因表现为下调。最后,使用LASSO-Cox回归模型,构建了一个16个gene signature来对每个PAAD患者进行风险评分,这16个基因的风险评分公式如下:Risk score=(−0.07744×CDHR3)+(−0.09939×CELSR3)+(0.29421×EGF)+(0.29019×FGF10)
+(−0.60946×GAD1)+(0.08812×MT1H)+(0.09277×NMUR2)+(−0.48445×PAH)+(0.05361×PGC)+(−0.23567×PGM5)+(−0.33405×POPDC2)+(0.06505×PPFIA3)+(0.05930×SERPINA4)+(−0.37688×TMEM145)+(0.20989×TNNT1)+(0.38926×ZPLD1)。
5. 基因风险评分与PAAD患者生存率之间关系
接下来,作者使用训练集携带的临床变量和Gene signature做OS和DFS做单因素和多因素cox回归,结果如表3所示;
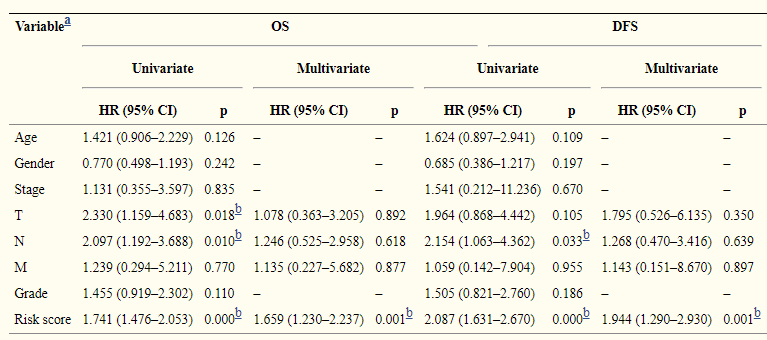
进一步在训练集中进行1年、2年、3年的ROC曲线分析以评估预后gene signature对PAAD患者的预后预测的准确性,结果显示1年、2年和3年的AUC分别为0.752、0.586和0.628(图3C)。最后,使用基因风险模型得分的中值将PAAD患者分为低风险组和高风险组。PAAD高风险组的OS较差(p <0.001;图3D)。在验证集(ICGC队列)中,作者得出与训练集相似的结论,1年、2年和3年ROC分析的AUC分别为0.535、0.636和0.602(图3E),高风险组的预后较差(p = 0.043)(图3F)
6. 分析风险模型中单个基因对患者生存情况的影响
作者讲下来分析风险模型中每个gene对患者生存情况的影响,结果如下所示:PAH (HR = 0.66; log-rank p = 0.048), ZPLD1 (HR = 1.8; log-rank p = 0.008), PPFIA3 (HR = 0.58; log-rank p = 0.0095), and TNNT1 (HR = 1.7; log-rank p = 0.0084) 这些基因可作为PAAD患者独立的预后因子(图1A-D)。最后作者还分析了基因ZPLD1和PPFIA3不同,PAH和TNNT1在TCGA数据库PAAD患者正常胰腺组织和肿瘤组织之间表达的差异(图4E-F)
04—
结果图
图1 PAAD患者中m6A导致不同突变模型以及不同突变模型对OS与DFS的影响
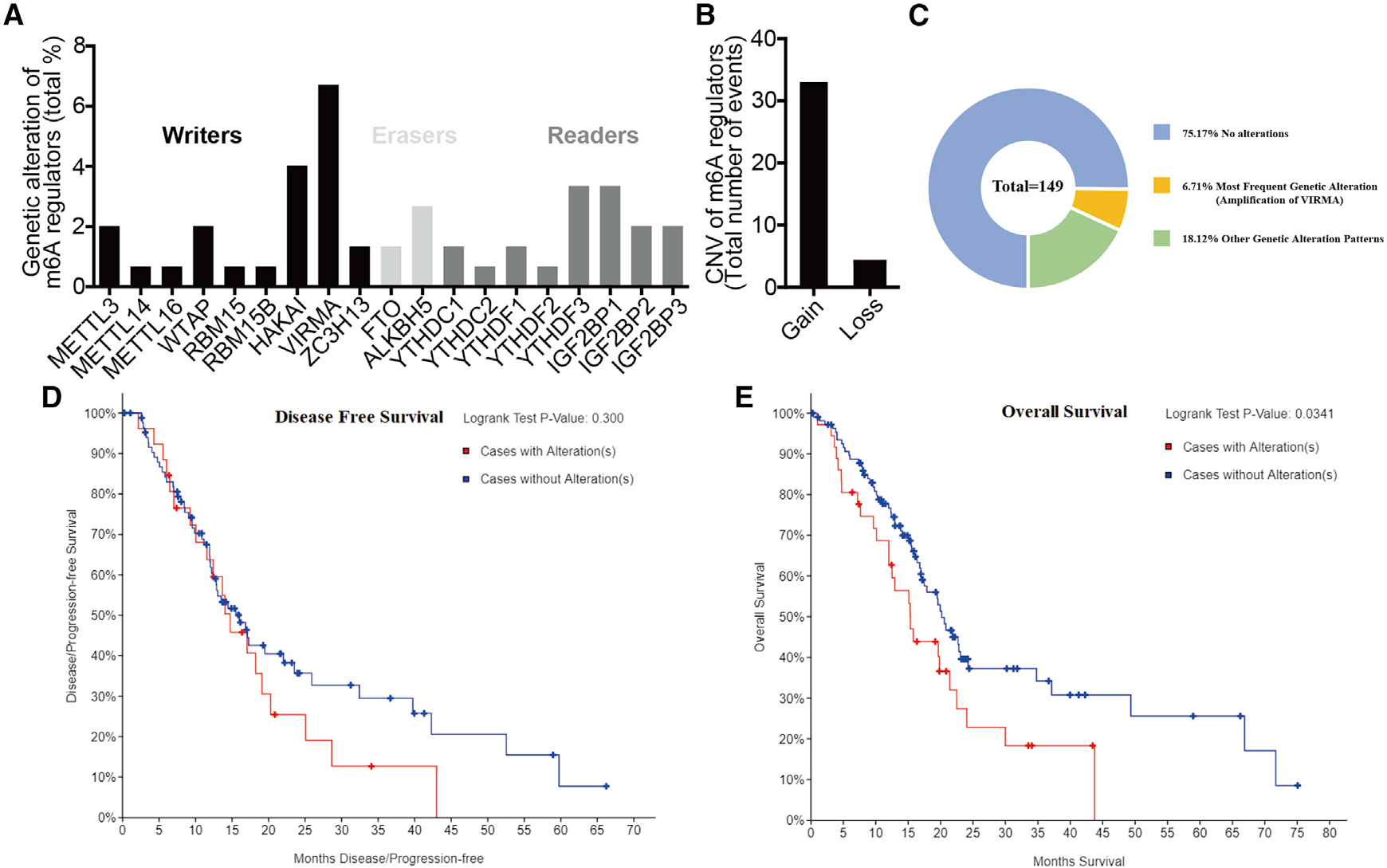
图2 m6A导致基因突变组和无突变组之间差异分析
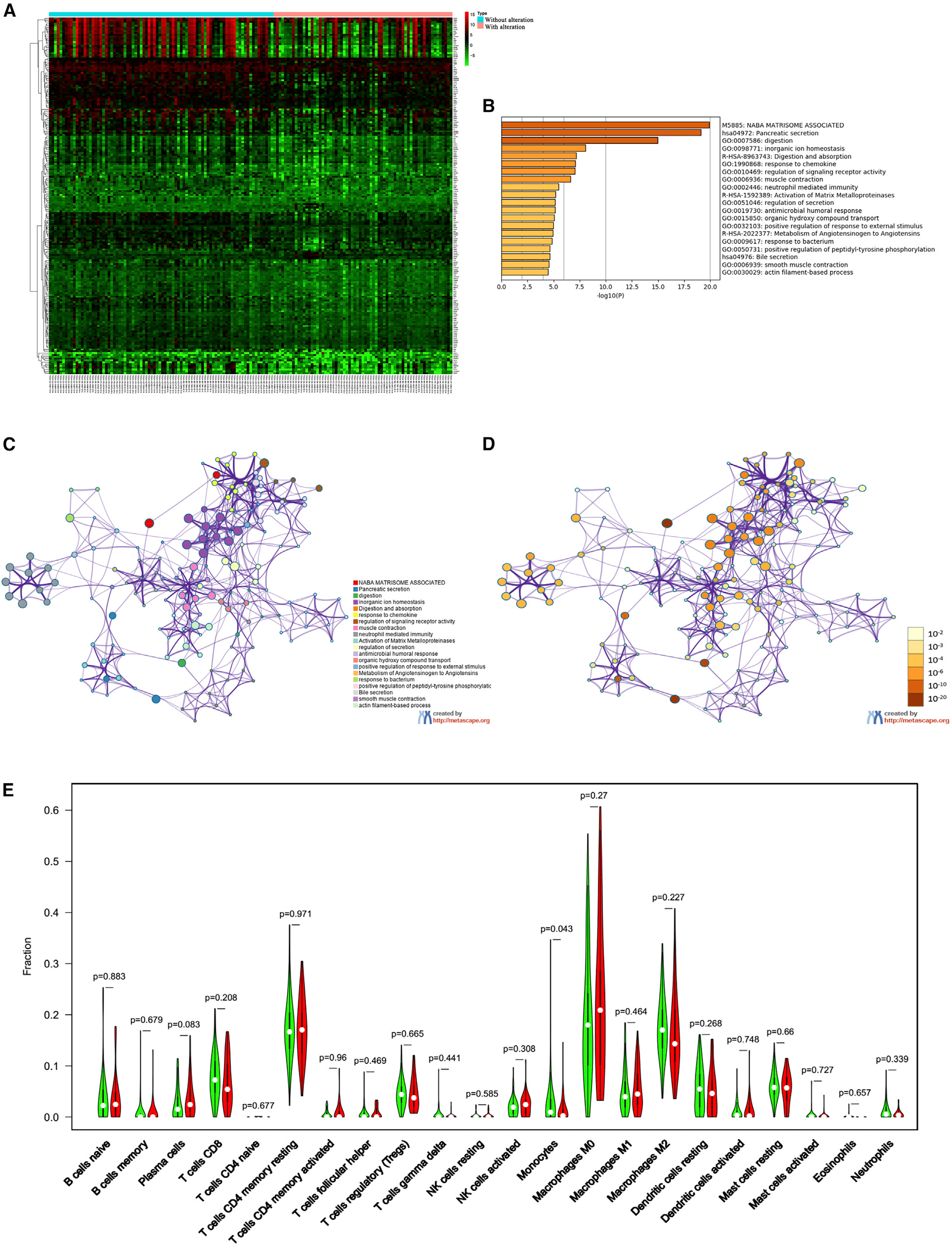
图3 基于DEGs构建风险模型
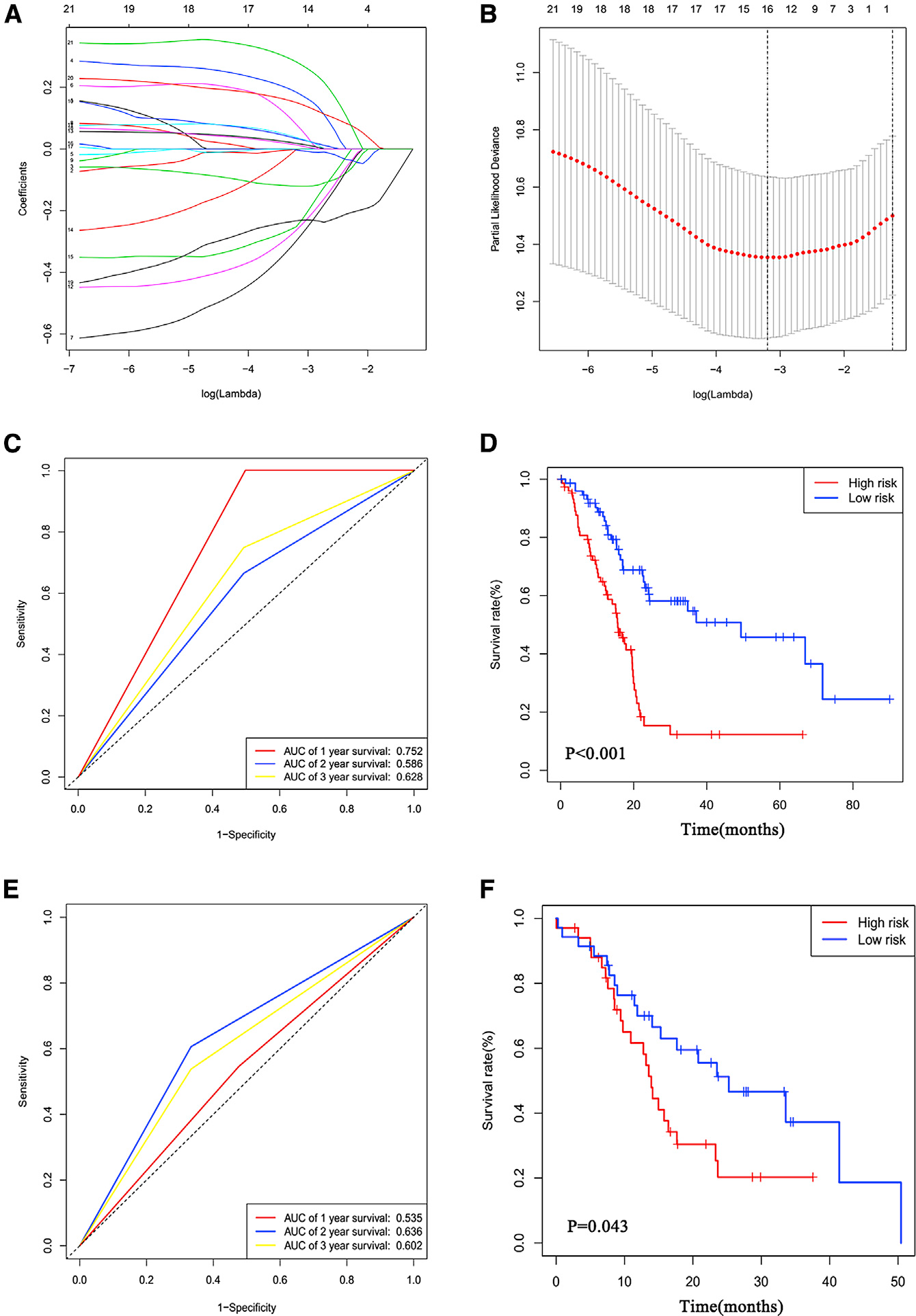
图4 gene signature对患者预后的分析
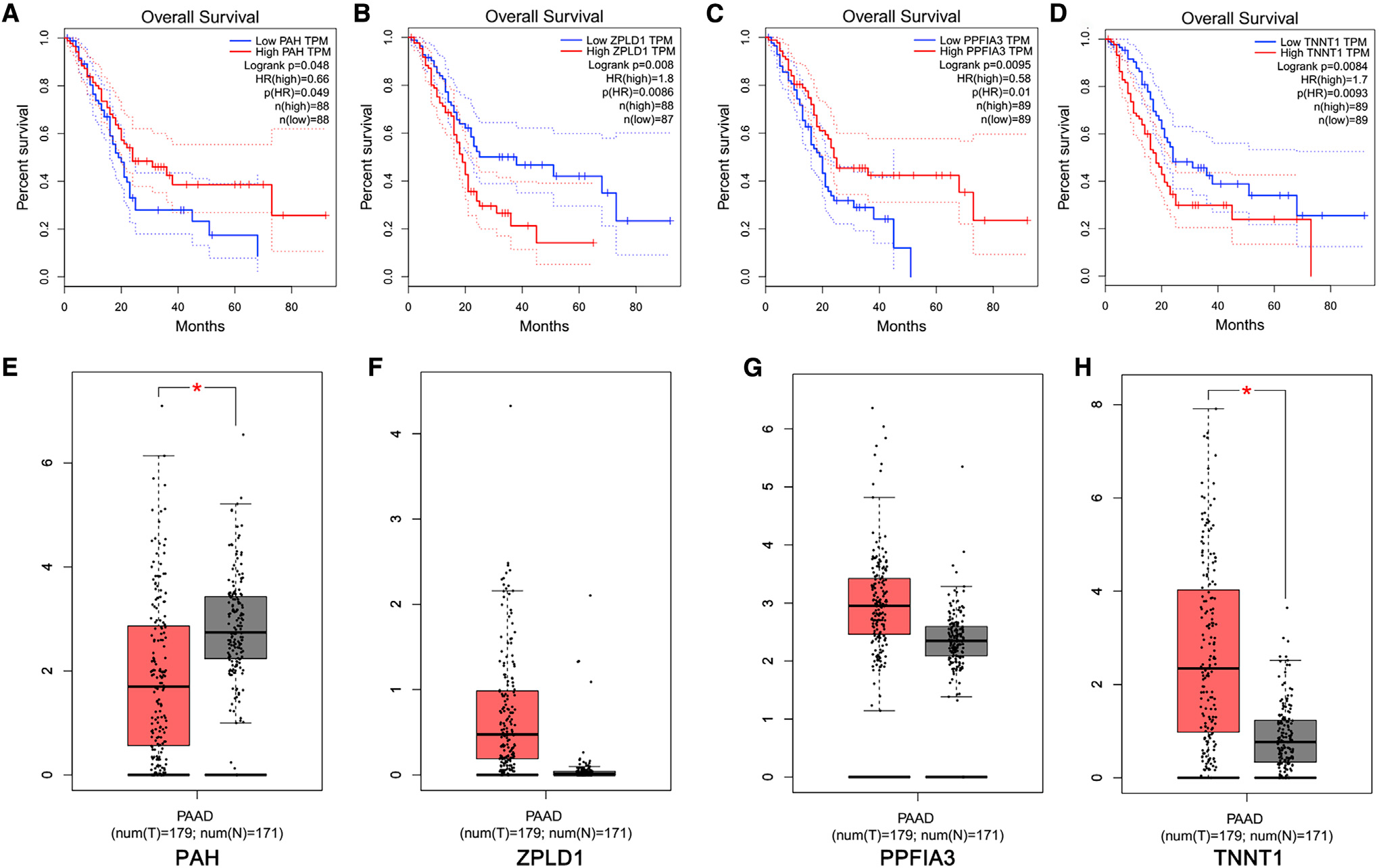
05—
结论
作者构建并验证了在不同m 6 A调调控状态的组之间DEG的mRNA signature,可以预测PAAD患者的预后状态。其次,模型风险基因可以参与PAAD的发育,因此可以作为潜在的治疗靶标。第三,我们没有对正常组和癌症组进行比较,而是使用常规的病理分期系统对亚组患者进行了分类,而是开发了一种新的方法来通过PAAD患者的m6A调节剂改变状态对其进行分类,从而提供了一种新的方法来识别不同亚型患者之间的异质性。

- 发表于 2020-06-17 14:20
- 阅读 ( 8049 )
- 分类:文献解读
你可能感兴趣的文章
相关问题
- geo数据库可以挖掘m6a吗 0 回答
- 用生信人工具批量分析预后,分类方式应该怎么设置? 1 回答
