RefSeq注释下载及加工
如何从UCSC下载refseq参考基因注释文件
看教程不够直观,那就看视频吧! >>点击加载视频
RefSeq注释下载及加工
date: 2017.11.21
author:jinwen
引
虽然是科班生信人,但是由于不咋用功,对于UCSC的使用也懵懵懂懂……
这里是最简单的使用
RefSeq基因下载
这个就不赘述了,直接上图
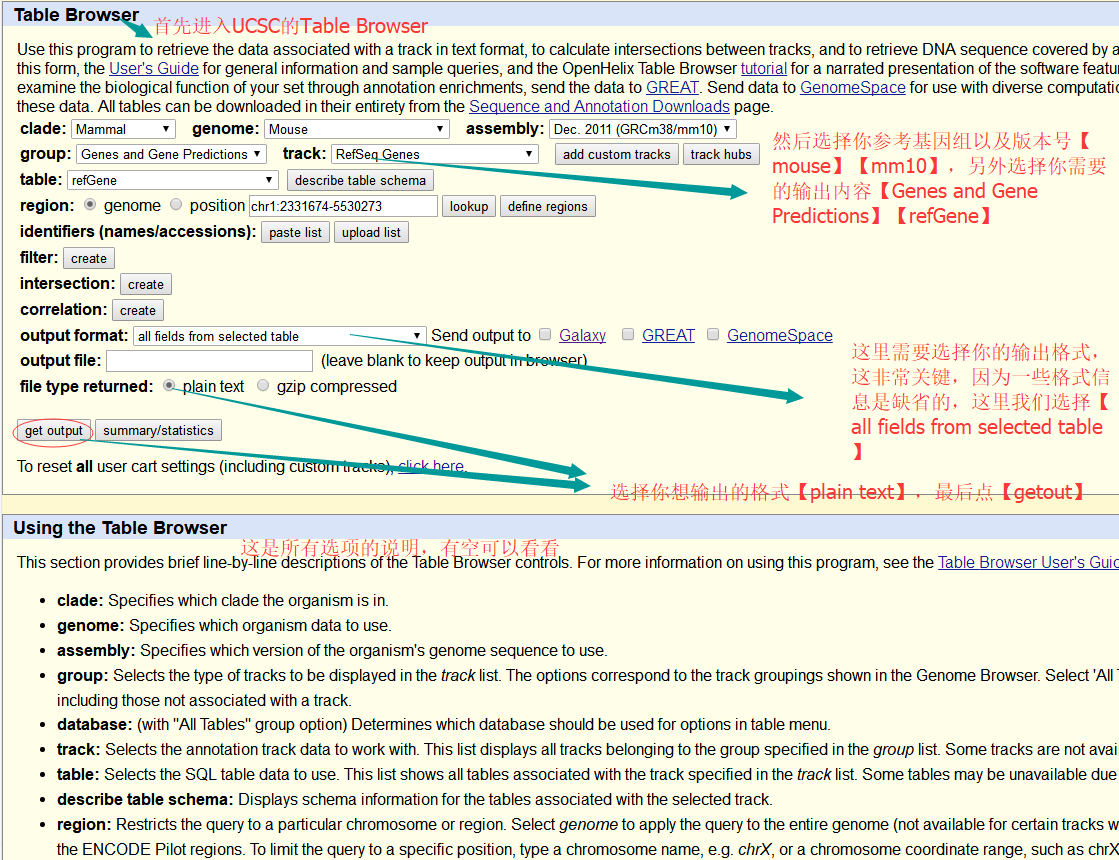
结果太长,看不出啥,看一下表头吧
#bin name chrom strand txStart txEnd cdsStart cdsEnd exonCount exonStarts exonEnds score name2 cdsStartStat cdsEndStat exonFrames

如何获得RefSeq的gtf文件
当然你可以自己编程从上面的文件提取,但似乎还是挺烦,所以UCSC提供了相应的工具genePredToGtf
1. 下载
64位linux系统下载地址:http://hgdownload.soe.ucsc.edu/admin/exe/external.x86_64/
在一大摞工具中选中genePredToGtf
注意:这是一个编译后的可执行文件,没有后缀,如果下载时带有后缀(比如我下载时有.txt)务必去除后缀
2. 安装
刚才已经说了这是一个可执行文件,所以不需要安装,直接使用
./genePredToGtf
或者加入到环境变量中
vim ~/.bashrc
添加
export PATH=$PATH:/你的目录/genePredToGtf
最后
source ~/.bashrc
3. 使用
假如在UCSC下载的文件是 mm10_ref.genePred ,由于该文件有表头#bin ...,所以要先去除,然后遵循使用说明
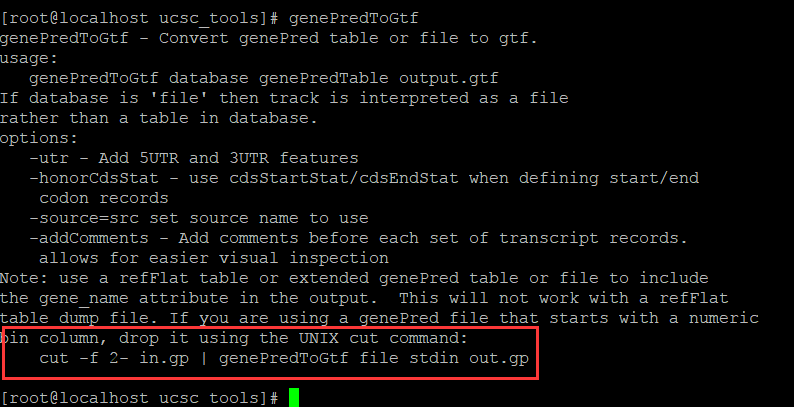
即
grep -v "#" mm10_ref.genePred > mm10_ref.genePred
cut -f 2- mm10_ref.genePred | genePredToGtf file stdin mm10_ref.gtf
好了,大功告成!
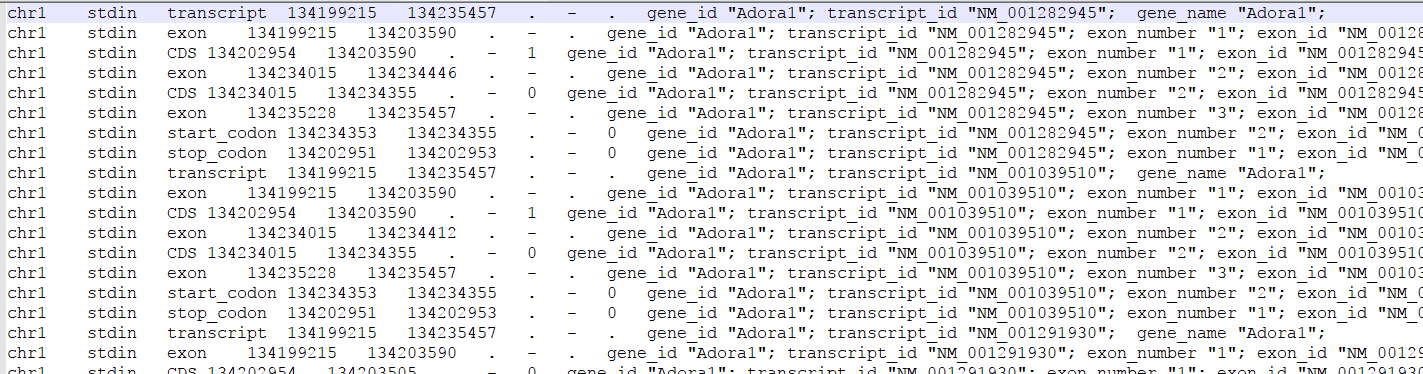
尾
当然,既然有genePredToGtf,当然也有gtfToGenePred。比如你想要把Ensembl的gtf文件转换一下,也是可以的!
- 发表于 2017-11-21 15:47
- 阅读 ( 15262 )
- 分类:转录组学
你可能感兴趣的文章
- 干货|基因注释方法论 9461 浏览
2 条评论
请先 登录 后评论
