ggbiplot-最好看的PCA作图:样品PCA散点+分组椭圆+变量贡献和相关
看教程不够直观,那就看视频吧! >>点击加载视频
写在前面
https://github.com/vqv/ggbiplot/blob/master/README.md
前几天在《宏基因组0》微信讨论群看到了有人发了一个上面链接,点开一看居然是一条命令出帅图,真是太实用了。我立即使用本领域的OTU表上进行了测试,效果很好,现分享给大家,欢迎大家留言补充。
ggbiplot简介
ggbiplot是一款PCA分析结果可视化的R包工具,可以直接采用ggplot2来可视化R中基础函数prcomp() PCA分析的结果,并可以按分组着色 、分组添加不同大小椭圆、主成分与原始变量相关与贡献度向量等。
An implementation of the biplot using ggplot2. The package provides two functions: ggscreeplot() and ggbiplot(). ggbiplot aims to be a drop-in replacement for the built-in R function biplot.princomp() with extended functionality for labeling groups, drawing a correlation circle, and adding Normal probability ellipsoids.
ggbiplot安装和官方示例
R包,建议在Rstudio中使用更方便
# 安装包,安装过请跳过 install.packages("devtools", repo="http://cran.us.r-project.org") library(devtools) install_github("vqv/ggbiplot") # 最简单帅气的例子 data(wine) wine.pca <- prcomp(wine, scale. = TRUE) # 演示样式 ggbiplot(wine.pca, obs.scale = 1, var.scale = 1, groups = wine.class, ellipse = TRUE, circle = TRUE) + scale_color_discrete(name = '') + theme(legend.direction = 'horizontal', legend.position = 'top') # 基本样式 plot(wine.pca$x) # 原始图,大家可以尝试画下,不忍直视
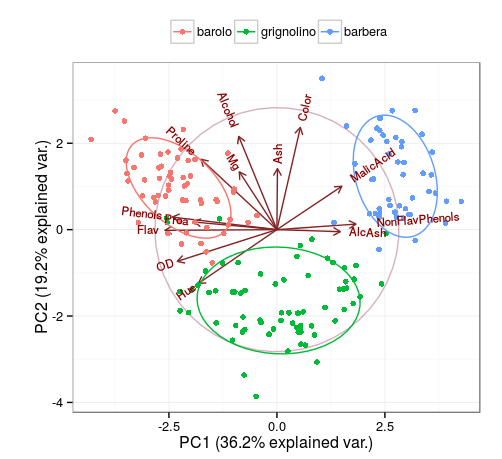
看,是不是一条命令就把prcomp()得到的主成分分析PCA的结果展示的淋漓尽致。是不是瞬间有了高分文章的B格。
主要结果说明:
- 坐标轴PC1/2的数值为总体差异的解释率;
- 图中点代表样品,颜色代表分组,图例在顶部有三组;
- 椭圆代表分组按默认68%的置信区间加的核心区域,便于观察组间是否分开;
- 箭头代表原始变量,其中方向代表原始变量与主成分的相关性,长度代表原始数据对主成分的贡献度。
更详细的PCA原理、推导、图解,请跳转《一文看懂PCA主成分分析》,再点击阅读原文。重点关注——PCA结果解释部分。
OTU表实战
本实战,基于本公众号之前发布文章 《扩增子分析教程-3统计绘图-冲击高分文章》中提供测试数据的OTU表、实验设计和物种注释信息,需要者可前往下载。
PCA分析OTU表和可视化
# 菌群数据实战 # 读入实验设计 design = read.table("design.txt", header=T, row.names= 1, sep="\t") # 读取OTU表 otu_table = read.delim("otu_table.txt", row.names= 1, header=T, sep="\t") # 过滤数据并排序 idx = rownames(design) %in% colnames(otu_table) sub_design = design[idx,] count = otu_table[, rownames(sub_design)] # 基于OTU表PCA分析 otu.pca <- prcomp(t(count), scale. = TRUE) # 绘制PCA图,并按组添加椭圆 ggbiplot(otu.pca, obs.scale = 1, var.scale = 1, groups = sub_design$genotype, ellipse = TRUE,var.axes = F)
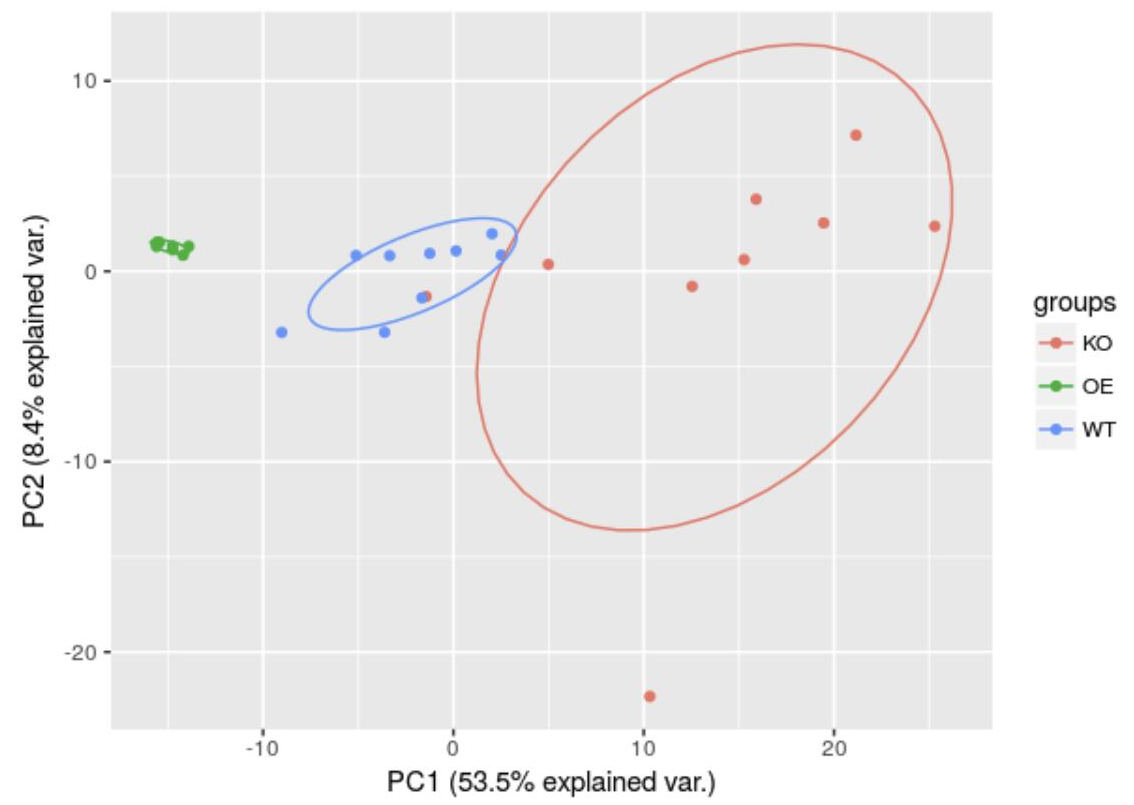
可以看到三个组在第一主轴上明显分开。
展示主要差异菌与主成分的关系
# 显著高丰度菌的影响 # 转换原始数据为百分比 norm = t(t(count)/colSums(count,na=T)) * 100 # normalization to total 100 # 筛选mad值大于0.5的OTU mad.5 = norm[apply(norm,1,mad)>0.5,] # 另一种方法:按mad值排序取前6波动最大的OTUs mad.5 = head(norm[order(apply(norm,1,mad), decreasing=T),],n=6) # 计算PCA和菌与菌轴的相关性 otu.pca <- prcomp(t(mad.5)) ggbiplot(otu.pca, obs.scale = 1, var.scale = 1, groups = sub_design$genotype, ellipse = TRUE,var.axes = T)
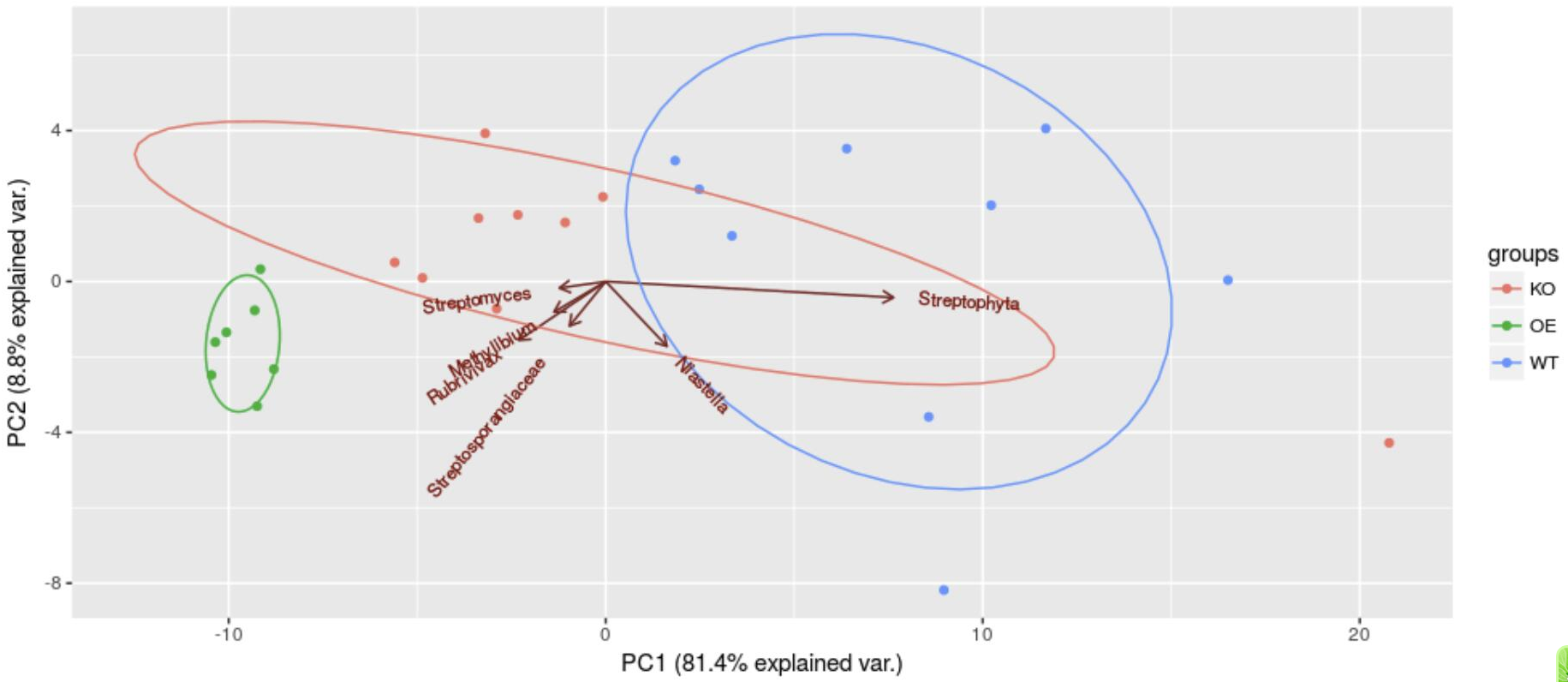
我们仅用中值绝对偏差(mad)最大的6个OTUs进行主成分分析,即可将三组样品明显分开。图中向量长短代表差异贡献,方向为与主成分的相关性。可以看到最长的向量Streptophyta与X轴近平行,表示PC1的差异主要由此菌贡献。其它菌与其方向相反代表OTUs间可能负相关;夹角小于90%的代表两个OTUs有正相关。
ggbiplot官网简介ggbiplot
An implementation of the biplot using ggplot2. The package provides two functions: ggscreeplot() and ggbiplot(). ggbiplot aims to be a drop-in replacement for the built-in R function biplot.princomp() with extended functionality for labeling groups, drawing a correlation circle, and adding Normal probability ellipsoids.
ggbiplot使用和参数
?ggbiplot ggbiplot(pcobj, choices = 1:2, scale = 1, pc.biplot = TRUE, obs.scale = 1 - scale, var.scale = scale, groups = NULL, ellipse = FALSE, ellipse.prob = 0.68, labels = NULL, labels.size = 3, alpha = 1, var.axes = TRUE, circle = FALSE, circle.prob = 0.69, varname.size = 3, varname.adjust = 1.5, varname.abbrev = FALSE, ...) pcobj # prcomp()或princomp()返回结果 choices # 选择轴,默认1:2 scale # covariance biplot (scale = 1), form biplot (scale = 0). When scale = 1, the inner product between the variables approximates the covariance and the distance between the points approximates the Mahalanobis distance. obs.scale # 标准化观测值 var.scale # 标准化变异 pc.biplot # 兼容 biplot.princomp() groups # 组信息,并按组上色 ellipse # 添加组椭圆 ellipse.prob # 置信区间 labels # 向量名称 labels.size # 名称大小 alpha # 点透明度 (0 = TRUEransparent, 1 = opaque) circle # 绘制相关环(only applies when prcomp was called with scale = TRUE and when var.scale = 1) var.axes # 绘制变量线-菌相关 varname.size # 变量名大小 varname.adjust # 标签与箭头距离 >= 1 means farther from the arrow varname.abbrev # 标签是否缩写
- 发表于 2017-12-23 09:27
- 阅读 ( 13990 )
- 分类:编程语言
你可能感兴趣的文章
- 存在cdf文件的芯片数据,但没有R包构建R包并安装cdf 6833 浏览
- 分享一段从KEGG通路中提取基因互作数据的R代码 8428 浏览
- 服务器里删过SSL的后遗症,R不能正常的安装包了,错误:status was 'Peer certificate cannot be authenticated with given CA certificates 7267 浏览
- 医学统计学-卡方检验 9535 浏览
- 生物信息之程序学习 6142 浏览
- ggplot2高效实用指南 (可视化脚本、工具、套路、配色) 16622 浏览
