五分钟搞定一个芯片的重注释,让那些没有genesymbol的数据再次好用
看教程不够直观,那就看视频吧! >>点击加载视频
我们做芯片数据分析的时候经常会遇到有些基因芯片根本没有geneSymbol,或者有些lncRNA芯片根本没有像样的名称,故此对我们后续的分析也造成了不少的麻烦,基于此 我们可以通过序列比对的方式将探针序列重新比对到转录本上,从而拿到最新的基因编号,本文以HuGene-1_0-st探针为例
芯片平台地址:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL6244
这个平台官网提供的注释信息根本没有genesymbol,只有genebank ID,这就很尴尬了,后续的分析很多都很不方便
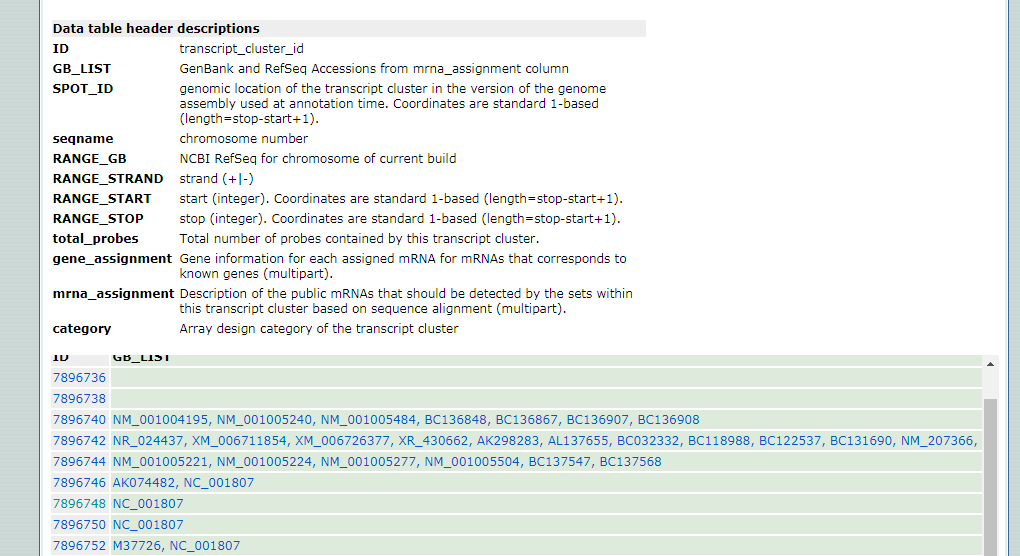 不过不要紧,我们可以使用芯片重注释技术 根据探针序列 将这些探针重新比对到各个转录本中,从而拿到统一的 基因ID
不过不要紧,我们可以使用芯片重注释技术 根据探针序列 将这些探针重新比对到各个转录本中,从而拿到统一的 基因ID
首先根据教程 https://shengxin.ren/article/439 我们下载作为参考转录本文件的序列
然后我们从芯片官网找到探针序列的文件
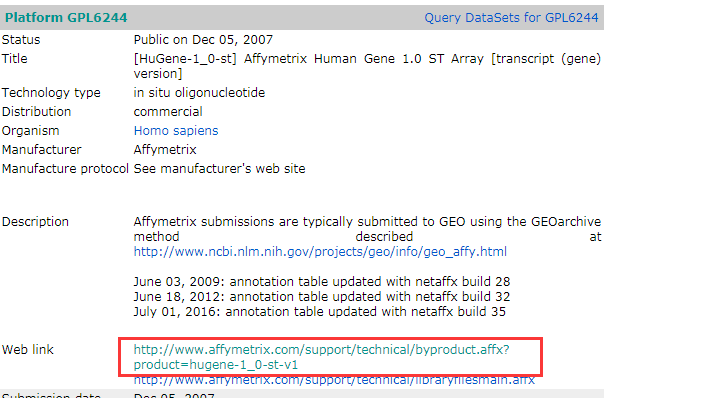
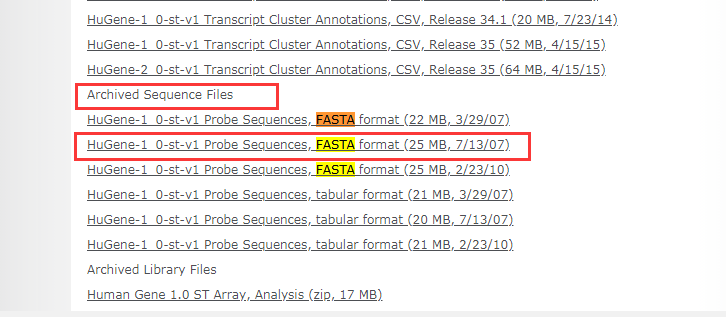
注:存档文件 上面的日期选择 越近越好,还有就是下载完后 打开序列文件 看看探针的ID 对不对的上,有时候探针序列上的ID不一定是 探针ID,此时需要再找找看其他的
导入 SeqMap序列比对工具
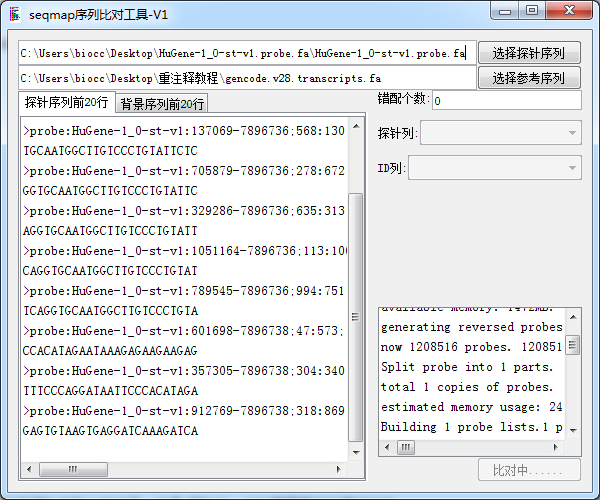 等待比对完成
等待比对完成
注意我们的探针ID
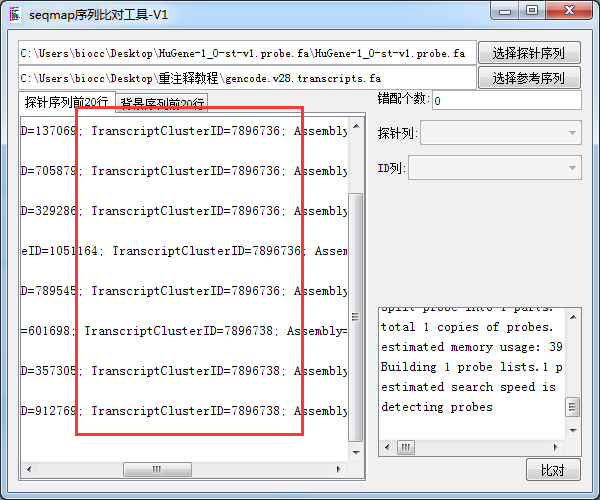
然后将结果导入到 基因注释工具
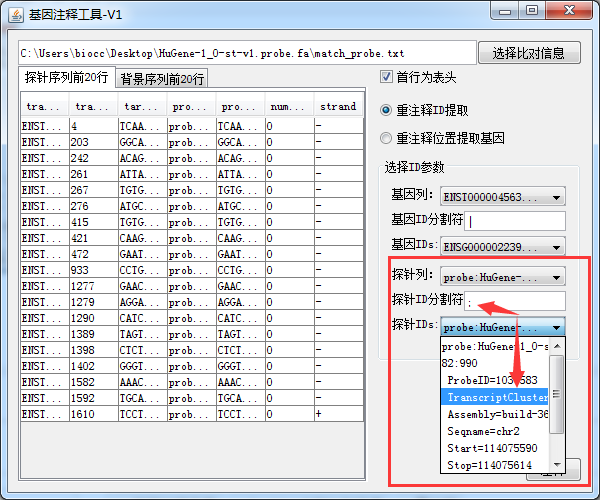 设置正确的分割条件,将最终的ID给提取出来,稍作等待之后,最终得到match_clear文件
设置正确的分割条件,将最终的ID给提取出来,稍作等待之后,最终得到match_clear文件
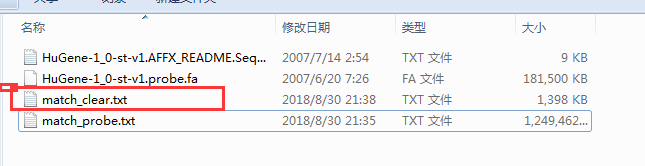 用Excel打开,我们可以看到文件分三列,第一列为探针,但是都多了 TranscriptClusterID=,我们把他替换掉,最终得到了探针ID对应基因ID的文件
用Excel打开,我们可以看到文件分三列,第一列为探针,但是都多了 TranscriptClusterID=,我们把他替换掉,最终得到了探针ID对应基因ID的文件
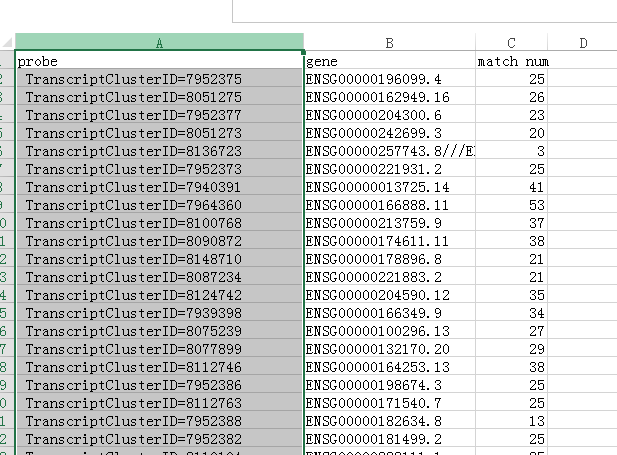 我们可根据这个文件 作为背景文件,使用ID转换器从探针表达谱中转换得到ENSG表达谱,再次转换 便可得到genesymbol表达谱
我们可根据这个文件 作为背景文件,使用ID转换器从探针表达谱中转换得到ENSG表达谱,再次转换 便可得到genesymbol表达谱
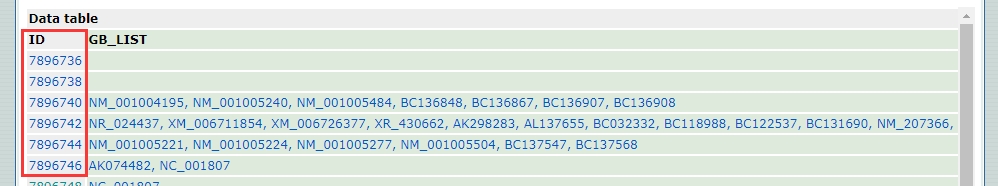
比如探针ID:7896746 原来的注释如下:
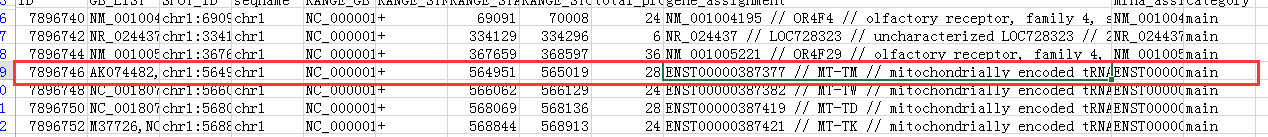
新注释的信息如下:

从数据库查阅到的如下:
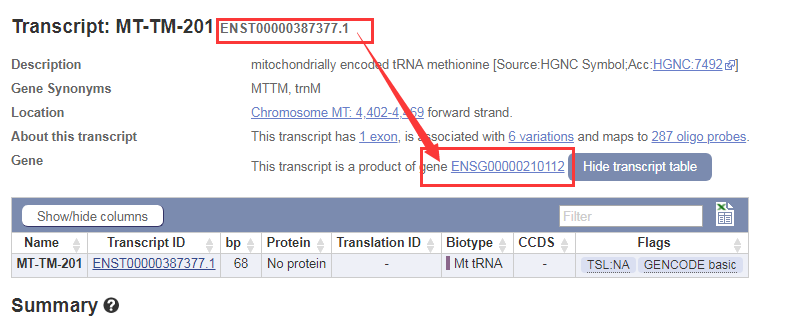
- 发表于 2018-08-30 21:44
- 阅读 ( 25667 )
你可能感兴趣的文章
- 根据芯片平台提供的基因组位置来提取基因 13763 浏览
- 一步一步教你做芯片重注释 30409 浏览
- 做重注释的简单思路记录一下 7926 浏览
