一步一步教你做芯片重注释
看教程不够直观,那就看视频吧! >>点击加载视频
这里以GPL570为例
首先我们需要下载GPL570的探针序列:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL570
找到官网链接:
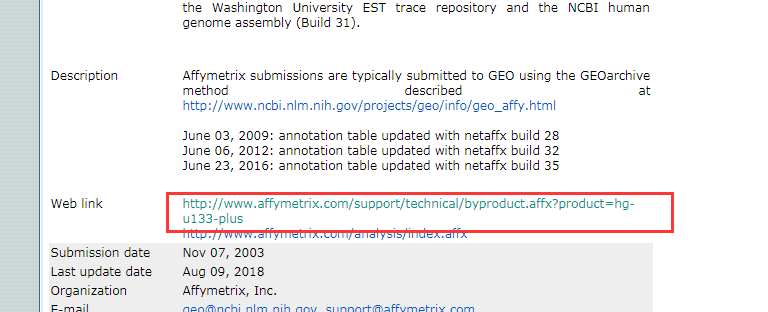 点击进入官网,搜索探针序列:fasta
点击进入官网,搜索探针序列:fasta
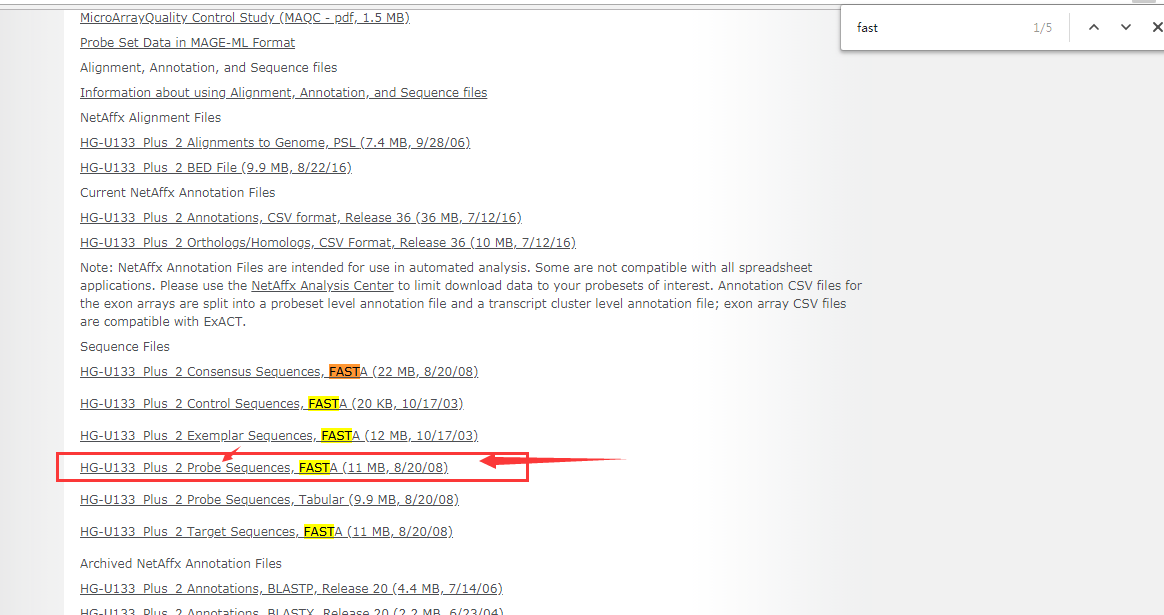 点击下载,即可得到我们这个平台的探针序列了
点击下载,即可得到我们这个平台的探针序列了
进一步的我们需要准备最新的参考基因组序列文件
我们可以从gencode上下载最新版的基因组参考序列:https://www.gencodegenes.org/releases/28.html
方案一:下载所有转录组序列作为参考序列
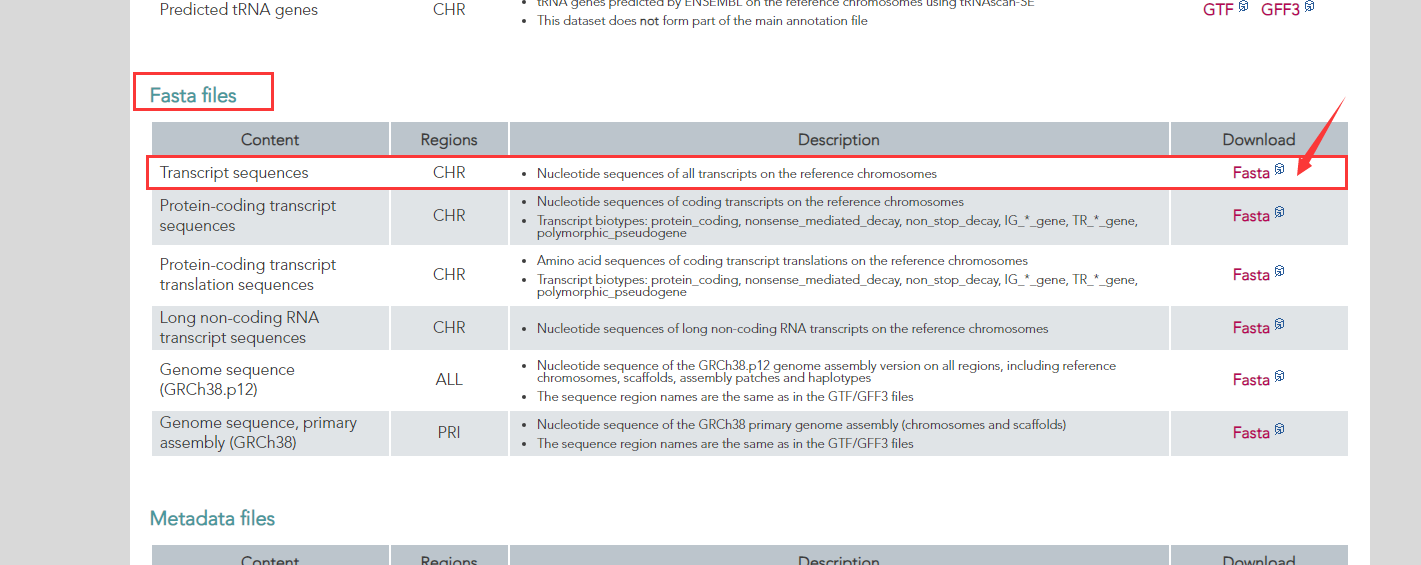
我们下载所有转录本的序列文件作为参考序列,进一步的使用简易seqmap软件进行比对,将探针序列比对到所有转录本上
那么我们已经准备好序列文件如:
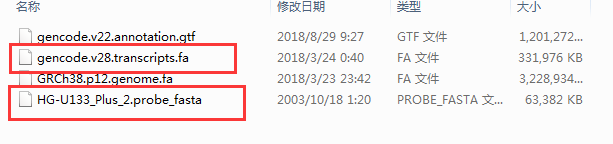 下面我们打开 SeqMap序列比对工具 将数据导入软件如:
下面我们打开 SeqMap序列比对工具 将数据导入软件如:
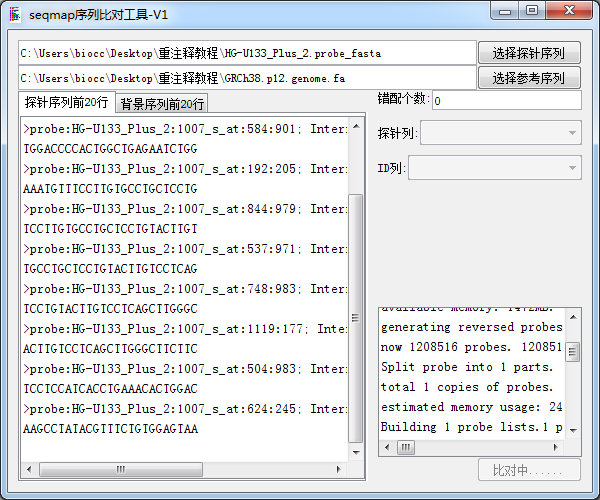
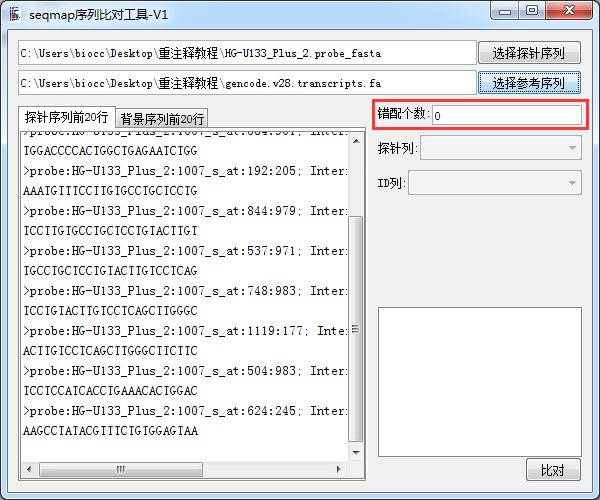 我们选择 错配为0,因为咱们这里的探针序列才25bp,如果比较长的话 可以适当增加错配的数目 这样可以匹配到更多
我们选择 错配为0,因为咱们这里的探针序列才25bp,如果比较长的话 可以适当增加错配的数目 这样可以匹配到更多
点击比对,等到比对完成。
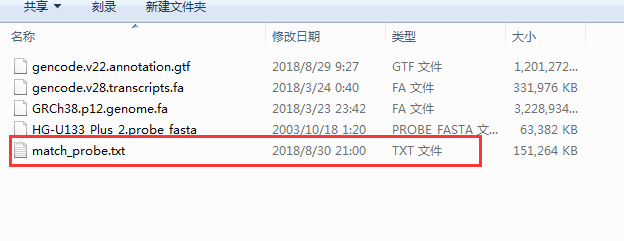
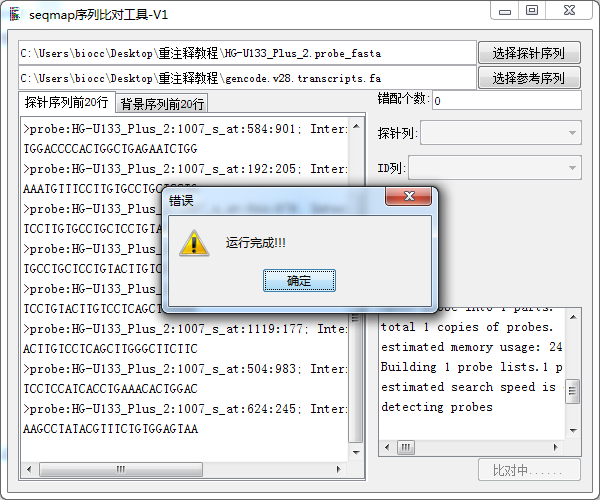 得到比对后的结果文件:
得到比对后的结果文件:
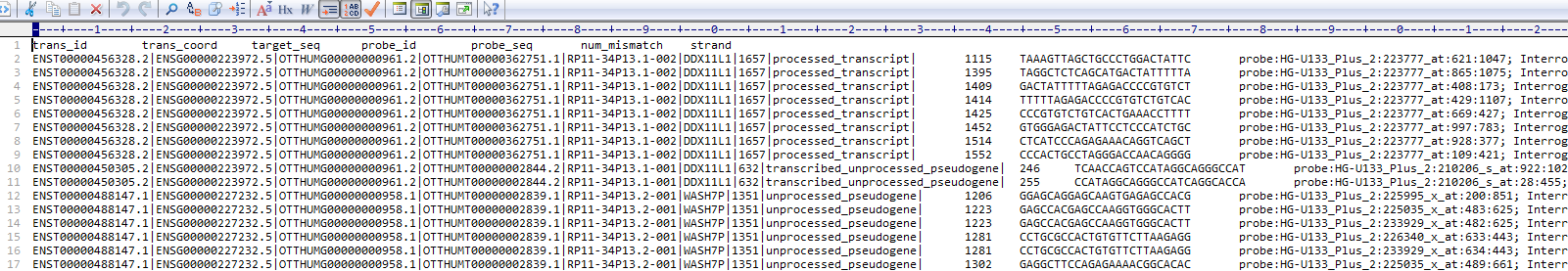 进一步的我们需要从比对结果中提取出基因和探针的关系,我们使用 基因注释工具
进一步的我们需要从比对结果中提取出基因和探针的关系,我们使用 基因注释工具
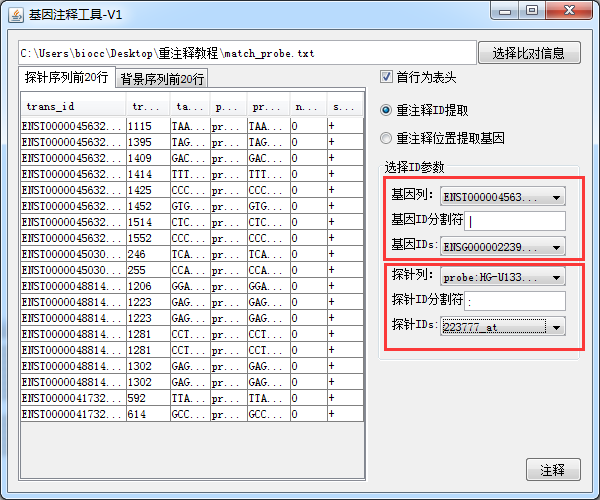
如图,我们将注释结果导入到基因注释工具中,从红色框处 按照一定的规则从里面提取出 探针ID和基因ID,最后 点击 注释 按钮,导出注释结果
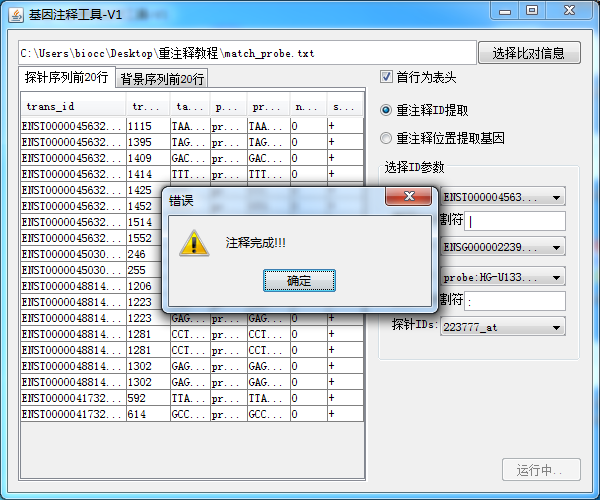
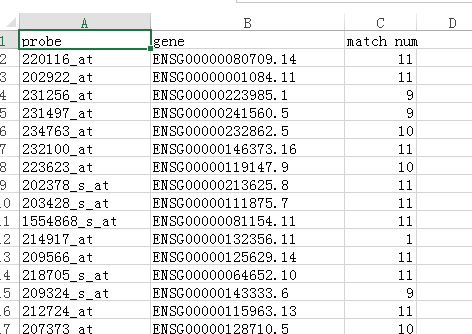 我们可以看到这样就可以将探针对应到ENSG ID上了,这和TCGA使用的ID 一致,我们可以使用ID转换器从中提取出lncRNA和编码基因的ID。
我们可以看到这样就可以将探针对应到ENSG ID上了,这和TCGA使用的ID 一致,我们可以使用ID转换器从中提取出lncRNA和编码基因的ID。
方案二:
下载基因组序列文件作为参考序列,使用简易seqmap软件进行比对,将探针序列比对到基因组

准备序列文件如下:
 将数据导入软件进行比对:
将数据导入软件进行比对:
 等待比对完成,再使用基因注释工具对比对结果进行注释,由于我们使用的是全基因组比对,所以探针可能会比对到的区域更多,我们需要一个文件来指导提取探针比对到基因组上的位置对应的基因信息,所以我们需要下载gtf文件,我们还是从gencode上下载gtf文件
等待比对完成,再使用基因注释工具对比对结果进行注释,由于我们使用的是全基因组比对,所以探针可能会比对到的区域更多,我们需要一个文件来指导提取探针比对到基因组上的位置对应的基因信息,所以我们需要下载gtf文件,我们还是从gencode上下载gtf文件

下载完成后倒入 基因注释工具中,进行下一步的基因注释
如图:
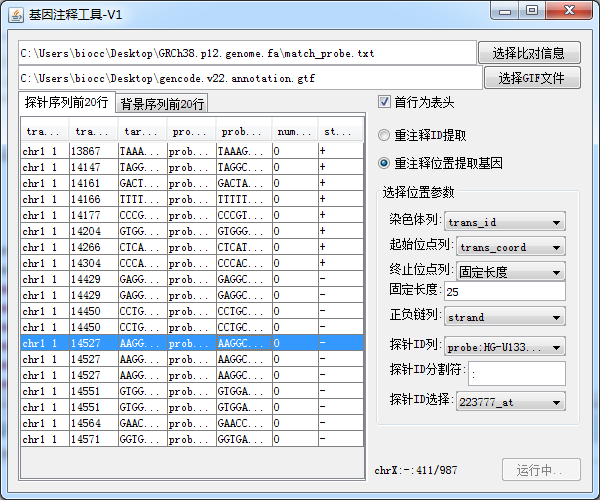
最后依然也可以得到相应的探针注释到基因的结果
- 发表于 2018-08-28 15:51
- 阅读 ( 30344 )
- 分类:软件工具
你可能感兴趣的文章
- 根据芯片平台提供的基因组位置来提取基因 13745 浏览
- 五分钟搞定一个芯片的重注释,让那些没有genesymbol的数据再次好用 25633 浏览
- 干货:如何将别人的芯片数据唯我所用,发自己的文章 8824 浏览
- 做重注释的简单思路记录一下 7912 浏览
