做重注释的简单思路记录一下
看教程不够直观,那就看视频吧! >>点击加载视频
做生物信息一项必备的技能是寻找可用的数据,很多时候手头的数据不够,有很多的公共数据库数据可以使用,如何充分利用这些公共数据则显得尤为重要,GEO数据库中有大量的芯片数据,很多常见的芯片平台的数据都有过比较全面的注释,但是一些比较偏的或者定制化的芯片往往注释信息不够,以至于很多人不知道怎么办?当然lncRNA的数据更是注释信息很少,在TCGA中有一万多的lncRNA,而GEO上很多lncRNA的数据就比较少,并且注释的也少,最坑爹的一个问题因为lncRNA的研究没有gene的研究那么成熟大部分ID也不一样,,这让TCGA和GEO的数据整合分析也变得尤为的困难,最近刚好有个朋友求助GSE36685这套数据如何变成看得懂的表达谱矩阵。
我们一起来简单的看一下这套数据,共有51个样本。GPL15368平台的数据,这是一个定制化的芯片,完全没有什么基因组注释。
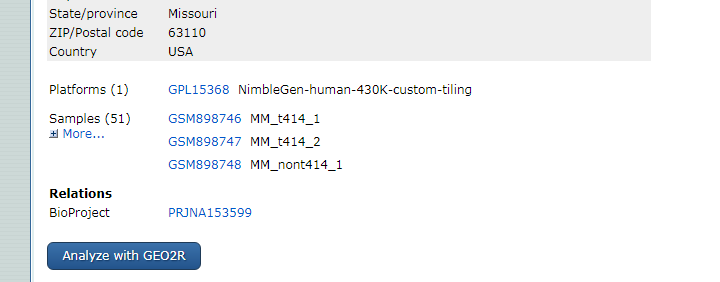
想要得到类似GeneSymbol的表达矩阵就需要费一番功夫了
我们打开这个平台的详细信息,从中可以看出只有位置信息(如图中红框),但是也没有找到是哪个基因组版本的位置(不同基因组版本的基因/lncRNA位置是不一样的)
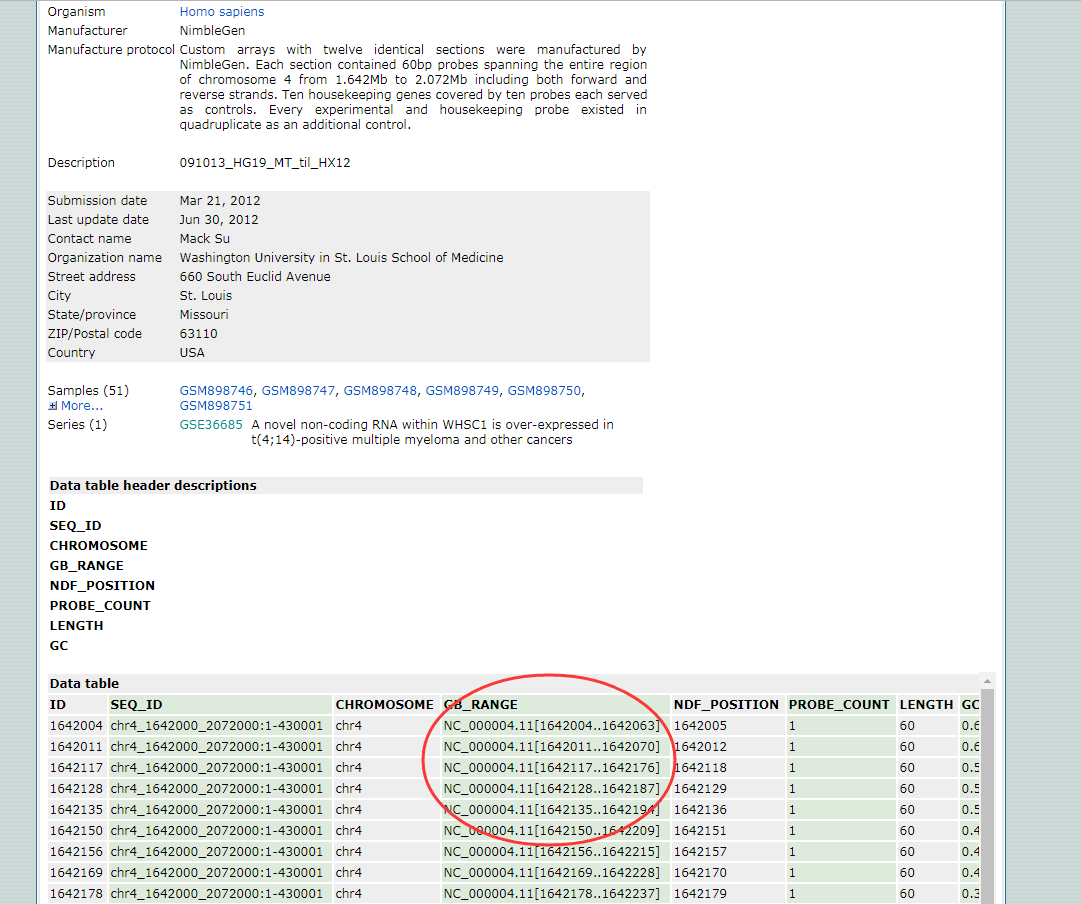
所以我们找这些探针的序列,把序列比对到基因组上,然后再得到比对到基因组上的探针的位置,再根据探针的位置找相关的lncRNA,再往下拉看到了8.2Mb的那个文件,猜测是探针序列文件,先下载下来。
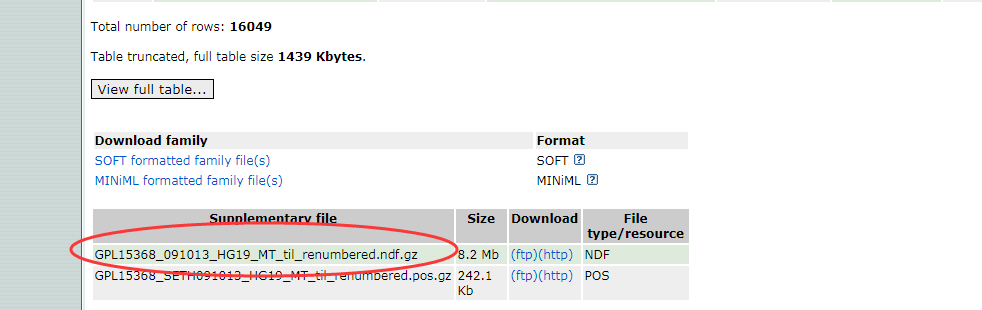
解压打开后长这个样子
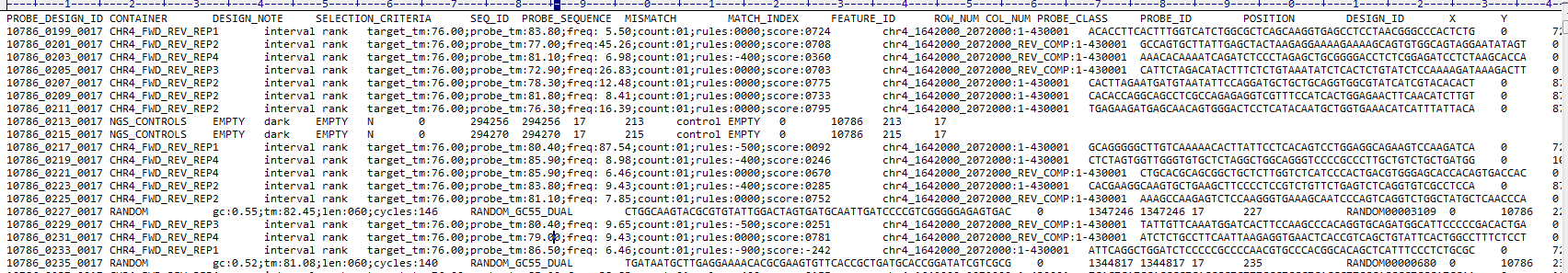 从里面大体上可以看出,有那么一列序列,我们可以使用Excel打开,这样看起来就整齐了
从里面大体上可以看出,有那么一列序列,我们可以使用Excel打开,这样看起来就整齐了
把这些序列使用在线工具Blat
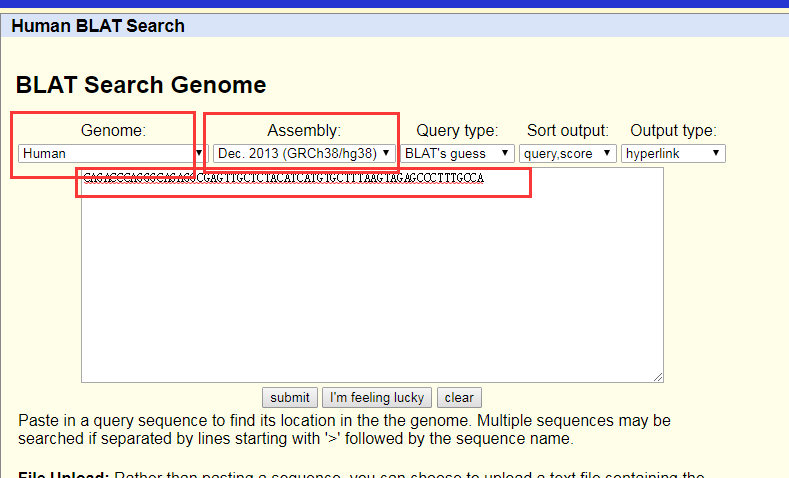 注意红色框选择 正确的基因组版本,然后把序列帖进去就好了
注意红色框选择 正确的基因组版本,然后把序列帖进去就好了
点击运行得到结果如下
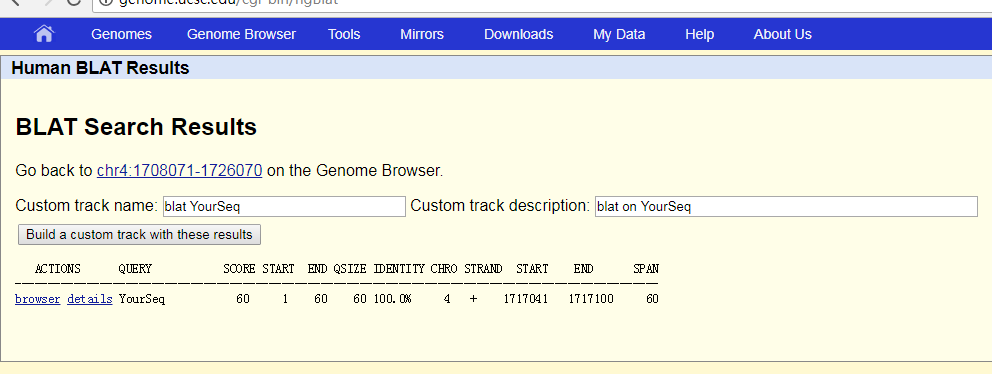 这里就展示了比对结果,这个探针比对到了基因组的哪个位置上
这里就展示了比对结果,这个探针比对到了基因组的哪个位置上
进一步的我们可以点击 browser 看看这个探针在哪个基因上如:
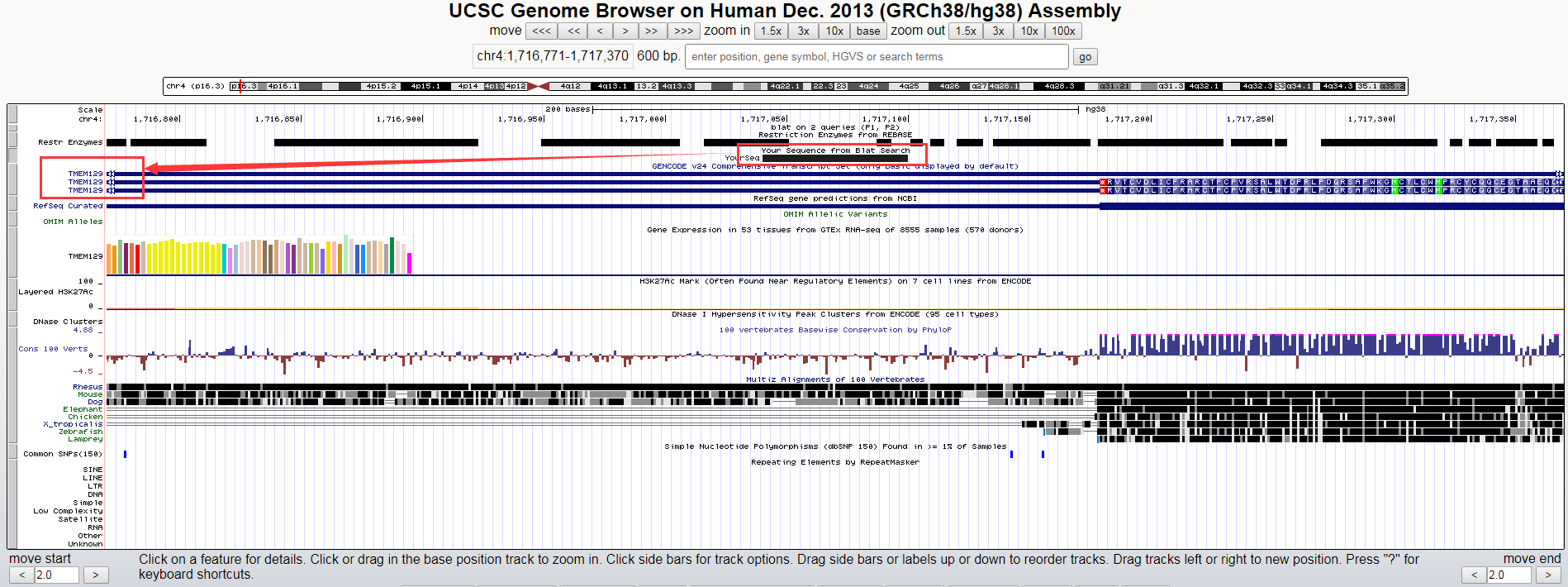
从这个可以看出来这个探针其实是在TMEM129这个基因上的,也就是说这个探针的表达水平代表了这个基因的转录水平,同理其他的探针也可以依次做。
最后得到每一个探针在基因组上的位置,再根据位置确定是哪个基因或者哪个lncRNA的,TCGA使用的是G38版本,比对的时候选择这个版本的基因组即可。
这便是重注释的过程。
原理知道了,剩下的就是写程序批量去做了,如此,大部分的基因芯片都可以统一的做,包括常见的基因芯片。
- 发表于 2018-05-21 17:03
- 阅读 ( 7913 )
- 分类:方案研究
你可能感兴趣的文章
- 根据芯片平台提供的基因组位置来提取基因 13745 浏览
- 五分钟搞定一个芯片的重注释,让那些没有genesymbol的数据再次好用 25634 浏览
- 一步一步教你做芯片重注释 30345 浏览
