文章解读——基于20个基因的预后模型预测肺腺癌的生存
看教程不够直观,那就看视频吧! >>点击加载视频

基于20个基因的预后模型预测肺腺癌的生存
Twenty-gene-based prognostic model predicts lung adenocarcinoma survival
肺腺癌(LAC)占非小细胞肺癌的一半以上,它具有高发病率和死亡率。治疗的进展并未对其预后有改善。因此,迫切需要为其早期诊断和治疗开发新型生物标志物。
材料与方法:
这项研究通过综合分析大规模基因表达谱来识别肺腺癌生存相关的基因。肺腺癌基因表达数据从TCGA数据库获得。首先TCGA数据库中的原始read数通过分位数标准化方法进行标准化,对数转换为正态分布。本文使用DESeq2包筛选了LAC与邻近正常肺组织之间的差异表达基因(p-value<0.01,log2-based fold change>1)。GEO数据库中的基因表达数据,使用GEO2R网络工具进行差异表达分析(p-value<0.01,log2-based fold change>1)。
然后本文通过两步方法进行筛选LAC生存相关基因。首先,进行单变量Cox回归分析以获得与LAC生存显著相关的基因(SurGenes)。进而,本文使用基于LASSO Cox惩罚回归模型进行确定的独立筛查(SIS)来识别更重要的变量,并构建用于生存时间预测的基于多基因的预后特征。两步筛选方法更鲁棒,通过SIS R包实现。另外两个来自TCGA和GEO的肺癌数据集用于验证预后特征。
统计分析:
本文通过对每个样本基于多基因的预后特征计算了风险得分(RS),通过RS的中值将样本分为两组。KM生存曲线被绘制,并通过对数秩检验比较两组之间的差异。基于最近邻方法对预测的3年生存期绘制ROC曲线,并计算曲线下面积(AUC)。
结果:
候选基因
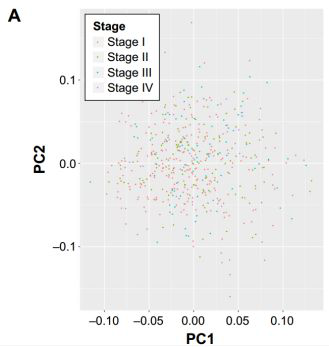
首先,本文通过前两个主成分对训练集样本的分期进行聚类分析。结果显示,1到4期的LAC样本彼此可以分开。因此,识别其他诊断生物标志物对于个性化治疗的帮助很重要。如下图所示:
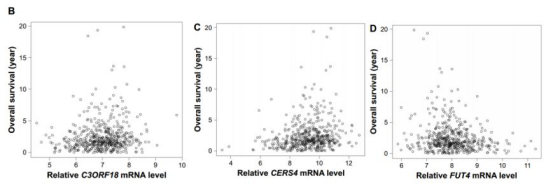
然后,2011个差异表达基因被识别,单变量COX回归分析识别出1425个SurGenes。下面三个图显示出C3ORF18,CERS4和FUT4三个基因的表达值与生存时间的Spearman相关性。P值都是显著的。
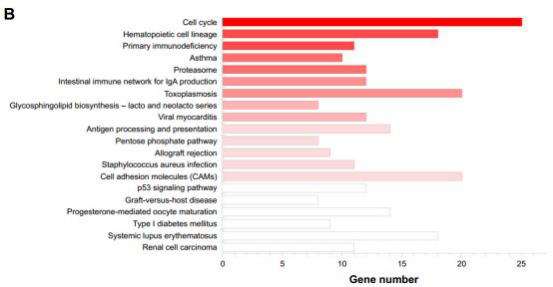
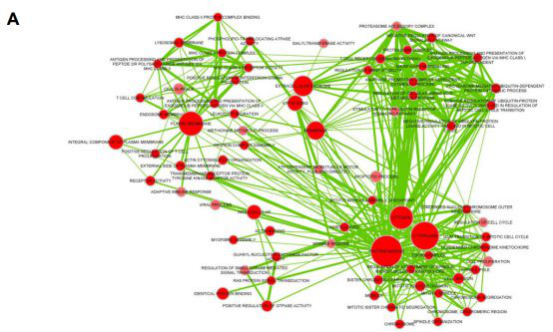
富集分析显示出1425个SurvGenes主要富集在细胞周期,免疫反应等生物学过程和通路中。如下图所示:
SIS分析用来识别鲁棒的基因组合。最终,共计20个基因被获得,这与LAC患者的总生存期(OS)显著相关。这20个基因显示在下表中:
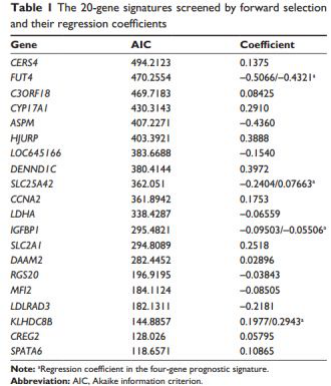
GEO数据集共识别出1282个差异表达基因,这些差异表达基因与20个候选基因取交集,最终得到4个overlap基因:FUT4, SLC25A42, IGFBP1, KLHDC8B,它们既是预后特征,又是GEO数据集中LAC的差异表达基因。
LAC患者的预后特征
本文通过层次聚类,根据20个候选基因的表达值及4个overlap基因的表达值将检验集中的患者分成两个亚组。如下图所示:
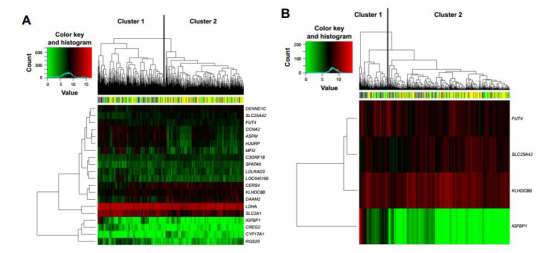
结果显示:
20个候选基因和4个overlap基因可显著的区分高OS的LAC患者与低OS患者。如下图所示:
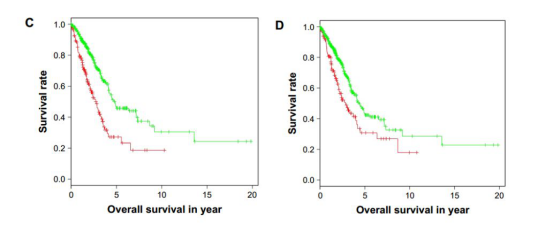
本文还根据患者的20个基因的预后评分和4个overlap基因的预后评分,将患者分为两组,分别根据患者的中位数进行分组,并通过对数秩检验检验亚组间OS差异的显着性。结果显示:20个基因的预后评分和4个overlap基因的预后评分都可以鲁棒的预测LAC的生存。如下图所示:
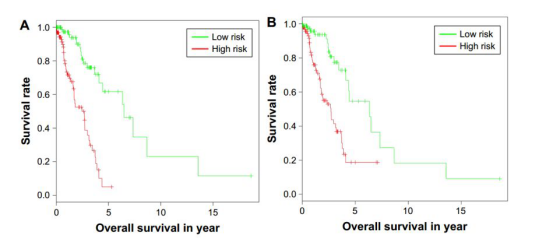
ROC曲线结果显示出它们可以有效预测3年OS(AUC20=0.615,AUC4=0.5731)。如下图所示:
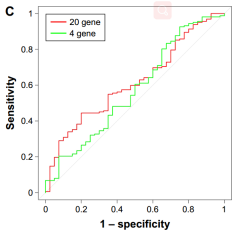
预后特征可以成功地将高生存时间的LAC与低生存时间的样本分开,并具有强大的生存预测性能。
讨论:
混合多基因表达谱的预后特征对LAC有着好的生存时间预测,它对于合适的治疗方法的选择有帮助。
做生信跟踢足球一样,
常规的分析就是这些,
如何利用这些常规分析,
实现不同的想法,这才是最重要的。
大家如果有相关的生信分析需求,可以QQ联系1615556104
- 发表于 2018-09-18 15:48
- 阅读 ( 12411 )
- 分类:文献解读
你可能感兴趣的文章
- 在GEO数据库如何找带有预后信息的数据?一分钟搞定,适用于找任何的样本特征数据 21042 浏览
- 当在文献中看到一个GEO数据集却在GEO中没有权限下载的时候,应该想到ArrayExpress 5764 浏览
- 如果导师丢给你一个基因让你去研究,不妨试一下这两个五分的套路 26095 浏览
- 根据芯片平台提供的基因组位置来提取基因 13754 浏览
- 简易矩阵操作工具使用教程 13619 浏览
