如果导师丢给你一个基因让你去研究,不妨试一下这两个五分的套路
看教程不够直观,那就看视频吧! >>点击加载视频
很多研究都是集中在大样本的基因集中去筛选目标基因,这也是常规做数据挖掘的思路,但是做基础实验的常常关注某个基因,那么一个基因怎么做公共数据挖掘?
这里有两个秘籍分享给大家
1、横向多组学关联篇:
High Expression of CPT1A Predicts Adverse Outcomes: A Potential Therapeutic Target for Acute Myeloid Leukemia
 这是一篇发表在六分多杂志上关于研究CPT1A 在急性髓性白血病中表达关系的文章
这是一篇发表在六分多杂志上关于研究CPT1A 在急性髓性白血病中表达关系的文章
主要步骤如下:
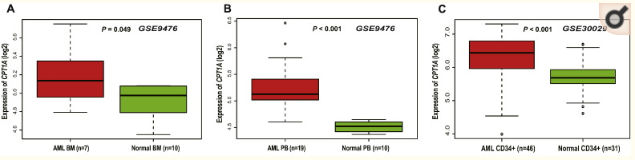
一、本文首先观察了CPT1A基因在AML中的表达情况,从GEO数据库中找了几套不同类型的白血病样本和健康样本的芯片数据,从中提取出这个基因在各个样本中的表达,然后分析正常与疾病组的差异,发现在CPT1A在白血病中显著高表达。
二、既然CPT1A在白血病中表达上调,那么他是否与白血病患者的临床特征有关系呢?于是我们又从GEO上找了一套带有样本临床信息的表达谱数据,然后提取CPT1A的表达数据将样本按照CPT1A表达中位数进行高低表达分组,首先分析了一下CPT1A高表达组中年龄和低表达组年龄上是否有差异,发现存在明显的差异,高表达组患者明显更年轻,然后再看看其他临床信息,发现没啥可分析的了,于是灵机一动,可以看看高低两组样本中一些已经报道的白血病相关的基因标志物他们的表达水平是不是存在差异呢,通过文献检索找了一些已报道的白血病相关的基因标志物分别分析了他们在高低表达组中的差异情况,发现有好多个是存在表达差异的,而这些基因更多是预后标志物。
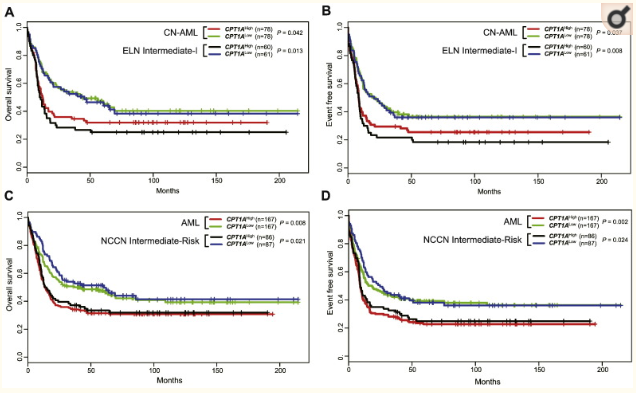
三、于是我们猜想CPT1A是否与预后有关呢?我们又从GEO上找了一套带有预后信息的数据,然后提取CPT1A的表达数据将样本按照CPT1A表达中位数进行高低表达分组,观察高表达和低表达组样本的预后差异,发现高表达组的预后更差。
下面该做啥,感觉只能做到这里了,但是发文章还欠点啥吧
四、想研究一下CPT1A的功能,但是CPT1A一个基因查一下文献就知道功能了,没操作空间,咋整?以前做lncRNA的时候,我们经常通过分析lncRNA共表达的基因来做lncRNA功能分析,难道在这里得不通吗,我们做了CPT1A全基因组表达关联分析(简单来说就是分别计算了CPT1A与其他所有基因的表达相关性),哦,不对,作者是根据CPT1A高低表达组进行了差异分析,然后找到了差异表达的基因,进一步的利用这些基因做功能富集分析,找到了显著富集的通路,观察这些通路与白血病的关系,同时在差异基因中观察这些差异基因有没有被报道过与白血病相关,有相关的则列出来讨论。
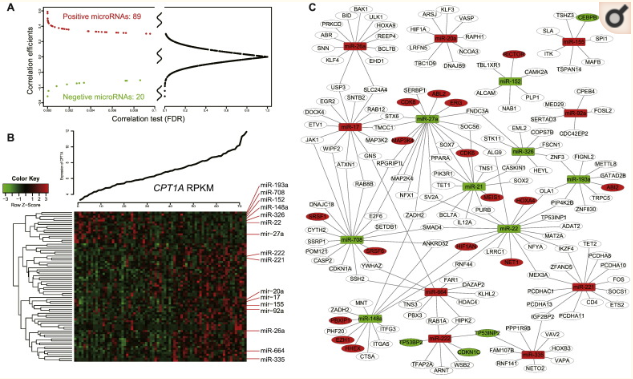
 五、基因表达谱分析完了,接下来我们可以看看与非编码RNA的关系,于是我们分析了高低表达组中miRNA的表达差异,并分析差异的miRNA与靶基因的关系,构建miRNA-mRNA网络,一顿分析差异的miRNA的功能,比如正相关microRNA包括miR-222,miR-221,miR-20a,miR-17,miR-155,miR-26a,miR-335等等。已发现所有这些microRNA在先前的研究中具有重要的肿瘤促进价值。miR-222 / 221可以增强黑素瘤细胞的增殖和分化阻断.......
五、基因表达谱分析完了,接下来我们可以看看与非编码RNA的关系,于是我们分析了高低表达组中miRNA的表达差异,并分析差异的miRNA与靶基因的关系,构建miRNA-mRNA网络,一顿分析差异的miRNA的功能,比如正相关microRNA包括miR-222,miR-221,miR-20a,miR-17,miR-155,miR-26a,miR-335等等。已发现所有这些microRNA在先前的研究中具有重要的肿瘤促进价值。miR-222 / 221可以增强黑素瘤细胞的增殖和分化阻断.......
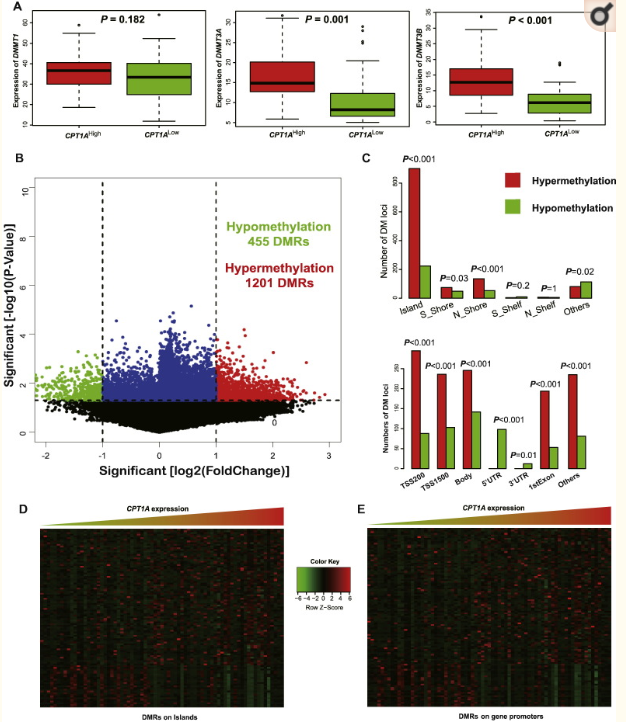
 六、接下来我们还可以分析CPT1A高低表达与甲基化的关系,与甲基化的关系主要有两方面,甲基转移酶的表达和全基因组甲基化水平;首先我们分析CPT1A高低表达组中甲基转移酶的表达差异,发现他们在高CPT1A组中表达高;进一步的我们分析CPT1A高低表达组中全基因组甲基化的差异(简单来说就是CPT1A高低表达组中的甲基化差异分析),得到差异的甲基化位点,根据这些位点的基因组位置,统计一下他们在启动子区域是怎样的一种分布形式以及在CpG岛上市怎样的一种分布形式(甲基化分析常规套路)。
六、接下来我们还可以分析CPT1A高低表达与甲基化的关系,与甲基化的关系主要有两方面,甲基转移酶的表达和全基因组甲基化水平;首先我们分析CPT1A高低表达组中甲基转移酶的表达差异,发现他们在高CPT1A组中表达高;进一步的我们分析CPT1A高低表达组中全基因组甲基化的差异(简单来说就是CPT1A高低表达组中的甲基化差异分析),得到差异的甲基化位点,根据这些位点的基因组位置,统计一下他们在启动子区域是怎样的一种分布形式以及在CpG岛上市怎样的一种分布形式(甲基化分析常规套路)。
以上六个方面便是此文的所有结果,看完是不是有新的思路和想法呢
2、功能联系分析篇
LAYN Is a Prognostic Biomarker and Correlated With Immune Infiltrates in Gastric and Colon Cancers
 这是一篇发表在五分多的杂志上关于LAYN是作为一种预后生物标志物与胃癌和结肠癌中的免疫浸润相关的分析的文章
这是一篇发表在五分多的杂志上关于LAYN是作为一种预后生物标志物与胃癌和结肠癌中的免疫浸润相关的分析的文章
主要步骤如下:
一、首先利用Oncomine数据库分析LAYN基因在各个肿瘤中的表达差异情况,我们发现在乳腺癌,结肠直肠癌,胃癌,肾癌,胰腺癌和淋巴瘤肿瘤中的LAYN表达更高,在膀胱癌,乳腺癌,结肠直肠癌,头颈癌,肺癌,卵巢癌和前列腺癌中观察到较低的表达,进一步的利用TCGA数据库的RNAseq数据分析验证了一遍,也观察到类似的现象。
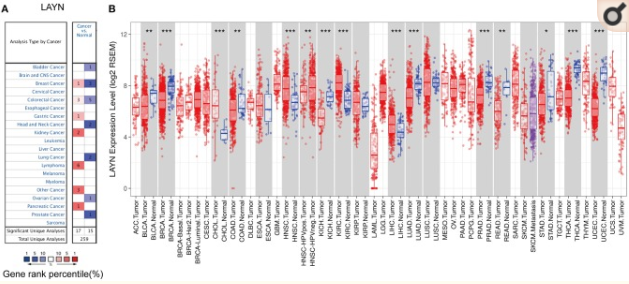
二、我们再看一下LAYN基因与预后的关系,我们利用PrognoScan、GEPIA等在线工具分别分析了LAYN在各个肿瘤中的预后差异,我们发现LAYN表达显着影响5种癌症的预后,包括结肠直肠癌,乳腺癌,眼癌、卵巢癌和胃癌
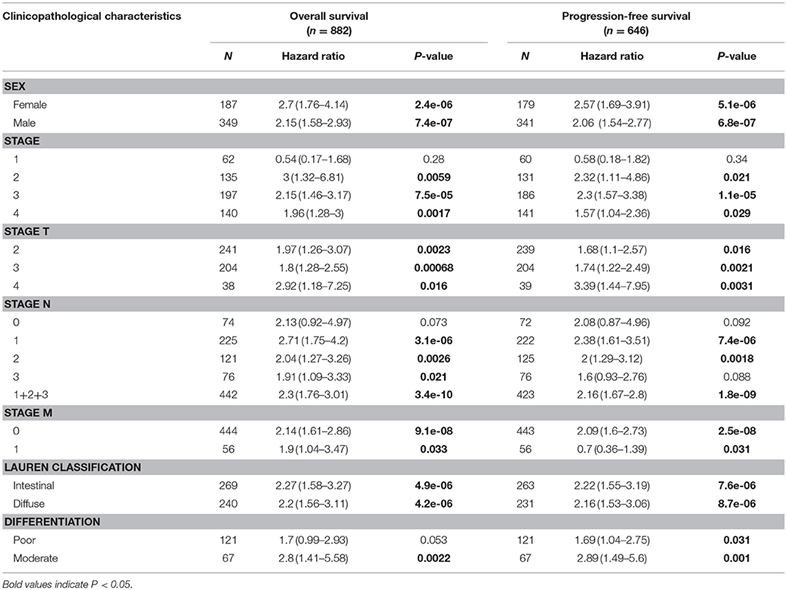
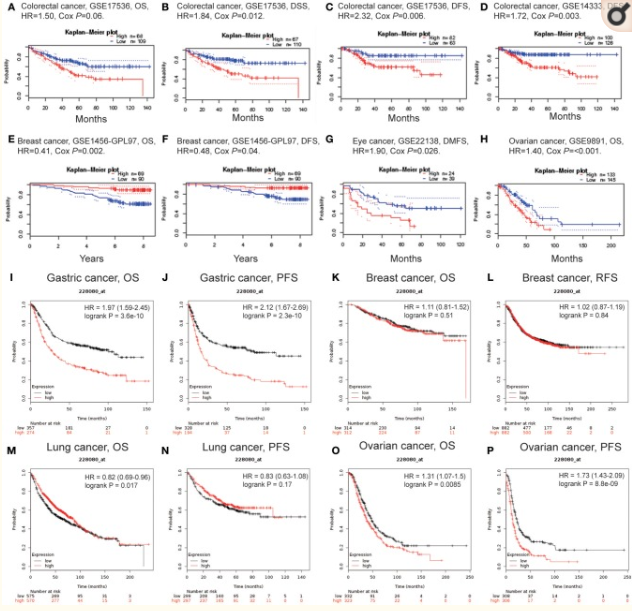 三、我们具体的分析了一下胃癌样本中LAYN基因表达与临床特征的关系,我们发现LAYN的过度表达与男性和女性患者的OS和PFS恶化以及两种Lauren分类和分化相关(P <0.05),高LAYN表达在四个淋巴结转移类别中具有最高的OS N和PFS的N 1值。这些结果表明LAYN表达水平可影响胃癌淋巴结转移患者的预后。(可能我们每个癌症都分析了一下这个结果,谁知道呢~)
三、我们具体的分析了一下胃癌样本中LAYN基因表达与临床特征的关系,我们发现LAYN的过度表达与男性和女性患者的OS和PFS恶化以及两种Lauren分类和分化相关(P <0.05),高LAYN表达在四个淋巴结转移类别中具有最高的OS N和PFS的N 1值。这些结果表明LAYN表达水平可影响胃癌淋巴结转移患者的预后。(可能我们每个癌症都分析了一下这个结果,谁知道呢~)
四、肿瘤浸润性淋巴细胞是在癌症前哨淋巴结状态和存活的独立预测因子,我们研究了LAYN表达是否与不同类型癌症中的免疫浸润水平相关,首先我们从TIMER数据库下载了39种癌症的六种免疫浸润细胞的得分数据,分别分析了LAYN表达与这些免疫细胞得分的相关性,发现在CODA和STAD中,LAYN表达水平与CD8+ T cells 、CD4+ T cells、macrophages 、neutrophils和DCs 有显著正相关。
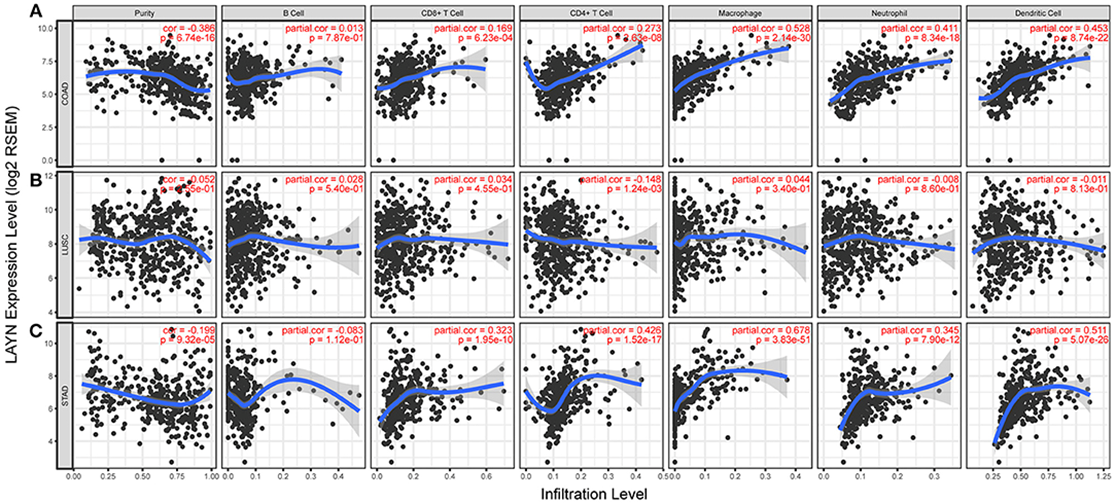
五、既然在CODA和STAD中LAYN表达于免疫细胞这么相关,我们便找来一些其他已报到的免疫浸润相关的基因集,看看这些基因集与LAYN的相关性,结果显示LAYN表达水平与各种免疫细胞和COAD和STAD中不同T细胞的大多数免疫标记物组显着相关。
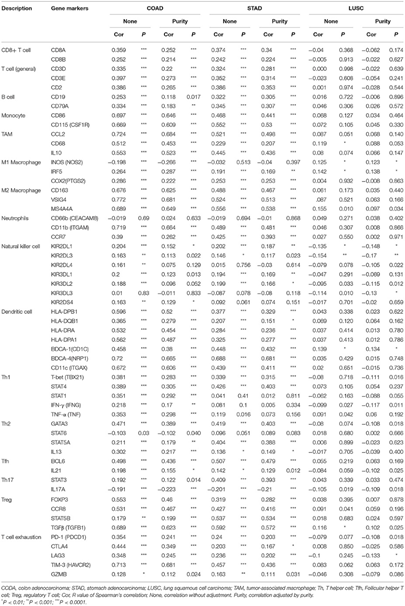
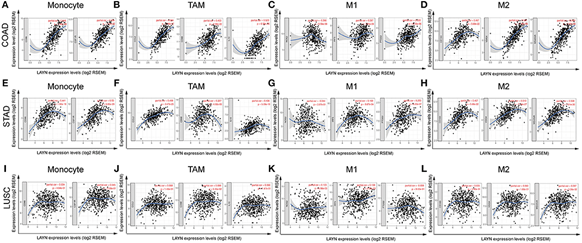
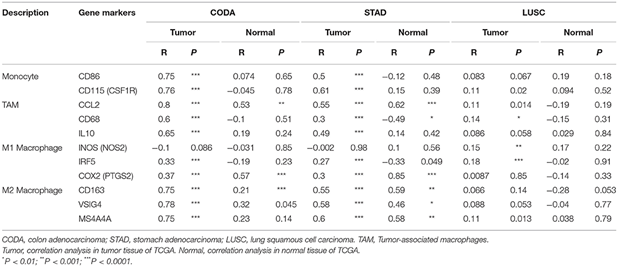
以上五个方面便是此文的所有结果,看完是不是有新的思路和想法呢
两者结合一下,当你面对只有一个基因时该怎么做应该明白了吧,总有一个阳性结果是你的。
- 发表于 2019-02-22 12:10
- 阅读 ( 26098 )
- 分类:方案研究
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9597 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10575 浏览
- TCGA数据库 7809 浏览
- 文献解读(1)用免疫风险评分预测黑色素瘤转移的风险 7245 浏览
- TCGA下载工具超级版,在线秒下载 7594 浏览
- 在GEO数据库如何找带有预后信息的数据?一分钟搞定,适用于找任何的样本特征数据 21048 浏览
