使用sigclust来评估你的聚类是否显著
看教程不够直观,那就看视频吧! >>点击加载视频
看到文献中(doi:10.1080/2162402X.2017.1392427)有人使用sigclust来评估聚类是否合理,于是乎研究了这个包。
sigclust主要针对生物信息学数据(高维低样本)产生的聚类结果进行评估,一个基本假设就是数据满足单样本正态分布,计算聚类系数cluster index,进而通过蒙特卡罗随机过程计算该系数的统计学显著性——p值。 根据p值评估聚类的效果以及聚类的结果是否是真实存在的簇,借鉴到二分k均值算法,还可以用来对多类的数据进行聚类操作
优点:计算速度快,特别适合生物信息学中的高维低样本数据
局限:只能对两分类的聚类结果进行评估,如果是多类就必须配对两两比较
参数
x 一个矩阵或者data frame数据。行代表样本,列代表属性,数据需要标准化并不包含缺失值
nsim 模拟正态分布样本数(迭代循环次数),最终求得主要p值
nrep 2-means聚类的步数(默认=1,用以优化速度),如果labflag为0,则无效
labflag 如果p值用于一类或两类所指定的变量,用户定义的簇labflag=1,其他的labflag=0
label 如果labflag=0,label通过2-means簇产生,如果labflag=1,用户自行定义,必须为1和2的整数向量,长度和行数一致
icovest 协方差估计方式:1.软阈值方法,constrained MLE(默认)
2.样本协方差估计(当诊断失败建议使用)
3.使用原始背景噪声阈值估计(硬阈值方法
返回值
raw.data 原始数据矩阵
veigval 样本特征值向量
vsimeigval 在模拟中使用的特征值向量
simbackvar 来自数据的背景方差拟合
icovest 协方差估计方式
nsim 模拟正态分布的样本个数
simcindex 基于nsim模拟数据集的cluster index
pval 基于经验分布分位数模拟的p值
pvalnorm 基于正态分布分位数模拟的p值
xcindex 基于给定数据集的cluster index
用法极其简单如下:
sigclust(data,nsim=1000,nrep=1,labflag=0,icovest=3)@pvalnorm
我的使用情景是这样的,我利用ConsensusClusterPlus来对数据进行分子分型,最后得到了四个亚型
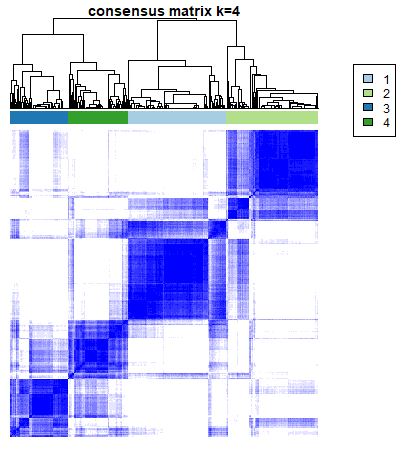 利用sigclust我想看一下这四个亚型的聚类是否显著于是乎就有了如下代码。
利用sigclust我想看一下这四个亚型的聚类是否显著于是乎就有了如下代码。
library(ConsensusClusterPlus)
output='conse'
pdf=F
results = ConsensusClusterPlus(data,maxK=8,reps=50,pItem=0.8,pFeature=1,
title=output,distance="euclidean",clusterAlg="km"
#,distance="pearson"
,seed=1262118388.71279,plot=ifelse(pdf,'pdf','png'))
rcc.ind=4
table(results[[rcc.ind]]$consensusClass)#查看各个亚型的样本数目
library(sigclust)
sitcl.p=matrix(1,nrow = rcc.ind,ncol = rcc.ind)
for(i in 1:(rcc.ind-1)){
for (j in (i+1):(rcc.ind)) {
sitcl.p[i,j]=sigclust(data[,which(results[[rcc.ind]]$consensusClass==i|results[[rcc.ind]]$consensusClass==j)]
,nsim=1000,nrep=1,labflag=1,label=ifelse(results[[rcc.ind]]$consensusClas[which(results[[rcc.ind]]$consensusClass==i|results[[rcc.ind]]$consensusClass==j)]==i,1,2),icovest=3)@pvalnorm#评论两个亚型的聚类显著性
sitcl.p[j,i]=sitcl.p[i,j]
}
}
后记:其实没什么用,大多数情况下都是显著的。
- 发表于 2019-02-23 14:36
- 阅读 ( 6862 )
- 分类:软件工具
你可能感兴趣的文章
- 简易一致性聚类分析软件(ConsensusClusterPlus) 11767 浏览
- ConsensusClusterPlus未选择最优阈值的用法案例 6965 浏览
