菜鸟第一次提取TCGA编码蛋白基因和lncRNA表达谱
看教程不够直观,那就看视频吧! >>点击加载视频
本人生物信息菜鸟一枚,第一次自己提取TCGA编码蛋白和lncRNA表达谱。
数据准备及介绍
利用简易TCGA下载工具,下载胶质瘤数据(表达谱数据)
双击所要选择的癌症(默认选择原发癌和癌旁正常组织)
单击重新检索按钮下载资源列表,并根据下载资源列表选择下载数据类型
点击下载,选择路径,开始下载任务
通过合并文件按钮将单个文件合并生成矩阵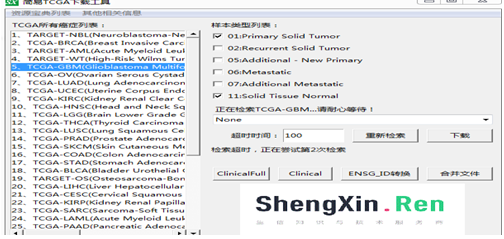
数据每一列代表一个ENSG_ID,每一列代表一个样本
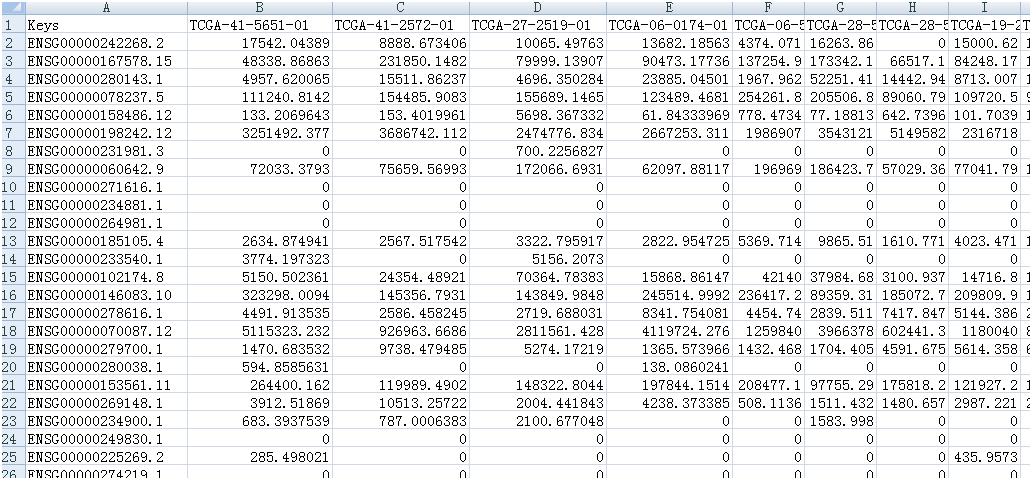
在ensembol数据库下载数据
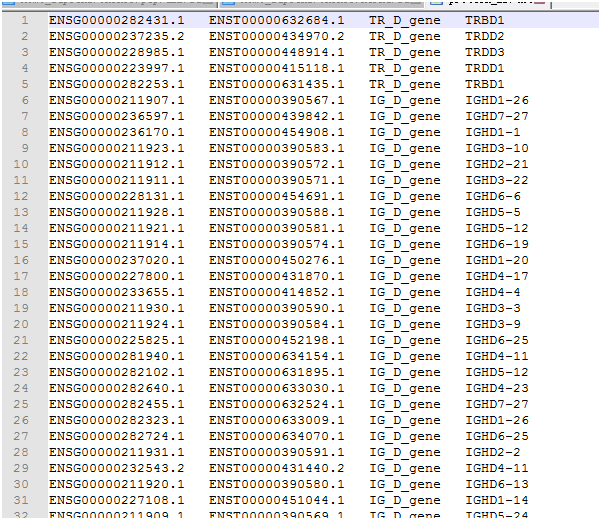
选human-protein sequence,数据大体内容如下
>ENSP00000487941.1 pep chromosome:GRCh38:7:142786213:142786224:1 gene:ENSG00000282431.1 transcript:ENST00000632684.1 gene_biotype:TR_D_gene transcript_biotype:TR_D_gene gene_symbol:TRBD1 description:T-cell receptor beta diversity 1 [Source:HGNC Symbol;Acc:HGNC:12158]
GTGG
数据包含ENSP_ID,染色体位置,ENSG_ID,ENST_ID, gene_symbol, gene_biotype,
transcript_biotype等信息(空格分隔 )
从中提取ENSG_ID ,ENST_ID, gene_symbol, gene_biotype制成表格 ,用于以后比对提取。
数据处理与结果
获取蛋白质基因列表
import re
def findlabel(line,opt,beg):
str1=line[line.find(opt):len(line)]
if str1.find(' ') > -1:
str1=str1[beg:str1.find(' ')]
else:
str1=str1[beg:len(str1)]
return str1.strip()
def searchprotein():
file1='D:/Homo_sapiens.GRCh38.pep.all.fa'
f=open(file1,'r')
lines=f.readlines()
f.close()
list1=[]
for line in lines:
line=line.rstrip('\n').strip()
if line.find('>')==0:
enst=findlabel(line, 'ENST',0)
ensg=findlabel(line, 'gene:',5)
type1=findlabel(line,'gene_biotype:',13)
gene_symbol=findlabel(line, 'gene_symbol:', 12)
list1.append((ensg,enst,type1,gene_symbol))
fw=open('D:/protein_ID.txt','w') #写文件
for l in list1:
fw.write('\t'.join(l)+'\n')
fw.close()
if __name__ == '__main__':
searchprotein()
形成文件
获取蛋白质表达谱:
def getprotein():
file1='D:/protein_ID.txt'
f=open(file1,'r') #读取文件
lines=f.readlines()
protein1=[]
for line in lines:
line=line.rstrip().strip()
pro=line.split('\t')[0]
if pro notin protein1:
protein1.append(pro) #在最末尾添加lnc
return protein1
def getproteinExpre():
protein1=getprotein()
file1='D:/Merge_matrix.txt'
f=open(file1,'r')
lines=f.readlines()
f.close()
fw=open('D:/proteinexp.txt','w')
for line in lines:
line=line.rstrip().strip()
list1=line.split('\t',1)
if(list1[0] in protein1):
fw.write(line+"\n")
fw.close()
if __name__ == '__main__':
getproteinExpre()
得到结果
lncRNA表达谱提取:
lncRNA表达谱获取与上述类似
只需更改下列内容
ensemble中选择下载human-ncRNA数据
在ncRNA数据中要选取lncRNA类型
程序做适当更改
list1.append((ensg,enst,type1,gene_symbol))改为:
match=re.match(r'lincRNA|macro_lncRNA|bidirectional_promoter_lncRNA',type1)
if match
list1.append((ensg,enst,type1,gene_symbol))
随后的处理不变。
本人菜鸟一枚对很多问题理解还很欠缺,程序参考了https://www.shengxin.ren/question/23内容,有问题还希望大家指出
- 发表于 2017-05-07 18:13
- 阅读 ( 12668 )
- 分类:方案研究
你可能感兴趣的文章
- cBioportal (含TCGA)数据的分组与下载 9655 浏览
- 最新5+套路:免疫基因对已成过去式,新的对子再次出发!细胞与细胞相互作用换个角度看肿瘤预后 10610 浏览
- TCGA数据库 7835 浏览
- TCGA下载工具超级版,在线秒下载 7639 浏览
- 生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物 10161 浏览
- 生信扯上免疫相关就是容易上五分啊 14834 浏览
