KEGG Pathway 中基因的颜色怎么简单的标记上去
看教程不够直观,那就看视频吧! >>点击加载视频
在组学的文章中经常会看到各种KEGG Pathway,上面的基因经常会被标记成不同的颜色,而不同的颜色代表基因的不同的差异水平,那么除了公司流程中能够将KEGG pathway的基因标上去颜色之外还有什么工具也能方便的标记呢,事实上这是一个数据可视化的过程,在做科研的过程中常常存在不同的需求,比如在富集的KEGG Pathway中标记出基因的上下调情况(这是最常见的),比如做实验的过程中找到了几个基因,想知道这几个基因的在KEGG pathway中的调控关系等,这种情况下就属于高度定制了,那么今天就来讲解一下自己在不编程的情况下如何给KEGG pathway中标上颜色。
所以今天介绍的这个KEGG Mapper工具能帮上你大忙,会用他能让你轻松好多,他的网址:http://www.genome.jp/kegg/tool/map_pathway2.html
KEGG Mapper工具主要的上色流程是这样的:线将你输入的基因匹配到KEGG后台的数据库,找到这些基因所参与的通路,然后再在这些通路中标记上颜色,over!
首先看看他的例子:
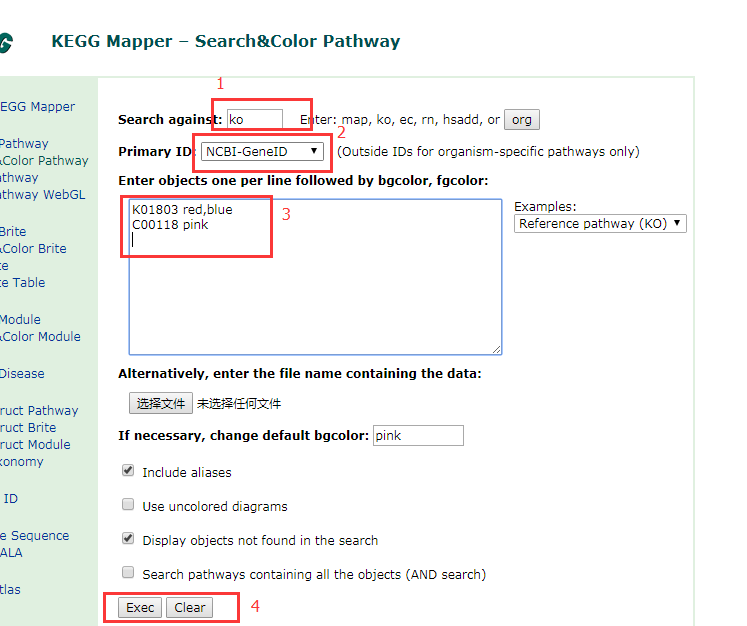 从图中可以看出只需要点3个地方,一个输入即图中的1、2、3、4
从图中可以看出只需要点3个地方,一个输入即图中的1、2、3、4
1、选择你的物种,看到右侧的org按钮没,点击他弹出物种选择栏,如果不选择那么默认就是ko,默认ko呢会有一个小问题就是你可能输入的是动物的基因,但是也能匹配到光合作用同了。。。。。因为你没限定物种呀
2、选择你输入的基因ID的类型,这里就比较坑爹了,就四种ID可选,不过也没关系,可以选使用其他工具转换成合适的ID,工具很多
3、输入你准备标记颜色的基因以及相应的颜色,以tab健隔开(就是两列啦),看到例子中的颜色没,第一个是red,blue 这里同时两个颜色表示啥意思呢,一试便知,静待结果
4、点击运行即可,结果如下:
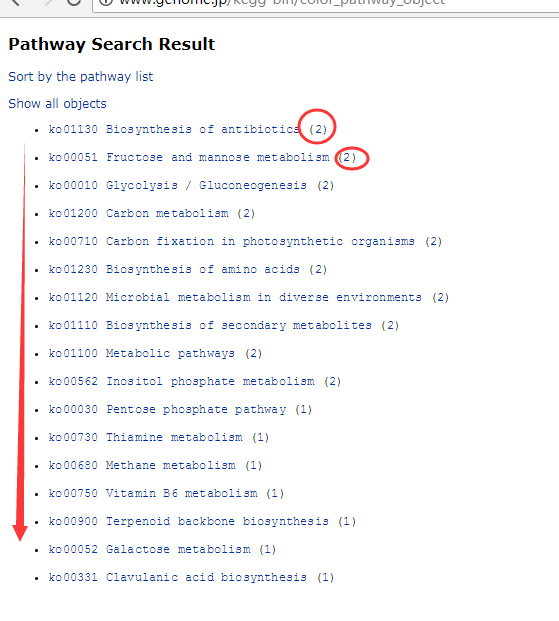
从结果中可以看出列出了这两个基因所参与的所有的通路,我们可以随意的点击一个进去看看
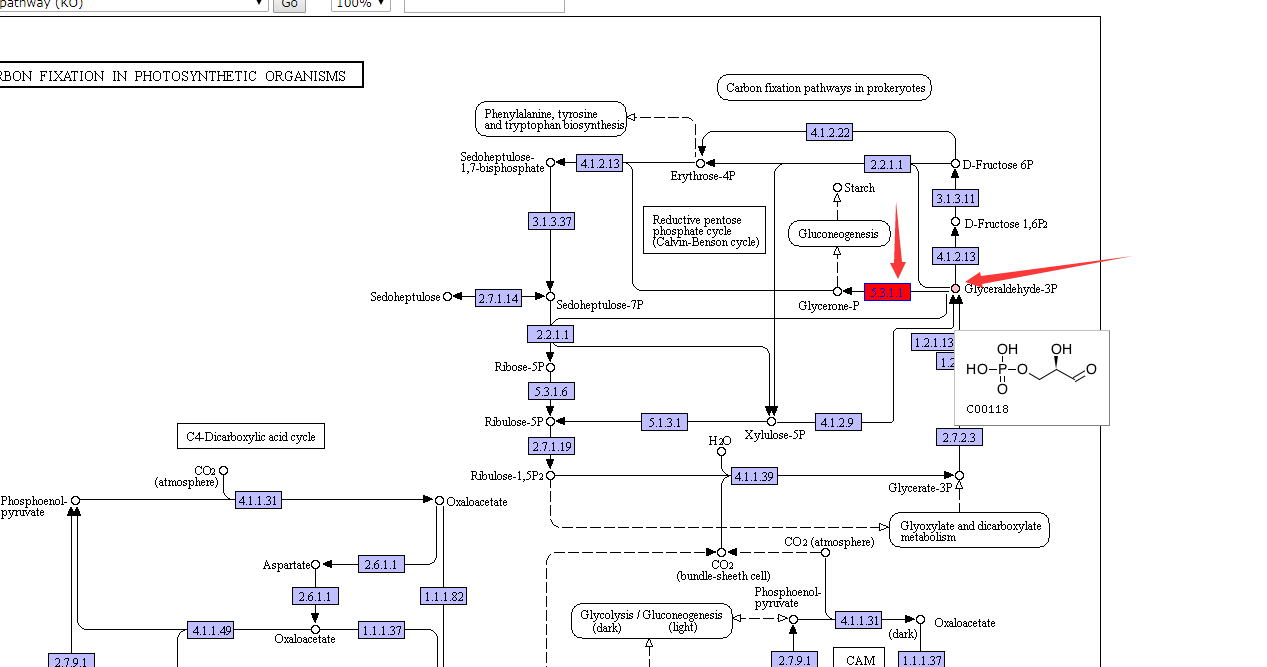
从图中可以看出两个基因都标上了颜色,还记得那个标记red,blue嘛,标记上的是边框blue,填充red
下面来个真实的例子,首先假装手上有一些DNA修复相关的基因,然后我们想将他标记成红色,从DNA修复通路中看他们的调控关系
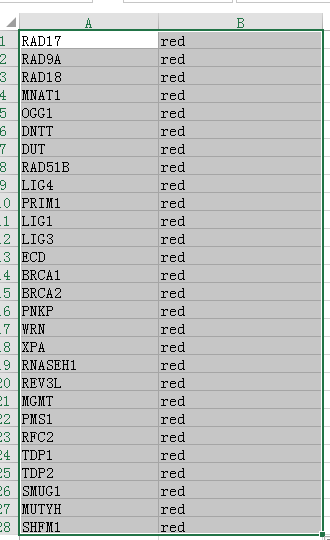
很显然KEGG不支持gene symbol的输入方式,所以我们选择uniprot的ID,此时我们使用http://www.uniprot.org/uploadlists/ idmapping工具进行ID转换
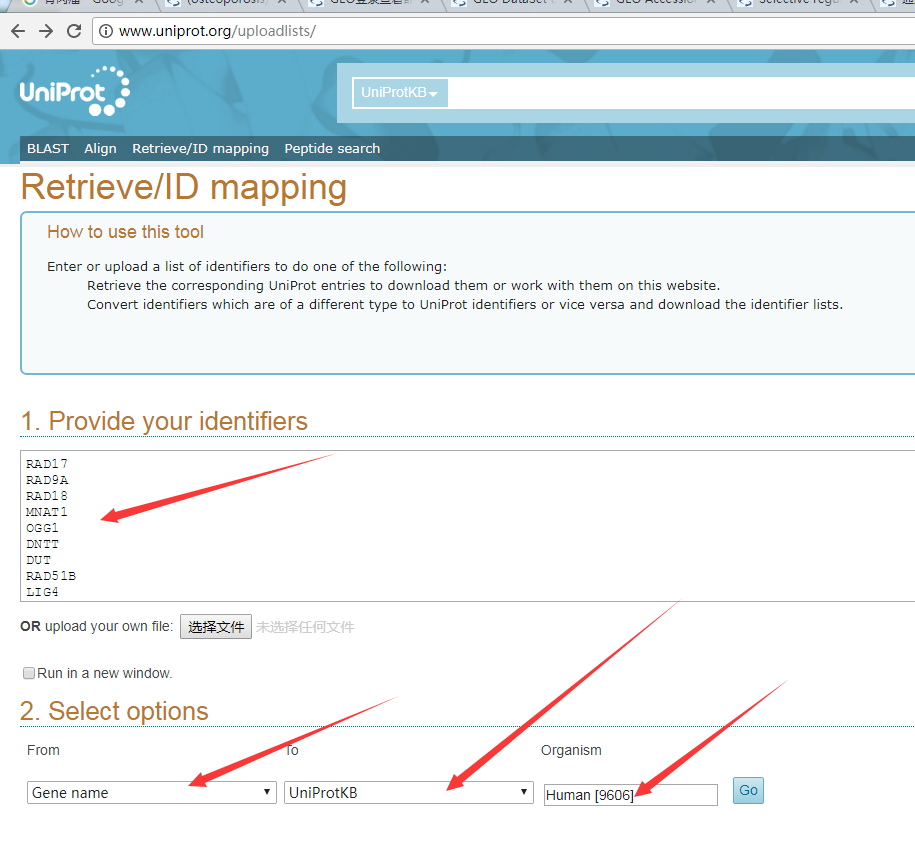
得到转换结果:
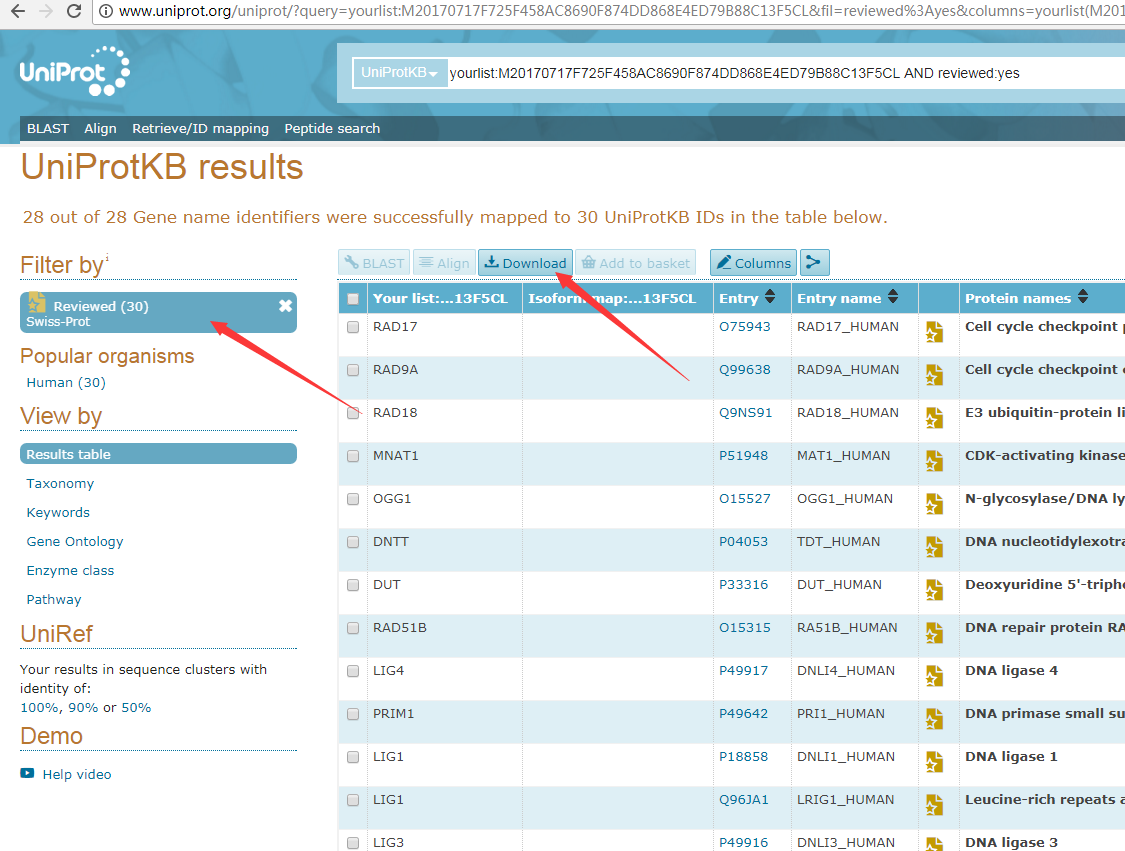 下载结果:
下载结果:
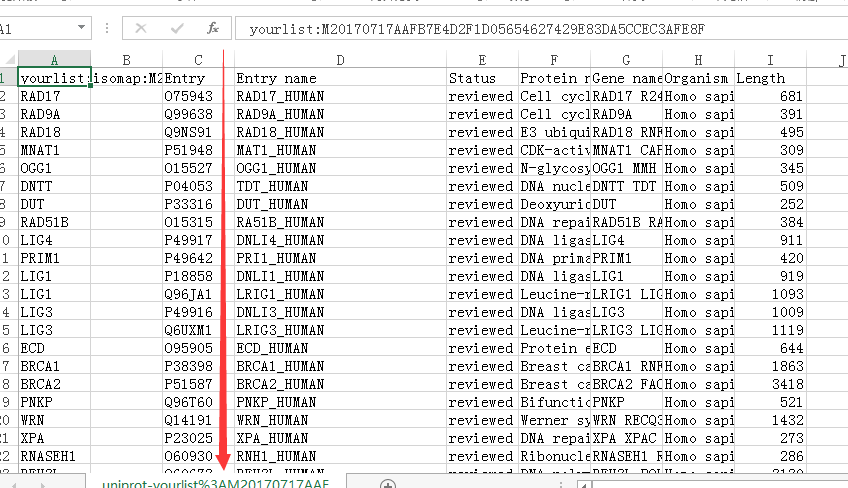 提交到KEGG Mapper:
提交到KEGG Mapper:
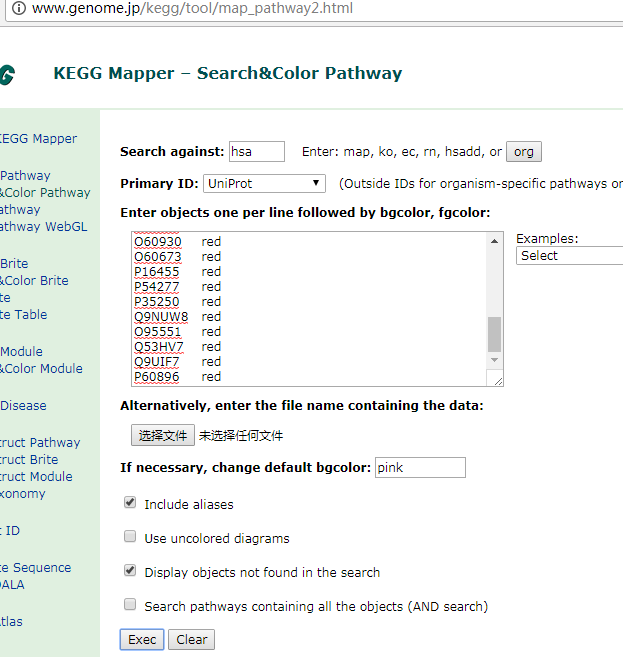
靓丽的结果出现:
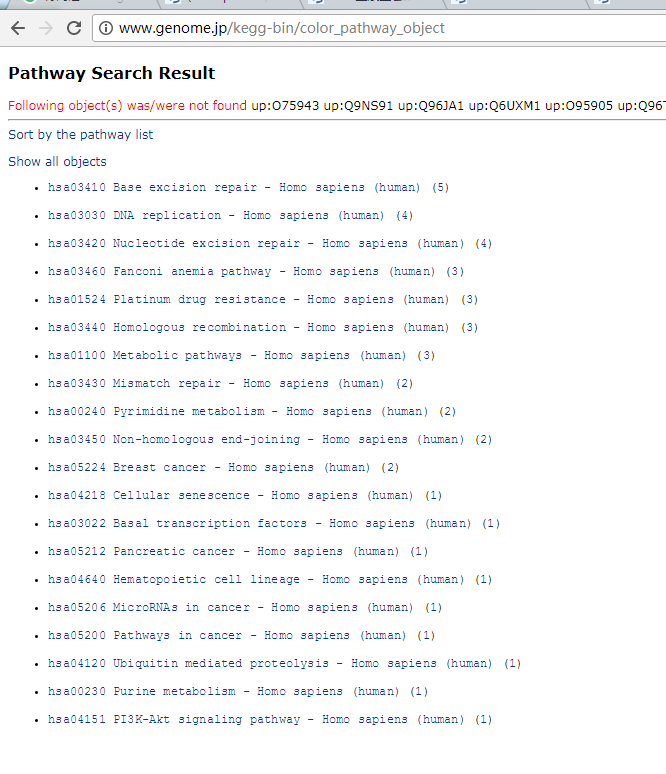
最终的显示结果如下:
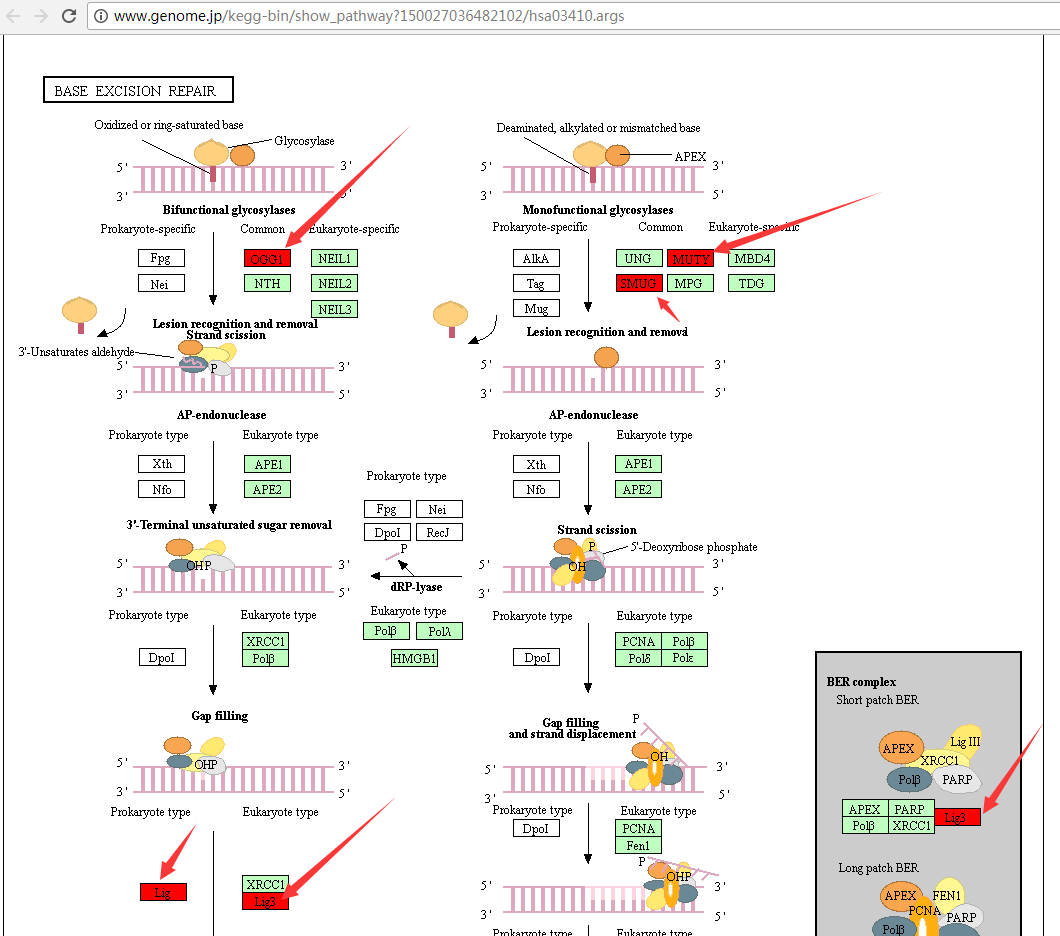
是不是很easy!!!
至于高级复杂版可以从两个角度去实现批量化,一个是KEGG API,一个是构建url,当然也可以使用程序绘图直接绘制上去,这里就不做介绍了,还有就是对于未知的物种该如何做,首先是进行比对,具体工具有KOBAS、KAAS等。
- 发表于 2017-07-17 13:49
- 阅读 ( 19954 )
- 分类:软件工具
你可能感兴趣的文章
- 分享一段从KEGG通路中提取基因互作数据的R代码 8428 浏览
- KEGG通路图定制化绘制工具 16788 浏览
- GEO数据挖掘开篇之文章解析 23367 浏览
相关问题
- 如何批量下载文件(xml文件),用迅雷下载的不是xml文件 1 回答
- 问下Gsea和go,kegg 优势在哪,写文章如何取舍 3 回答
- 使用R的clusterProfiler包无法正确匹配 3 回答
- 各位老师,请问怎么用KEGG对共表达基因做KEGG通路富集呢?(试过DAVID,里面的结果不是很理想) 2 回答
- 得到基因的想问一下 已经拿到了基因的COG注释,NR注释,SWISSPROT注释,KEGG注释,GO注释,INTEPRO注释 怎么进行下一步功能预测 1 回答
- 本人生信小白一枚,想问大家一个问题,最近在做RNA_seq分析,想问你kegg pathway怎么分析?如何画下图的B和D? 1 回答
