这应该是史上最简单好用的GSEA富集分析工具了
看教程不够直观,那就看视频吧! >>点击加载视频
GSEA是什么,这个请自行百度,在使用之前要知道GSEA是啥
工具下载链接:http://gap.shengxin.ren/tool/4/
然后上工具截图:
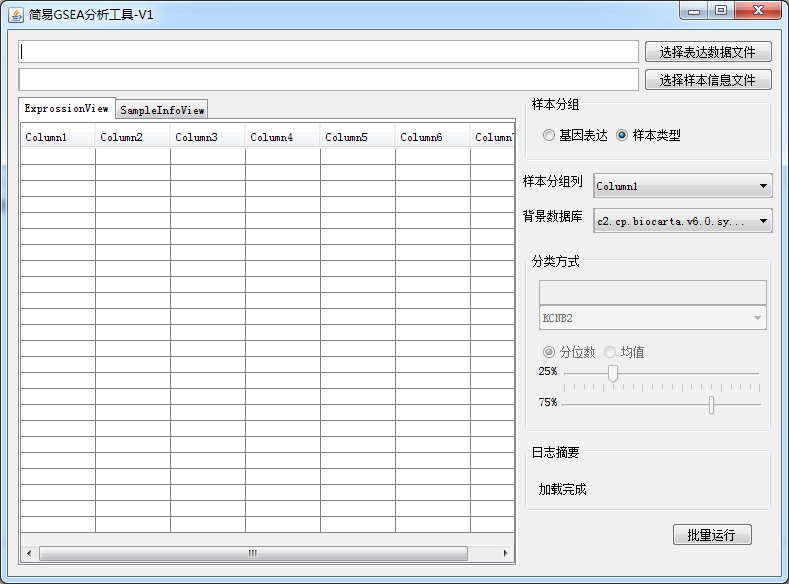 一般情况下咱们要做GSEA需要准备三个文件
一般情况下咱们要做GSEA需要准备三个文件
1、gct格式的表达谱
2、分组文件
3、富集的背景文件,也就是gmt格式的文件
有了这三个文件之后就可以做GSEA富集分析啦
但是这对于很多种情况下都极不方便,比如想看一下某个表达谱中某个基因的高表达和低表达的样本的通路差异,这就需要咱们自己编程手动分组后再做GSEA,这要是一个还好,那多个基因呢
本工具着重解决这些繁琐问题
从软件界面中可以看出输入文件只有表达谱,和样本信息表,而样本信息表并不是必须的,如果你要按照样本信息对样本进行分类然后做GSEA,那么可以导入样本信息表即可,这里注意哦,软件会自动匹配样本信息,无需一一对应,很亲民。
那么就用一个例子来玩一玩,
使用GSE44076这套数据,这套数据下载和预处理教程看这里:https://www.shengxin.ren/article/135
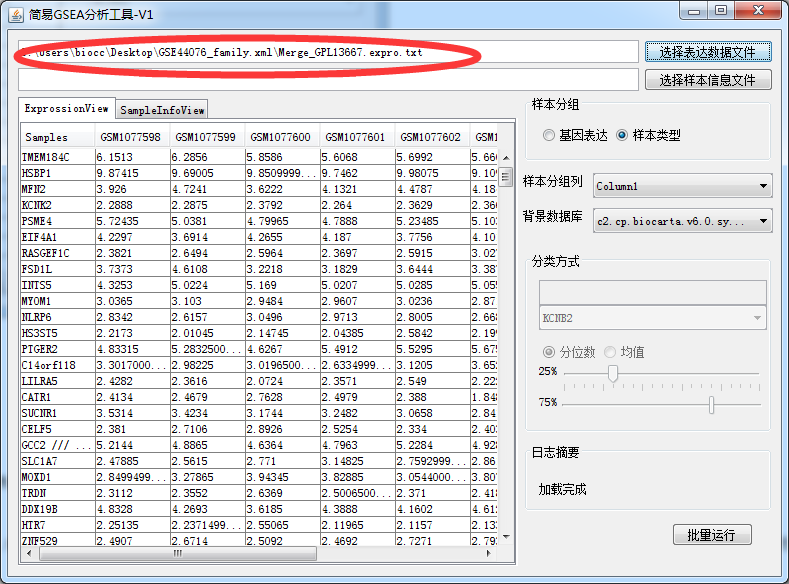
现在咱们使用已经处理好的表达谱矩阵和样本信息,首先导入表达谱矩阵
 然后咱们看看ERBB2的表达量最高的前25%和最低的前25%的样本的KEGG 通路富集差异,如图:
然后咱们看看ERBB2的表达量最高的前25%和最低的前25%的样本的KEGG 通路富集差异,如图:
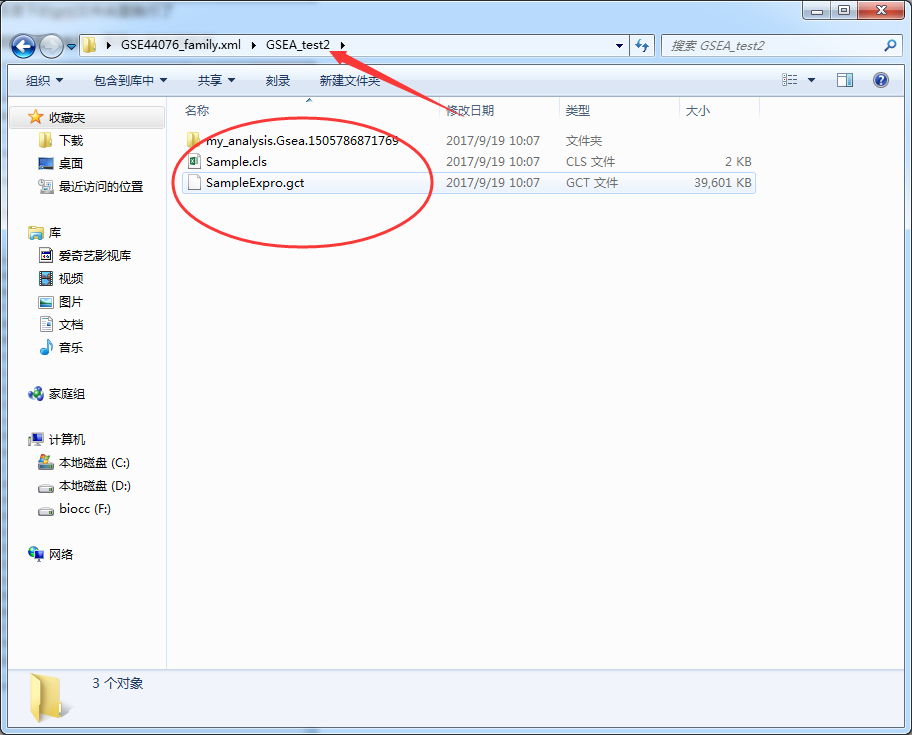
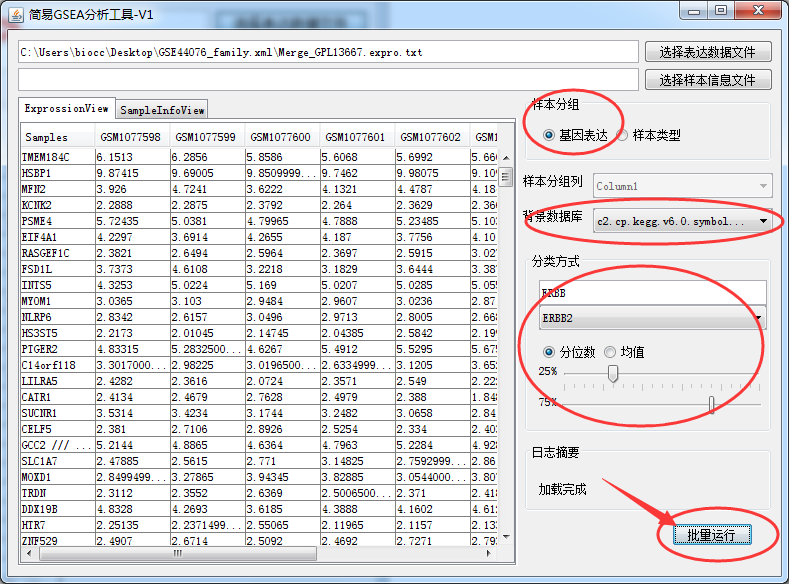 点击批量运行,选择结果保存目录,稍微等一会儿就可以了,最终在保存目录下生成了三个文件,gct和cls可以用于自己去重复这个结果啦
点击批量运行,选择结果保存目录,稍微等一会儿就可以了,最终在保存目录下生成了三个文件,gct和cls可以用于自己去重复这个结果啦
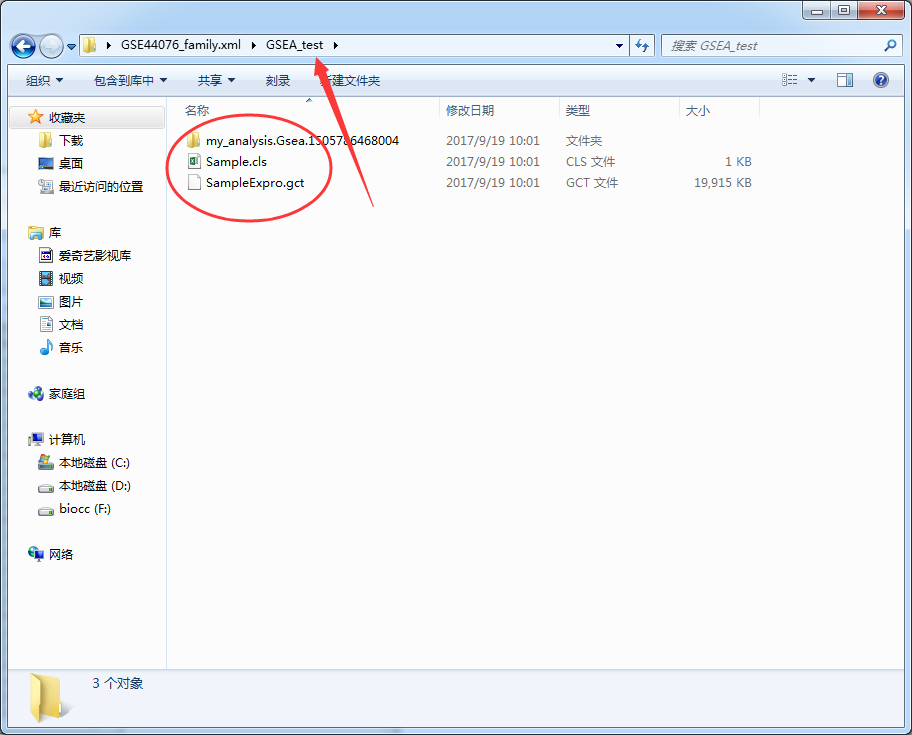 my_analysis.Gsea.开头的文件夹保存的就是GSEA富集分析的结果,来看一眼
my_analysis.Gsea.开头的文件夹保存的就是GSEA富集分析的结果,来看一眼
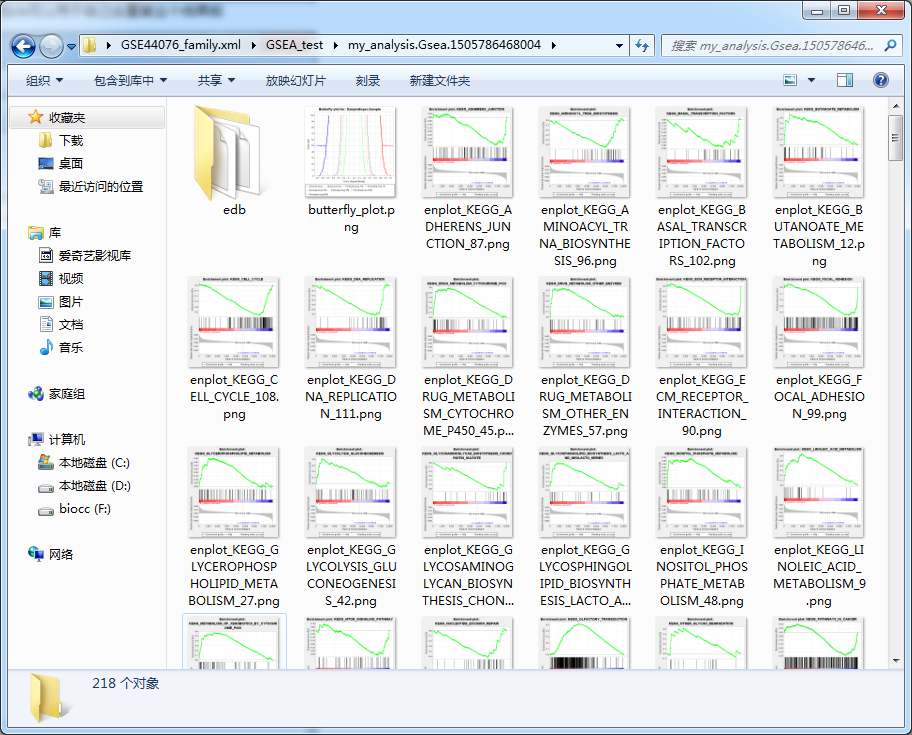 当然这个软件默认提供了六种gmt数据,如果需要自己定制的话也很简单,只有把gmt文件拷贝到软件目录下的gmt文件夹里就行了
当然这个软件默认提供了六种gmt数据,如果需要自己定制的话也很简单,只有把gmt文件拷贝到软件目录下的gmt文件夹里就行了
然后咱们刚才演示了做单个基因的,现在来使用样本信息,比如使用性别分类来看男和女对于KEGG通路富集的差异,再导入样本信息表
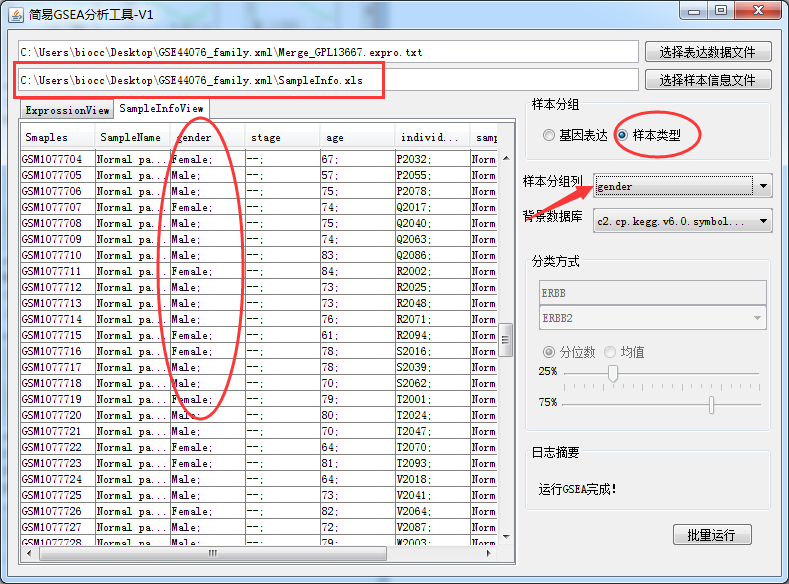 点击批量运行,选择结果保存目录,稍等一会儿
点击批量运行,选择结果保存目录,稍等一会儿
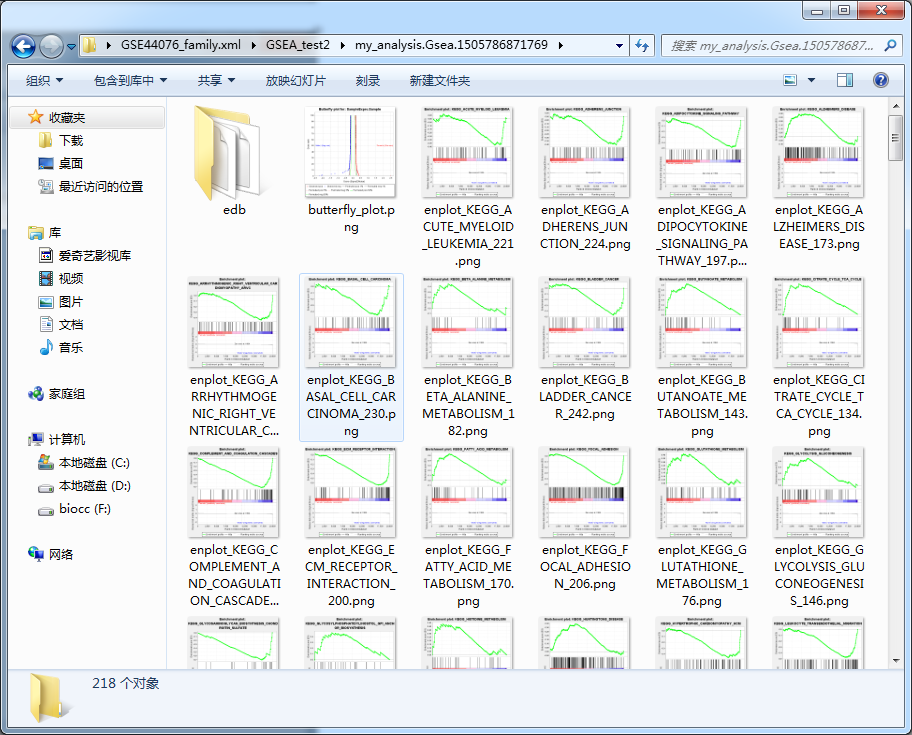
 华丽丽的结果出来了
华丽丽的结果出来了
 还有一点不完美的是结果解读和美化部分,坐等小编有时间给大家分享吧~~
还有一点不完美的是结果解读和美化部分,坐等小编有时间给大家分享吧~~
- 发表于 2017-09-19 10:12
- 阅读 ( 64631 )
你可能感兴趣的文章
- GSEA简易分析及可视化工具 7170 浏览
- GSEA 富集分析简述 19407 浏览
- ssGSEA算法原理解析 36164 浏览
- GEO和TCGA数据库挖掘文章思路! 31816 浏览
- 手把手五分钟教会你如何做GESA富集分析 24938 浏览
