WGCNA 一键式分析工具
看教程不够直观,那就看视频吧! >>点击加载视频
加权基因共表达网络分析 (WGCNA, Weighted correlation network analysis)是用来描述不同样品之间基因关联模式的系统生物学方法,可以用来鉴定高度协同变化的基因集, 并根据基因集的内连性和基因集与表型之间的关联鉴定候补生物标记基因或治疗靶点。
相比于只关注差异表达的基因,WGCNA利用数千或近万个变化最大的基因或全部基因的信息识别感兴趣的基因集,并与表型进行显著性关联分析。一是充分利用了信息,二是把数千个基因与表型的关联转换为数个基因集与表型的关联,免去了多重假设检验校正的问题。
理解WGCNA,需要先理解下面几个术语和它们在WGCNA中的定义:
共表达网络:定义为加权基因网络。点代表基因,边代表基因表达相关性。加权是指对相关性值进行冥次运算 (冥次的值也就是软阈值 (power, pickSoftThreshold这个函数所做的就是确定合适的power))。无向网络的边属性计算方式为 abs(cor(genex, geney)) ^ power;有向网络的边属性计算方式为 (1+cor(genex, geney)/2) ^ power; sign hybrid的边属性计算方式为cor(genex, geney)^power if cor>0 else 0。这种处理方式强化了强相关,弱化了弱相关或负相关,使得相关性数值更符合无标度网络特征,更具有生物意义。如果没有合适的power,一般是由于部分样品与其它样品因为某种原因差别太大导致的,可根据具体问题移除部分样品或查看后面的经验值。
Module(模块):高度內连的基因集。在无向网络中,模块内是高度相关的基因。在有向网络中,模块内是正相关的基因。把基因聚类成模块后,可以对每个模块进行三个层次的分析:1. 功能富集分析查看其功能特征是否与研究目的相符;2. 模块与性状进行关联分析,找出与关注性状相关度最高的模块;3. 模块与样本进行关联分析,找到样品特异高表达的模块。
Connectivity (连接度):类似于网络中度 (degree)的概念。每个基因的连接度是与其相连的基因的边属性之和。
Module eigengene E: 给定模型的第一主成分,代表整个模型的基因表达谱。
Intramodular connectivity: 给定基因与给定模型内其他基因的关联度,判断基因所属关系。
Module membership: 给定基因表达谱与给定模型的eigengene的相关性。
Hub gene: 关键基因 (连接度最多或连接多个模块的基因)
Adjacency matrix (邻接矩阵):基因和基因之间的加权相关性值构成的矩阵
TOM (Topological overlap matrix):把邻接矩阵转换为拓扑重叠矩阵,以降低噪音和假相关,获得的新距离矩阵,这个信息可拿来构建网络或绘制TOM图
基本分析流程:
构建基因共表达网络:使用加权的表达相关性。
识别基因集:基于加权相关性,进行层级聚类分析,并根据设定标准切分聚类结果,获得不同的基因模块,用聚类树的分枝和不同颜色表示。
如果有表型信息,计算基因模块与表型的相关性,鉴定性状相关的模块。
研究模型之间的关系,从系统层面查看不同模型的互作网络。
从关键模型中选择感兴趣的驱动基因,或根据模型中已知基因的功能推测未知基因的功能。
总结如上WGCNA分析共分为三步:
1、选择合适的软阈值
2、确定共表达模块
3、分析模块和表型的关系
在这里我们开发了一个 一键式的可交互的WGCNA分析工具,仅需要提供表达谱即可进行分析,输入数据界面如下:
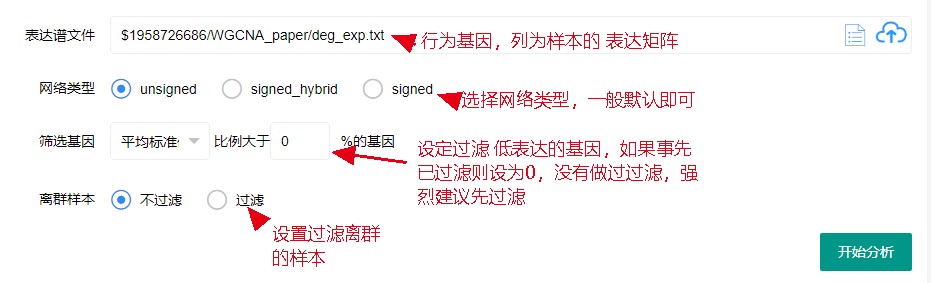
从输入界面看 已经是非常简单,你只需要准备好表达谱矩阵就可以了,但是 需要注意的是 并不是啥表达谱矩阵都能往里放的,首先 你表达谱的基因个数不能太少,太少的话容易找不到合适的软阈值,基因个数不能太多,太多计算量非常大,很多时候并没有必要,建议事前先做过滤,载入之后就点击分析
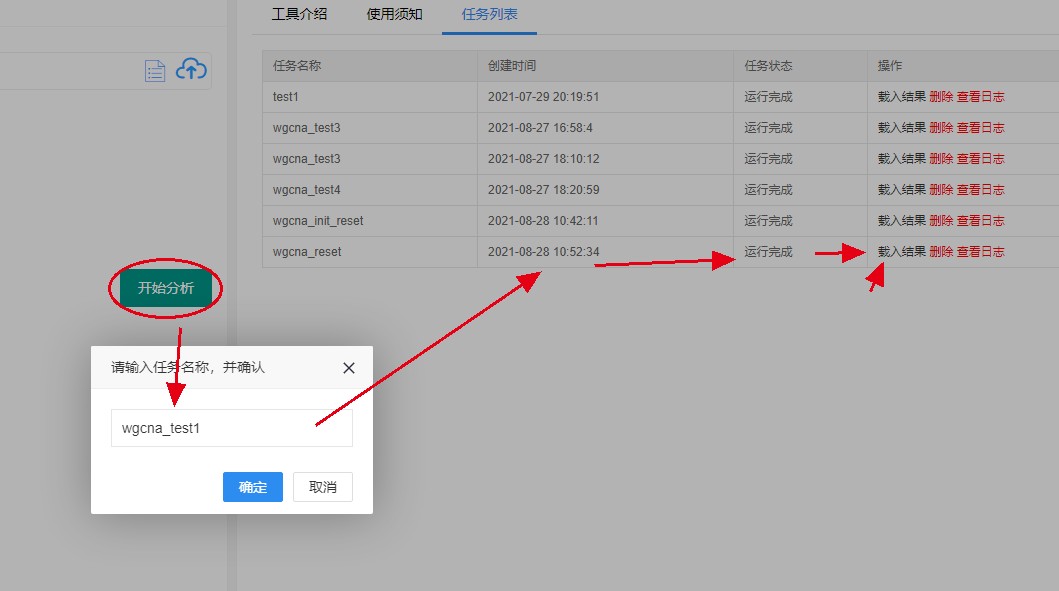
耐心等待运行完成,这里 根据输入的表达谱的行数不同时间长短不同,一般几千行的话很快就运行完,运行完成后后侧可以看到 载入结果 四个字,点击 打开页面即可看到运行结果了
载入结果之后,页面下方分五大板块:
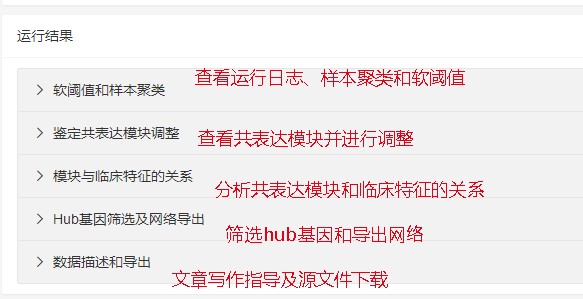
第一个板块展示如下:
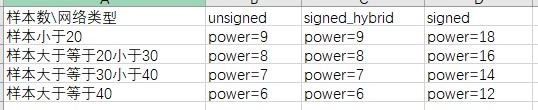
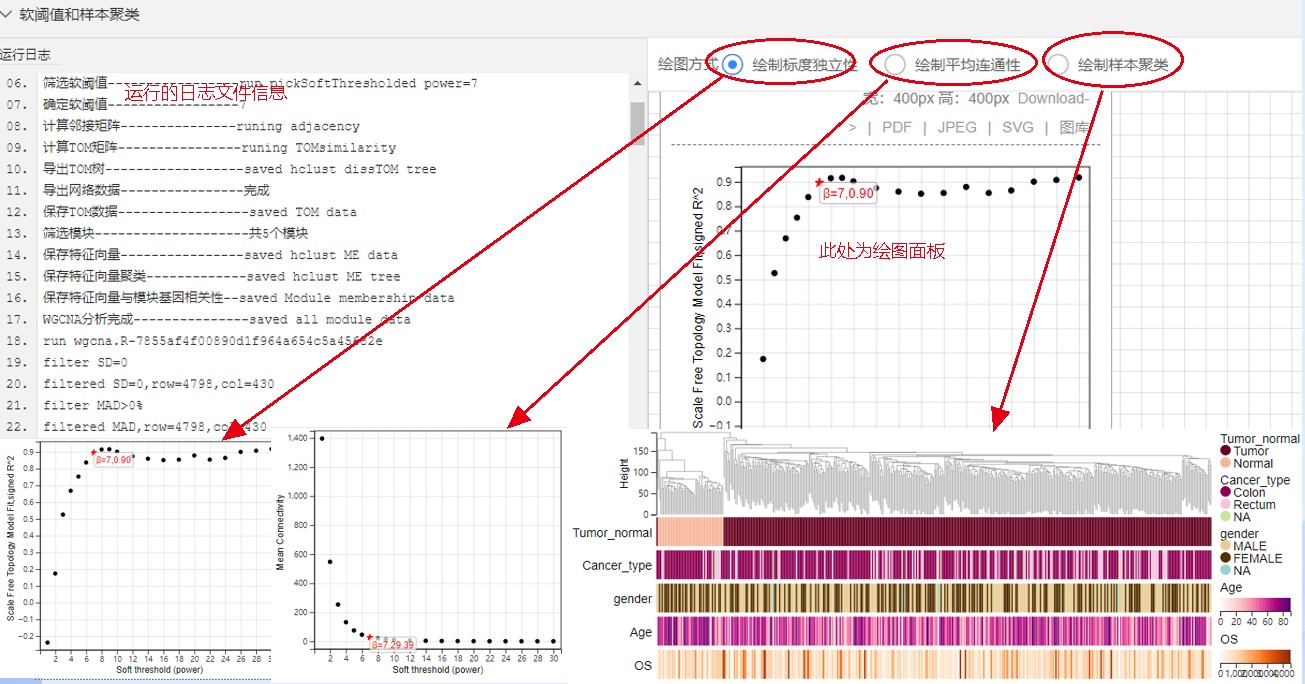 这里需要特别注意的是 软阈值 ,当程序找不到最优软阈值时,使用 经验软阈值
这里需要特别注意的是 软阈值 ,当程序找不到最优软阈值时,使用 经验软阈值
# 无向网络在power小于15或有向网络power小于30内,没有一个power值可以使用
# 无标度网络图谱结构R^2达到0.8,平均连接度较高如在100以上,可能是由于部分样品与其他样品差别太大。这可能由批次效应、样品异质性或实验条件对表达影响太大等造成。可以通过绘制样品聚类查看分组信息和有无异常样品。
程序自动会使用下面的经验power值。
第二个板块,模块的调整
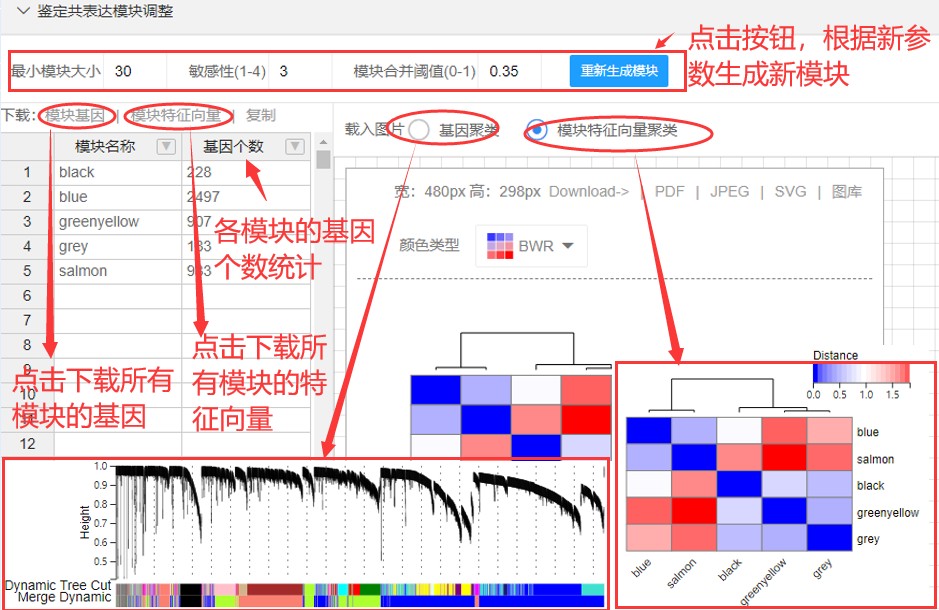 这个板块主要包含 模块特征下载,模块基因下载,模块中基因个数统计,模块基因聚类、模块聚类 及 模块鉴定的阈值设置并重新筛选模块,这几个部分中 需要提一下的是 模块鉴定的阈值筛选参数,共有三个:
这个板块主要包含 模块特征下载,模块基因下载,模块中基因个数统计,模块基因聚类、模块聚类 及 模块鉴定的阈值设置并重新筛选模块,这几个部分中 需要提一下的是 模块鉴定的阈值筛选参数,共有三个:
1、最小模块大小,这个值越大则 那些小的模块会被合并到大的模块中或者被剔除掉,最终 鉴定的模块个数越少
2、敏感性,这个值只能为1、2、3、4,四个值,值越大越敏感,最终鉴定的模块个数越多
3、模块合并的阈值,这个值为0-1之间的值,表示距离在该值内的合并到一个模块中,因此 值越大合并的范围也越大,更多的模块会被合并成一个模块,最终鉴定的模块个数越少
以上三个参数 没有金标准,所以 自己可以适当设置以确定 看似好的结果
第三个板块,临床特征与模块的相关性分析
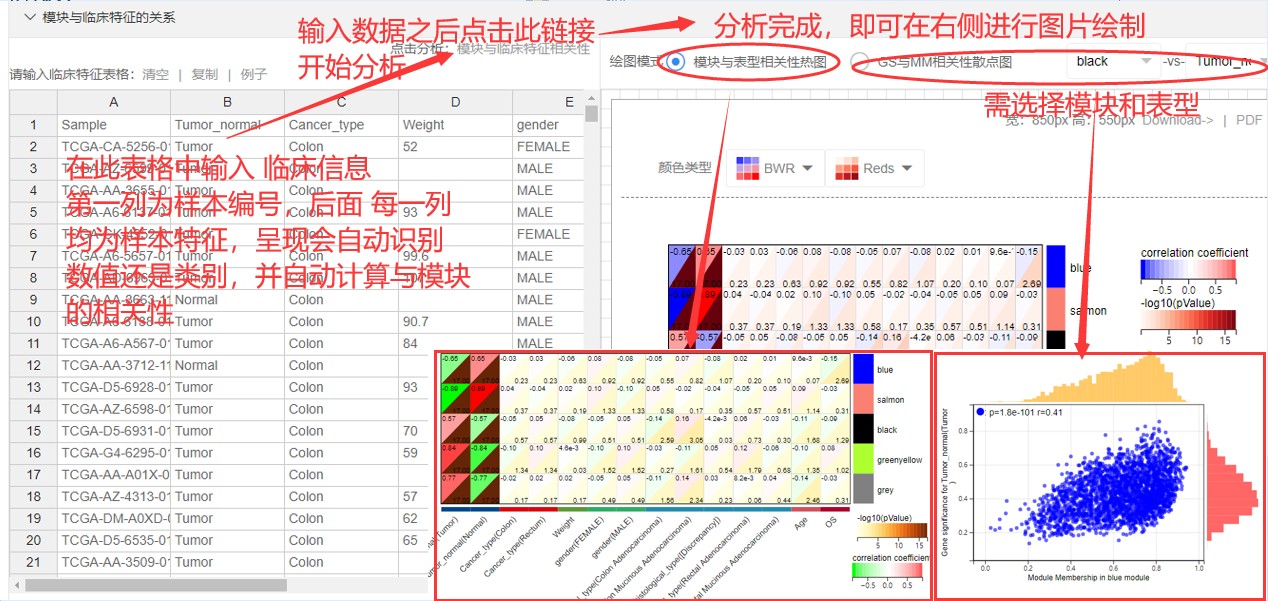
这部分 主要包含 临床特征 输入,模块与临床特征相关性分析 和 MM与GS相关性分析,值得注意的是 临床特征 不再先先前的几个软件版本中那样需要实现编制成0-1的矩阵了,这里 会自动根据 临床特征分类 进行编制成 数值矩阵,当然 自己也可以事先编制成数值矩阵 再复制到 这里的表格里
第四个板块,hub基因筛选和网络导出
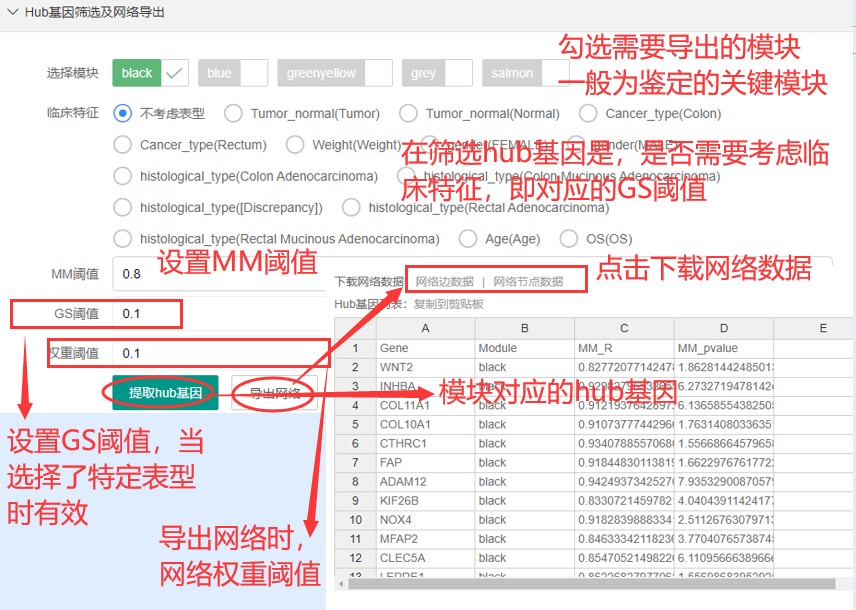
这个板块主要包含 hub基因筛选和导出模块的网络,没啥特别需要注意的,关键 是阈值的设定,不同阈值 会有不一样的结果
第五个板块 方法学写作及源文件下载

以上简单明了,写作部分 是动态生成的,但还是建议 自己再改一改,不要一昧的复制黏贴
- 发表于 2021-08-28 12:06
- 阅读 ( 16068 )
- 分类:软件工具
你可能感兴趣的文章
- 文献解析(8)这篇文章为研究癌症干细胞特性相关基因提供了一个很好的思路,一起来看看把! 6592 浏览
- 零代码系列之三教你如何进行功能富集 5981 浏览
- 零代码系列之三教你如何看懂WGCNA分析结果 20430 浏览
- 零代码系列之三教你如何做WGCNA数据准备 14058 浏览
- 用TCGA数据不用编程轻轻松松构建CeRNA网络 7705 浏览
