关于TCGA RNA-Seq数据ID的转换方法
使用TCGA简易下载工具的同志们一直很好奇,RNA-Seq数据集中的ID是怎么转换的,今天就介绍一下原理
首先我们根据TCGA官网提供的RNA-Seq pipline(https://docs.gdc.cancer.gov/Data/Bioinformat...
看教程不够直观,那就看视频吧! >>点击加载视频
使用TCGA简易下载工具的同志们一直很好奇,RNA-Seq数据集中的ID是怎么转换的,今天就介绍一下原理
首先我们根据TCGA官网提供的RNA-Seq pipline(https://docs.gdc.cancer.gov/Data/Bioinformatics_Pipelines/Expression_mRNA_Pipeline/)里面可以看出TCGA使用的gtf是从gencode上面下载的,如图:
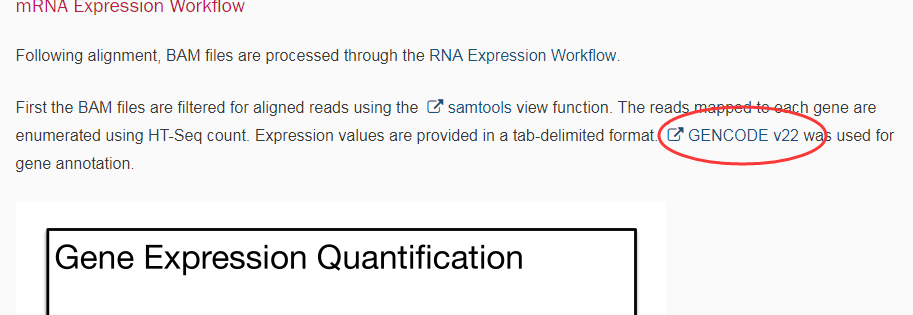
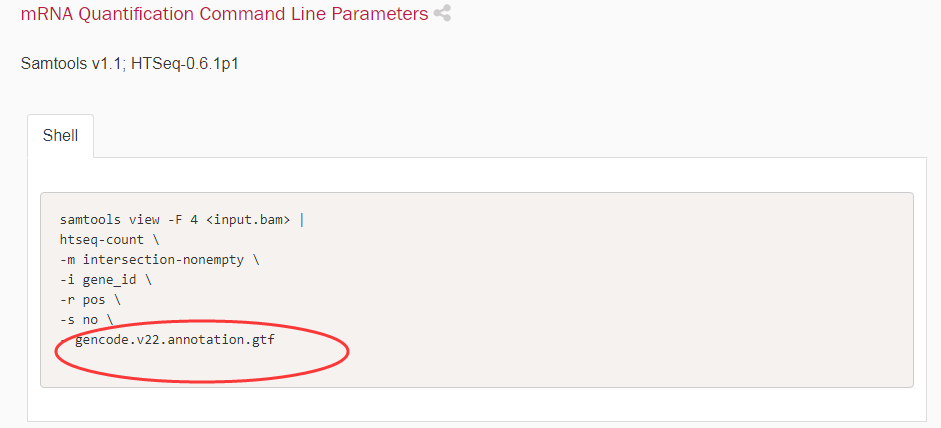 所以我们去GENCODE(http://www.gencodegenes.org/releases/22.html)上下载了对应的gtf文件,如图
所以我们去GENCODE(http://www.gencodegenes.org/releases/22.html)上下载了对应的gtf文件,如图
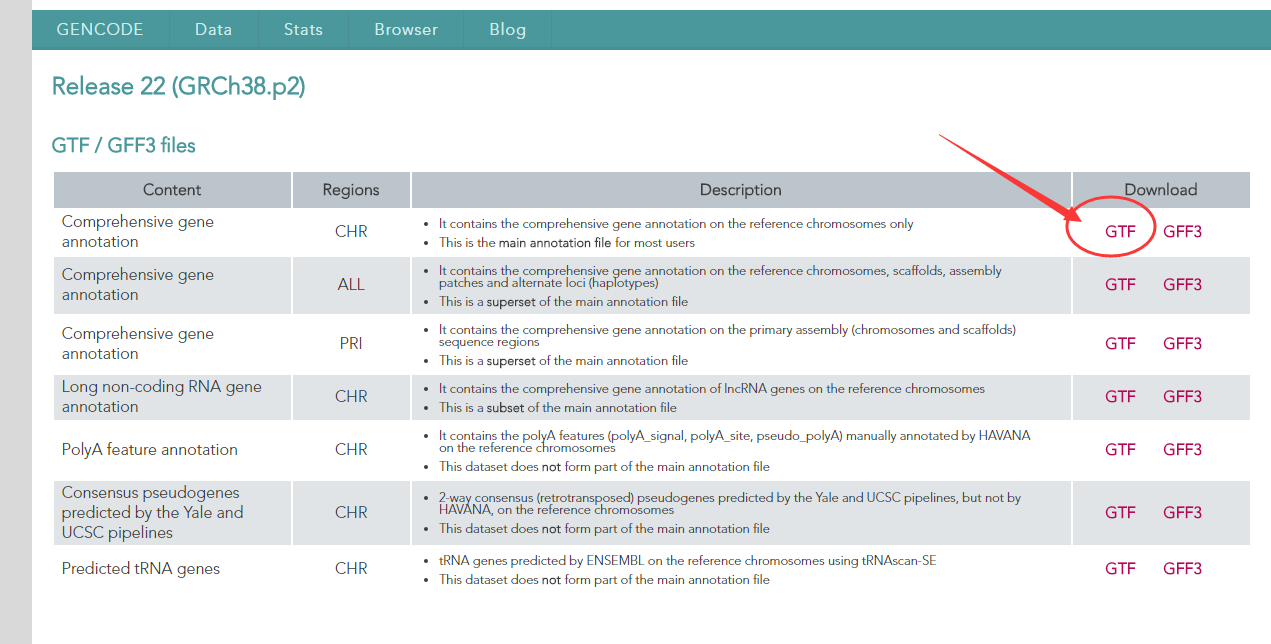 下载完之后是一个标准的gtf格式的数据,我们进一步解析这个数据,写一个简单的统计小脚本统计一下里面的基因个数
下载完之后是一个标准的gtf格式的数据,我们进一步解析这个数据,写一个简单的统计小脚本统计一下里面的基因个数
f=open('gencode.v22.annotation.gtf')
line=f.readline()
_list=[]
n=0
while line:
line=line.strip()
if line.find('#')!=0:
cols=line.split('\t')
_list.append(cols[2])
if cols[2]=='gene':
#print cols
n+=1
line=f.readline()
f.close()
print(set(_list))
print n
 可以看出里面的基因个数是60483,与TCGA里面下载的RNA-Seq中的ENSG编号个数一模一样
可以看出里面的基因个数是60483,与TCGA里面下载的RNA-Seq中的ENSG编号个数一模一样
打开里面的文件可以看到有基因的描述文件如下:

其中包含:
1、gene_id=ENSG 也就是TCGA RNA-Seq中使用的ID
2、gene_type 也就是用来描述这个基因的属性,一般为编码基因,miRNA,lncRNA,假基因等,我们可以根据这个信息提取lncRNA的表达谱
3、gene_name 也就是大家常见的gene_symbol
使用以上信息就可以进行TCGA的ID转换了
至于lncRNA的定义可以参考这里:https://www.shengxin.ren/question/23
最终提取各个属性的ID对应关系的代码如:
f=open('gencode.v22.annotation.gtf')
line=f.readline()
_list=[]
fw=open('GeneTag.txt','w')
fw.write('ENSGID\tSYMBOL\tTYPE\tLoc\tGENESTATUS\tGENETYPE\n')
while line:
line=line.strip()
if line.find('#')!=0:
cols=line.split('\t')
_list=[]
if cols[2]=='gene':
_ids=cols[8].split(';')
ensg=_ids[0][_ids[0].find('ENSG'):_ids[0].rfind('.')]
gene_type=_ids[1][_ids[1].find('"')+1:_ids[1].rfind('"')]
gene_status=_ids[2][_ids[2].find('"')+1:_ids[2].rfind('"')]
gene_name=_ids[3][_ids[3].find('"')+1:_ids[3].rfind('"')]
_list.append(ensg)
if gene_status=='NOVEL':
_list.append(ensg)
else:
_list.append(gene_name)
if gene_type=='processed_pseudogene' or gene_type=='unprocessed_pseudogene' or gene_type=='pseudogene' or gene_type=='unitary_pseudogene' or gene_type=='polymorphic_pseudogene':
_list.append('pseudogene')
elif gene_type=='lincRNA' or gene_type=='sense_intronic' or gene_type=='sense_overlapping' or gene_type=='antisense' or gene_type=='processed_transcript' or gene_type=='3prime_overlapping_ncrna':
_list.append('lncRNA')
elif gene_type=='protein_coding':
_list.append('protein_coding')
else:
_list.append('Other')
_list.append(cols[0]+":"+cols[3]+"-"+cols[4]+cols[6])
_list.append(gene_status)
_list.append(gene_type)
fw.write('\t'.join(_list)+'\n')
line=f.readline()
f.close()
fw.close()
引用:转录本属性定义http://vega.archive.ensembl.org/info/about/gene_and_transcript_types.html
- 发表于 2018-01-14 19:18
- 阅读 ( 19413 )
- 分类:方案研究
你可能感兴趣的文章
- 便捷ID转换器-V3使用教程 12706 浏览
- TCGA ID转换器使用案例 11865 浏览
- TCGA ID转换器使用说明 9191 浏览
相关问题
- RNA-Seq数据进行ID转化,许多基因转不出来,用以前的版本是可以做出来的,是版本的问题还是矩阵处理有问题 1 回答
- 生信工具盒打开数据转换一直提示网络连接异常,下载不了 2 回答
- 请问老师,我的基因ID转化后从MIMAT开头的变成了has-mir开头的,是转换成功了吗,后面这个就是基因名吗?以及我的火山图做不出来,无论是用Tumor-vs-Nomal.edgeR_Dif.txt的文件还是转换后的文件都不行(如图),两个都是文本格式txt,不知道哪里出问题了,希望老师们帮我看下,真的很感谢啊! 3 回答
- 老师,请问在使用便捷ID转换工具时,我已经关闭了工具盒子主界面,还是无法转换怎么办? 1 回答
- 请问生信小工具中的id转换工具中的id数据文件是在哪得到的? 1 回答
- TCGA简易数据库转换的时候,是ENSG_ID最后一位要加上1再去biomart转换?如:ENSG00000111224.12转换为gene名的时候将ENSG00000111224.12变为ENSG00000111224.13才转换,只有这样才能得到工具转换出来的结果,为什么加上1呢? 1 回答
6 条评论
请先 登录 后评论
