组装的贵人,原来是它们......
看教程不够直观,那就看视频吧! >>点击加载视频
在基因组研究中,组装可谓是万物之源。组装不好,后续的基因预测、功能注释等工作都没法保证准确性;发个文章吧,人家拿出一个组装比你好的基因组,一下子就能把你拍在沙滩上,于是你从此成为前任,小三上位,一时风光无两。。。
那么,怎么办呢?别担心,有三代、光学图谱、Hi-C来拯救!
所谓有备无患,开始组装基因组前,我们得先根据复杂程度,明白所研究的基因组是简单基因组or复杂基因组。简单基因组是指杂和率<1%,重复序列含量<50%,如鸟类、哺乳类;相对的,复杂基因组是指杂和率>1%,重复序列含量>50%。如何判断杂和率和重复序列含量?那就得做调研图分析啦,小编暂且不做赘述。
明白了原因,就得对症下药。一听简单基因组和复杂基因组就知道,组装的难度肯定是不相等的,因此需要设计不同的建库测序方案。基因组越复杂,测序深度当然越高越好啦。
进入正题——组装。先来看看当前基因组组装技术的发展概况:
我们先来简单介绍纯二代组装。首先,利用Allpath将二代短reads组装成contig,接着利用Sspace将contig搭建成scaffold,最后利用GapCloser补gap。至于具体的原理及参数设置,由于篇幅有限,小编还是那句话:“暂且不做赘述”~到此,经典的二代组装流程:contig→scaffold→补洞,就完成了!
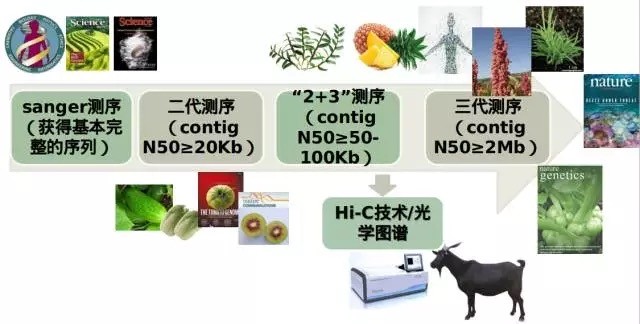
如果你并不满足于纯二代组装效果,我们推荐“2+3”组装模式。即在二代组装基因组的基础上,再利用三代长reads补洞,以弥补二代无法组装出重复序列和复杂区域的缺憾。这种方案使用的三代数据较少,较为经济实惠。苹果基因组可谓是典型代表:

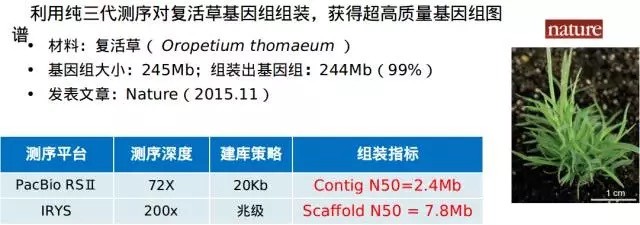
若你是不差钱的壕,不妨试试纯三代组装。对于高杂合度、高度重复区、异常GC含量区等组装难题,那都不是事儿!组装指标的提升简直分分钟刷新你的世界观~这也是目前大部分已发表文章的组装策略,举栗:
在组装重复区方面,还有一匹黑马,便是“光学图谱”,它比三代数据还!要!长此,一些连三代都无法跨越的重复区,找光学图谱就没错了。关于光学图谱的介绍,小编在之前的推送中已有所提及,详见文章“简单点,组装的方式简单点”。这里,小编就po出组装前后对比图,毕竟没有对比就没有伤害嘛:
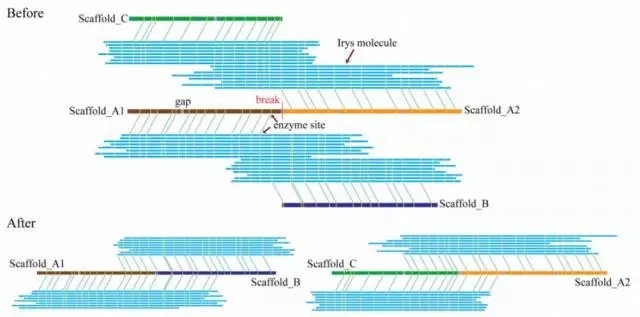
既然光学图谱那么牛,为什么还要用三代呢?两者当然是有区别的。三代主要作用于补gap,从而延长contig长度,最终提升contigN50;而光学图谱主要作用于连接scaffolds成为超长scaffold,致力于提升scaffoldN50。而他们的共同点就是打倒组装敌人——重复区。
也许scaffold级别的基因组已经无法满足你的需求了,那么我们还有Hi-C助你组装到染色体水平!现在已经证明Hi-C技术在人、小鼠、果蝇、拟南芥等基因组辅助组装中都取得了较好的效果: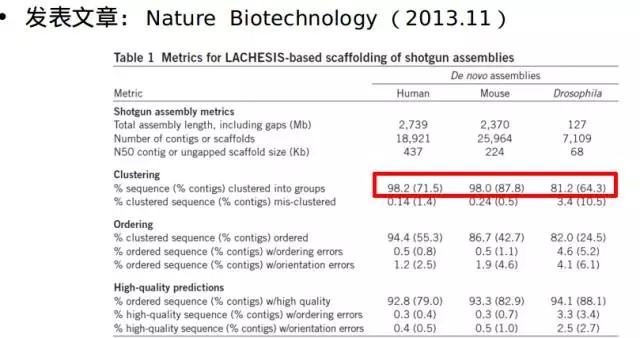
当然,最后的最后,我们还得从单碱基准确性、组装完整性、基因保守性等方面验证组装的准确性,进而展开新世界的探索。。。
- 发表于 2017-04-11 11:33
- 阅读 ( 5633 )
- 分类:基因组学
你可能感兴趣的文章
- 三维基因组常用分析工具汇总 10570 浏览
- 收藏|已公布的三代全长转录组数据 7573 浏览
- 测序简史 16565 浏览
- 畅想未来的测序 3288 浏览
- 2017年柑橘Nature Genetics文章解读 7166 浏览
