解析一篇三分多的甲基化文章:全基因组甲基化分析鉴定结肠腺癌中的新型预后甲基化标志物
看教程不够直观,那就看视频吧! >>点击加载视频
该文发表与2018年9月,二区,3.4分
 DNA甲基化是一种主要的表观遗传机制,是基因表达的重要调节因子,可以抑制转录因子的结合或抑制蛋白的募集。以前的研究表明,表观 遗传基因调控中的DNA 甲基化在正常发育和细胞功能中发挥着至关重要的作用,包括印记,X-失活和组织特异性基因表达。以前的研究也表明癌症和正常组织之间的DNA甲基化谱有显着差异。越来越多的关于癌细胞中DNA甲基化失调的报道表明,异常的甲基化变化导致不适当的基因表达,是大多数人类癌症发病机制的关键和早期事件,并有助于肿瘤发生。此外,它们可以很容易地在癌症患者的血浆或血清中检测到,从而突出了DNA甲基化作为癌症诊断和预后的新分子标记的潜力。
DNA甲基化是一种主要的表观遗传机制,是基因表达的重要调节因子,可以抑制转录因子的结合或抑制蛋白的募集。以前的研究表明,表观 遗传基因调控中的DNA 甲基化在正常发育和细胞功能中发挥着至关重要的作用,包括印记,X-失活和组织特异性基因表达。以前的研究也表明癌症和正常组织之间的DNA甲基化谱有显着差异。越来越多的关于癌细胞中DNA甲基化失调的报道表明,异常的甲基化变化导致不适当的基因表达,是大多数人类癌症发病机制的关键和早期事件,并有助于肿瘤发生。此外,它们可以很容易地在癌症患者的血浆或血清中检测到,从而突出了DNA甲基化作为癌症诊断和预后的新分子标记的潜力。
本研究是使用来自大量患者的高通量甲基化谱来研究COAD样品和邻近组织样品之间改变的DNA甲基化模式,确定特定的DNA甲基化位点作为潜在的生物标志物,具有预测COAD患者总体生存的能力。
目的很明确:使用结肠腺癌样本的甲基化数据筛选差异DNA甲基化并进一步挖掘DNA甲基化预后标志物。
数据来源是什么?
使用Illumina Human Methylation 450k Array 产生的353个样品(315个COAD样品和38个匹配的肿瘤相邻正常组织样品)的DNA甲基化数据获自The Cancer Genome Atlas(TCGA,https ://cancergenome.nih.gov/)。使用Illumina HiSeq产生的329个COAD样品的RNA-Seq基因表达数据来自UCSC Xena(https://xenabrowser.net/datapages/?dataset=TCGA.COAD.sampleMap/HiSeqV2&host=https://tcga.xenahubs.net)。从GDC数据门户(https://portal.gdc.cancer.gov/)获得COAD样品的临床信息,包括肿瘤分期,存活状态和时间。
数据预处理及差异甲基化区域筛选(minfi包搞定):
TGCA获得485,577个基因座的DNA甲基化数据。在预处理数据和质量控制后,保留了467,971个探针用于进一步分析,使用minfi包对DNA甲基化数据进行预处理和标准化,并对阵列标准化中的子集分位数进行探针过滤,色偏校正和背景减法,以及子集分位数归一化。使用minfi包鉴定COAD样品和相邻组织样品之间的差异甲基化区域(DMR)。使用映射到DMR的多个CpG探针的平均甲基化水平估计DMR的甲基化水平。
具体minfi参数为:
resamples = 100, cut off = 0.25, length = 200 bp and probe number ≥5
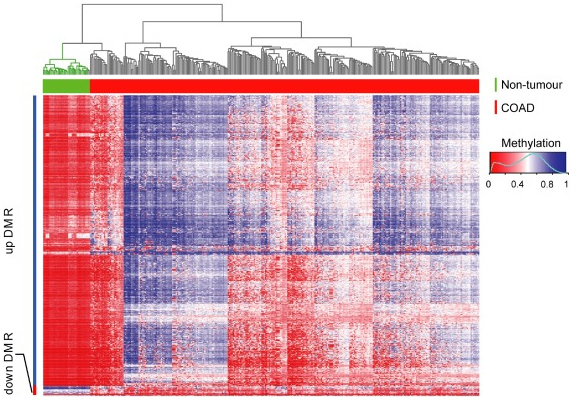
最终得到675个差异甲基化区域,其中654个上调。
差异甲基化区域的基因组注释:
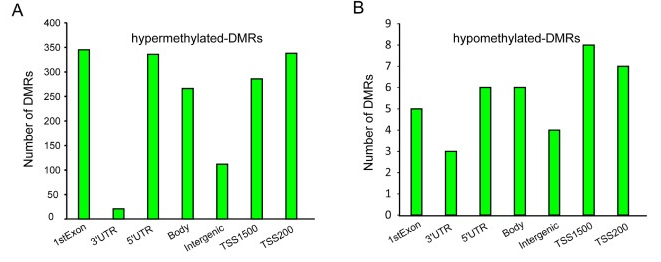
我们筛选得到了基因组上的差异甲基化区域,那么這些区域与基因的关系是什么呢?我们利用這些差异甲基化区域的位置与基因的各个元件位置的关系,观察這些差异甲基化区域主要分布在基因的哪些位置上,如图,从中可以看出上调的甲基化区域大多数位于基因的第一外显子,5'UTR,TSS200,TSS150和基因体中,而只有少数UMR位于基因间和3'UTR中区域,同样的下调的甲基化区域也有相同的现象。
 进一步的我们观察這些差异甲基化区域与CpG岛的关系如图,从中可以看出上调的差异甲基化区域主要聚集在CpG岛区域,而下调的差异甲基化区域主要聚集在低CpG岛密度区域。
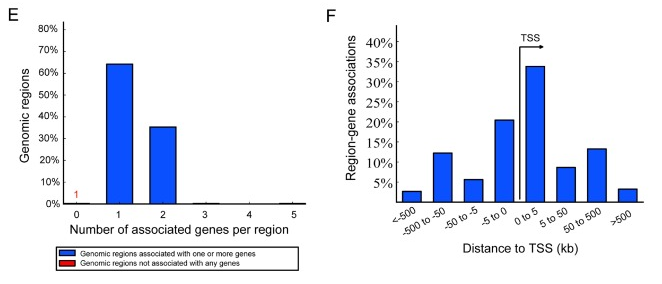
进一步的我们观察這些差异甲基化区域与CpG岛的关系如图,从中可以看出上调的差异甲基化区域主要聚集在CpG岛区域,而下调的差异甲基化区域主要聚集在低CpG岛密度区域。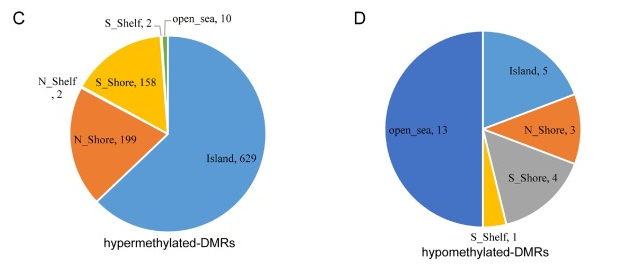 再观察這些差异甲基化区域与转录起始位点的位置关系如图,从左图中可以看出大部分差异甲基化区域只与一个基因相关,少部分与两个基因相关,分析這些差异甲基化区域与转录起始位点的位置关系如右图,大部都在转录起始位点附近。
再观察這些差异甲基化区域与转录起始位点的位置关系如图,从左图中可以看出大部分差异甲基化区域只与一个基因相关,少部分与两个基因相关,分析這些差异甲基化区域与转录起始位点的位置关系如右图,大部都在转录起始位点附近。
 为了更好的分析差异甲基化区域的功能,我们根据与差异甲基化区域存在关联的基因作为目标基因集,用這些基因的功能来代表這些差异甲基化区域的功能,使用R软件包clusterProfiler进行GO功能富集分析,使用在线工具KOBAS 进行KEGG富集分析,结果如图,很常规的富集分析了。
为了更好的分析差异甲基化区域的功能,我们根据与差异甲基化区域存在关联的基因作为目标基因集,用這些基因的功能来代表這些差异甲基化区域的功能,使用R软件包clusterProfiler进行GO功能富集分析,使用在线工具KOBAS 进行KEGG富集分析,结果如图,很常规的富集分析了。
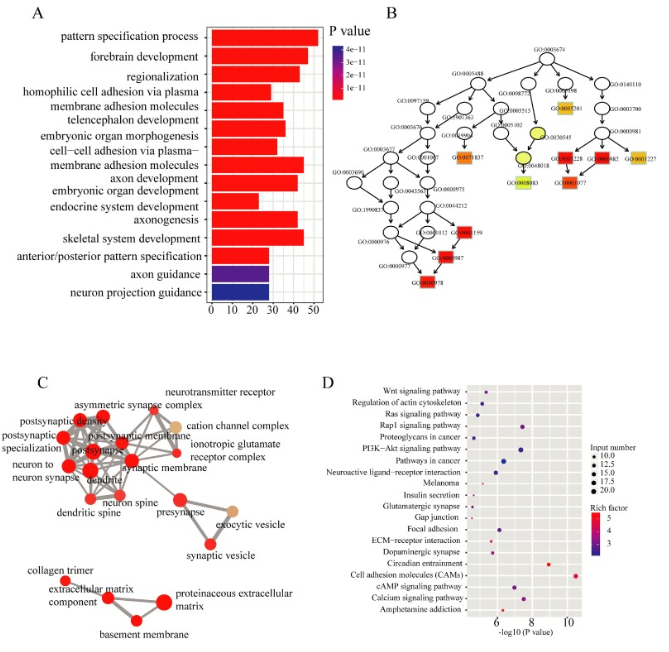
以上便是整个差异甲基化区域的筛选和功能分析,下一步进行预后标志物筛选首先进行单因素生存分析。
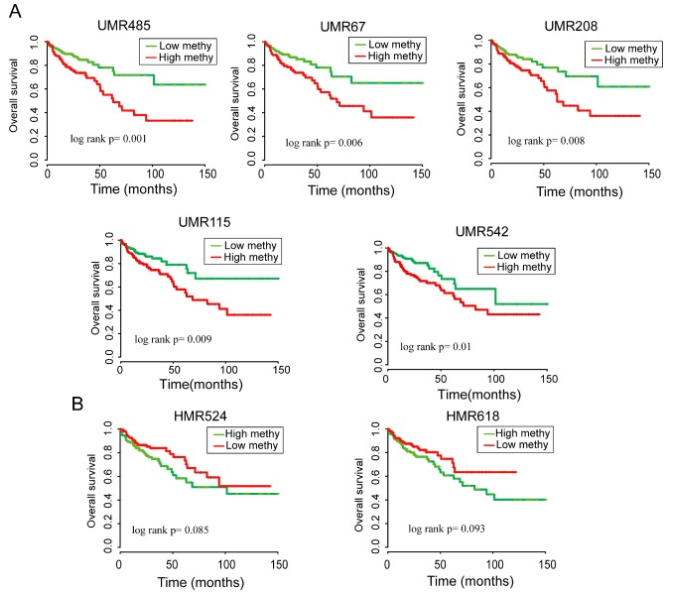
我们根据每个差异甲基化区域中甲基化位点的甲基化水平分别以中位数作为改差异甲基化区域的甲基化水平,进一步根据差异甲基化区域在各个样本中的甲基化水平的中位数对样本进行划分,高甲基化组和低甲基化组,然后结合病人的预后信息,使用Kaplan-Meier方法分析两组之间的预后差异,最终筛选出有预后差异的差异甲基化区域
共得到了7个预后差异的差异甲基化区域,如图。
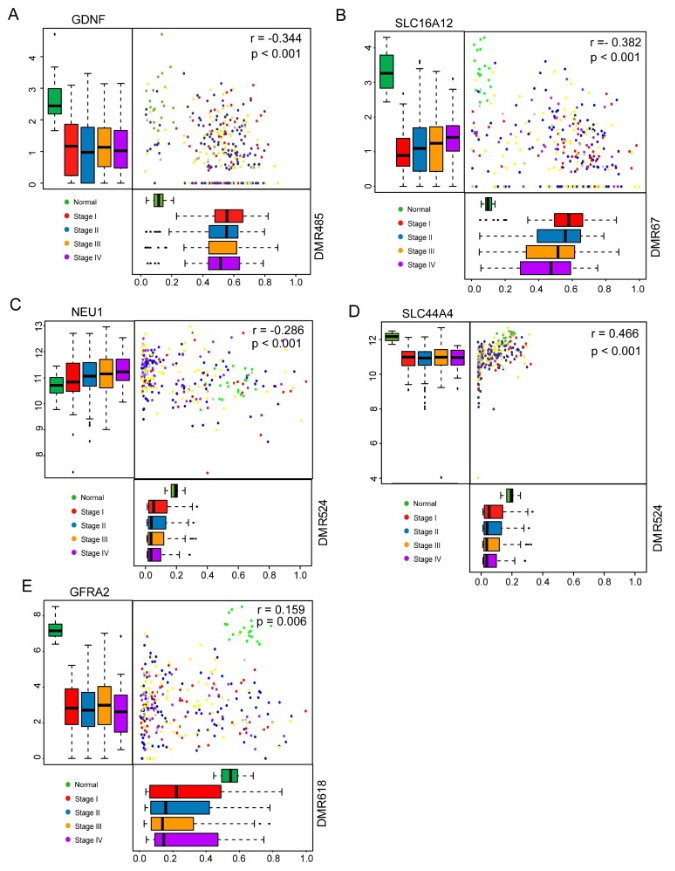
进一步分析预后差异的差异甲基化区域与基因表达的关系我们根据先前的报道,选择差异甲基化区域的100kb以内的转录起始位点对应的基因,分别计算差异甲基化区域的甲基化水平与這些基因的表达水平的皮尔森相关系数,选择p<0.05作为阈值,筛选与预后差异的差异甲基化区域显著表达相关的基因。
共有五个预后差异的差异甲基化区域有显著表达相关的基因,如图,這些区域中有三个时负相关,两个正相关,进一步分别分析了差异甲基化区域的甲基化水平、基因表达水平在Normal和四个Stage的表达分布,从中明显可以看出五个状态下表达水平具有明显的差别。
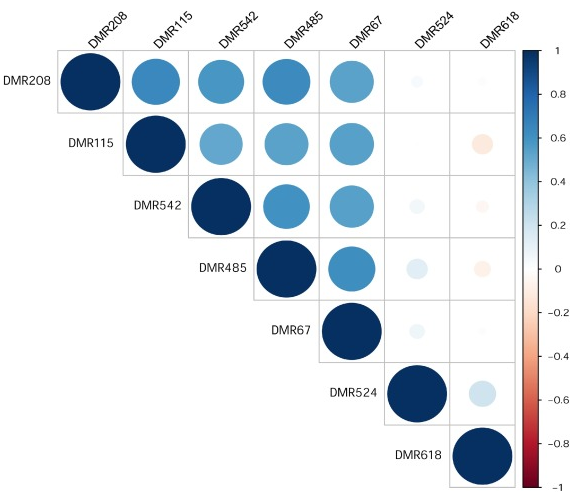
七个预后差异的差异甲基化区域的甲基化水平的相关性为了观察這些预后差异的差异甲基化区域的甲基化水平之间是否存在联系,我们分别分析了这七个预后差异的差异甲基化区域的甲基化水平的相关性如图,从中可以看出5个UMR之间存在强烈的正相关性,然而,2个HMR和5个UMR之间没有明确的相关性
在本研究中,在大量COAD样品中进行了DNA甲基化谱的综合分析,以研究COAD中存在的改变的DNA甲基化模式。COAD样品和邻近组织样品之间的DNA甲基化谱的比较揭示了COAD样品中异常的DNA甲基化变化,并导致675个DMR的鉴定,包括654个高甲基化和21个低甲基化DMR。这些结果与先前的研究结果一致,即DNA高甲基化是结直肠癌的常见特征。此外,这些DMR可用于有效区分COAD样品和相邻组织样品,这表明DMR可能在COAD的形成中具有致病作用。基因组分析显示,DMR主要位于启动子区域(包括第1 外显子,5'UTR和TSS)和体区,这与之前在其他类型癌症中的观察结果一致。在基因间和 3'UTR 区域中仅发现了一小部分DMR。此外,大多数高甲基化DMR位于CpG岛中,而大多数低甲基化DMR不位于CpG岛或注释基因中。
- 发表于 2019-02-18 13:27
- 阅读 ( 10281 )
- 分类:方案研究
你可能感兴趣的文章
- 建模的精髓:可靠性>方法 3862 浏览
- 文章分享(三)iCCA的亚型与基因组、表观组、预后以及细胞来源的差异的关系 4685 浏览
- 节后第一篇--如何用生信去挖掘新型甲基化预后标记 4923 浏览
- 基因表达与甲基化表达谱纯生信分析4分文章,你也可以 7167 浏览
- 这篇五分多的signature建模文章有点机智啊 11235 浏览
相关问题
- 从GEO数据库下载下来的甲基化芯片要什么处理呢?下载来的芯片,通过R语言处理后得到了甲基化差异区域,通过注释获取了相关基因。但是我不明白什么这些区域哪些是低表达还是高表达的,对于基因的影响,希望大佬们能帮忙一下,好几天了很绝望啊 3 回答
- GSE数据 甲基化分析 0 回答
- 得到的甲基化差异基因有一万多怎么找到比较好的做富集分析 1 回答
- 请问在TCGA下载的miRNA数据中hsa-mir-21怎么转换为相应的基因名称?还有甲基化的数据类似cg00003578怎么转换? 1 回答
- TCGA胃癌甲基化数据下载很长时间下载进度是0,是什么原因呢? 1 回答
- 甲基化的合并文件,只合并了每个样本的beta value,可否合并TSS position 1 回答
