R语言做主成分分析实例
主成分分析法是数据挖掘中常用的一种降维算法,是Pearson在1901年提出的,再后来由hotelling在1933年加以发展提出的一种多变量的统计方法,其最主要的用途在于“降维”,通过析取主成分显出的最大的个别差异,也可以用来削减回归分析和聚类分析中变量的数目,与因子分析类似。
看教程不够直观,那就看视频吧! >>点击加载视频
在做多样本的RNA-Seq数据中经常会用到主成分分析(PCA)来分析,那么什么是PCA呢,这个可以百度一下,大概意思如下:
主成分分析法是数据挖掘中常用的一种降维算法,是Pearson在1901年提出的,再后来由hotelling在1933年加以发展提出的一种多变量的统计方法,其最主要的用途在于“降维”,通过析取主成分显出的最大的个别差异,也可以用来削减回归分析和聚类分析中变量的数目,与因子分析类似。
比如你要做一项分析人的肥胖的因素有哪些,这时你设计了50个你觉得都很重要的指标,然而这50个指标对于你的分析确实太过繁杂,这时你就可以采用主成分分析的方法进行降维。50个指标之间会有这样那样的联系,相互之间会有影响,通过主成分分析后,得到三五个主成分指标。此时,这几个主成分指标既涵盖了你50个指标中的绝大部分信息,这让你的分析得到了简化(从50维降到3、5维)。
今天就用一个实例来讲解一下在R语言中如何实现PCA分析:
数据准备:
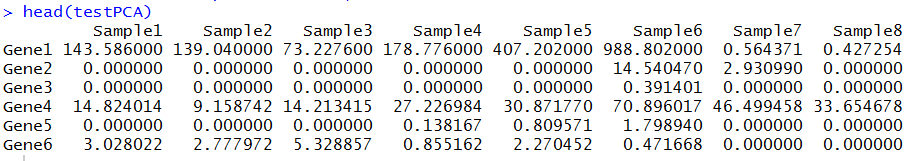
一个表达矩阵:testPCA,行为基因,列为样本
我们使用princomp()函数来做主成分分析,使用的格式为:
princomp(formula,data = NULL,subset,na.action,...)
其中formula是没有响应变量的公式,类似于回归分析和方差分析中但是没有响应的变量.data是数据框,类似于回归分析和方差分析.
使用代码如下:
head(testPCA)
pca1 <- princomp(testPCA,cor = T)
summary(pca1,loadings=T)

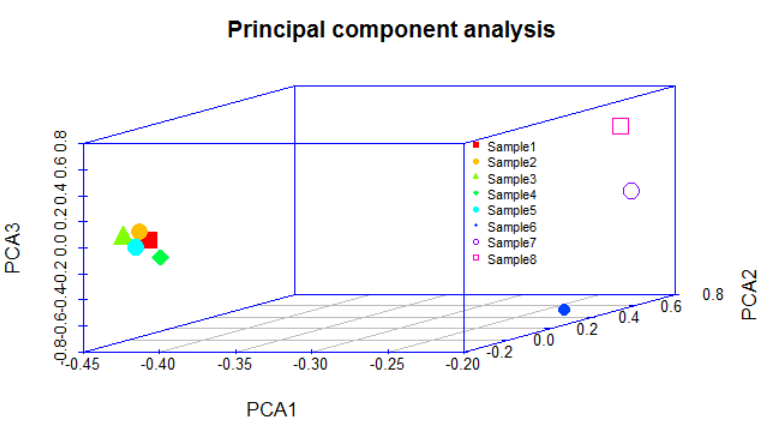
从PCA结果中可以看出,前4个变量Comp.1,Comp.2,Comp.3已经贡献了89.2%的信息,Loadings中的矩阵分别对应主成分与各样本之间的系数关系,我们选择前三个主成分进行后续可视化,观察八个样本之间的关系。
library(scatterplot3d)
PCA1=pca1$loadings[,1]
PCA2=pca1$loadings[,2]
PCA3=pca1$loadings[,3]
colors=rainbow(24)
s3d=scatterplot3d(PCA1,PCA2,PCA3
#, highlight.3d = TRUE
, col.axis = "blue",angle = 40,
color=colors[seq(1,24,3)], main = "Principal component analysis", pch = ' ')
s3d$points(PCA1,PCA2, PCA3, pch =15:22,
cex = 2,col=colors[seq(1,24,3)])
legend(s3d$xyz.convert(-0.2, -0.2, -0.2), pch = 15:22, yjust=0,
legend =colnames(testPCA), cex = .7,col=colors[seq(1,24,3)],bty="n")
PS:使用princomp,当列数超过行时就会报错:'princomp' can only be used with more units than variables
此时使用prcomp可避免此问题,如下:
pca1<-prcomp(testPCA)
PCA1=pca1$rotation[,1]
PCA2=pca1$rotation[,2]
- 发表于 2017-06-01 12:37
- 阅读 ( 14863 )
- 分类:编程语言
你可能感兴趣的文章
10 条评论
请先 登录 后评论