文献解析(4)这篇研究确定了一个高效的DNA损伤修复相关风险信号作为预测低度胶质瘤预后的指标!
看教程不够直观,那就看视频吧! >>点击加载视频


胶质瘤是颅内最常见的恶性肿瘤。标准的治疗方法是手术切除加放疗和化疗。然而,由于DNA损伤修复导致的耐辐射和耐药的出现,限制了治疗效果。同时根据2016年WHO中枢神经系统肿瘤分类,将分子标志物与组织学特征相结合,完善了胶质瘤分类,并且提高了诊断准确性,指导个体化治疗。此外,分子分类的应用推动了靶向治疗的发展,目前已有PDGFR抑制剂、AKT抑制剂、IDH1抑制剂等多种方法用于临床试验。同时随着抗PD-1或PD-L1单克隆抗体在黑色素瘤和非小细胞肺癌治疗中的成功,免疫治疗被认为是一种有希望的胶质瘤治疗方法。然而尽管人们为提高胶质瘤的治疗效果做了大量的努力,但仍未获得实质性的生存改善。
今天小编推荐的这篇研究确定了一个高效的DNA损伤修复相关风险信号作为预测低度胶质瘤预后的指标。
标题:Transcriptomic profiling identifies a DNA repair-related signature as a novel prognostic marker in lower-grade gliomas。 杂志:Cancer Epidemiology Biomarkers Prevention IF:4.142 发表时间:2019.9.18

① 数据:训练集:来自CGGA的172个低级胶质瘤的RNA-seq数据及相关的临床数据。
验证集:来自TCGA的451个胶质瘤样本的RNA-seq和芯片数据以及临床信息
② 生物信息学分析方法:作者在CGGA数据集中对先前研究发现的513个DNA修复相关的基因使用单因素cox风险比例回归模型筛选出266预后基因。进一步使用LASSO开发出一个风险得分:
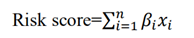
根据风险得分将患者分成高低风险组,进一步分析预后差异。同时使用Cox回归模型以及时间ROC曲线评估模型的分析效能,同时对模型中的基因进行通路以及功能富集分析。
③ 构建以及验证预测模型:为了预测患者的总体生存,整合分析患者的风险得分以及临床因素建立一个nomogram。在训练集以及验证集中对其进行评估。
④ 免疫组化:为了分析预后基因的功能,作者进行了免疫组化分析,对细胞周期蛋白A2以及FANCD2进行染色分析。
 1.预后signature的构建:
1.预后signature的构建:
作者首先在CGGA数据集中对先前研究发现的513个DNA修复相关的基因使用单因素cox风险比例回归模型筛选出266个预后基因。对这些基因进一步用LASSO进行筛选。最终得到一个风险得分预后模型,对模型中的风险基因经行评估,评估其与风险得分以及患者生存的关系(图1)。同时将这些发现在验证集中进行验证(图2)
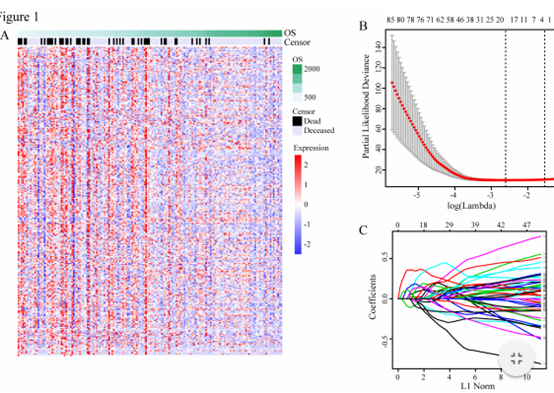 图1
图1
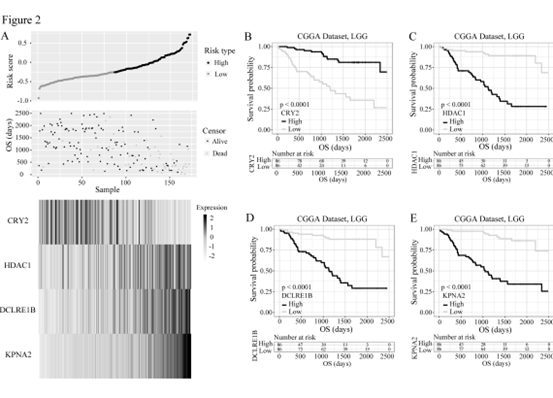 图2
图2
2.对风险signature的分子刻画以及风险特征:
作者首先在两个数据集中研究了风险得分以及临床特征之间的关系(图3A)。同时对高低风险组的患者进行生存分析(图3B),在分过程中考虑到了IDH突变以及1p/19q的情况(图3C-E),同时对风险得分的AUC进行评估。
 图3
图3
3.风险得分的预后效能:
为了验证风险得分的价值,作者对CGGA以及TCGA两套数据进行了cox回归分析,单因素cox展示在图4A,而图4B则展示了多因素cox分析的结果,可以看出不管是单因素cox还是多因素cox风险得分都与生存显著相关,接下来作者将风险得分与IDH突变相结合进行研究(图4C)。并在验证集中进行同样分析。
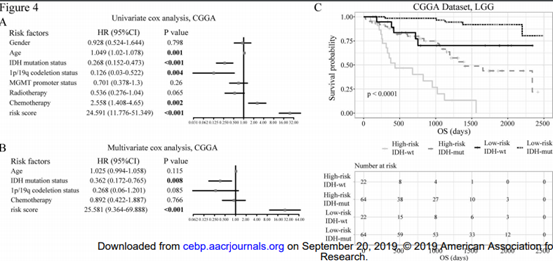 图4
图4
4 构建一个独立的预后模型:
对患者的全局生存相关的独立预后因素进行选择以及整合得到nomogram模型(图5A),并对模型在验证集中进行了评估,如图5B,C所示。
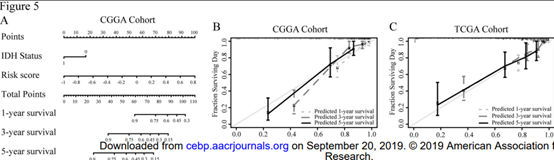 图5
图5
5.功能注释以及通路富集:
为了探索识别出的和风险得分相助相关的基因的潜在功能,使用Metascape对其进行功能注释,功能富集结果如图6所示,结果表明这些相关基因主要参与与细胞周期,DNA修复相关的通路,参与肿瘤的发生发展。同时这些结果在TCGA数据集中得到了验证
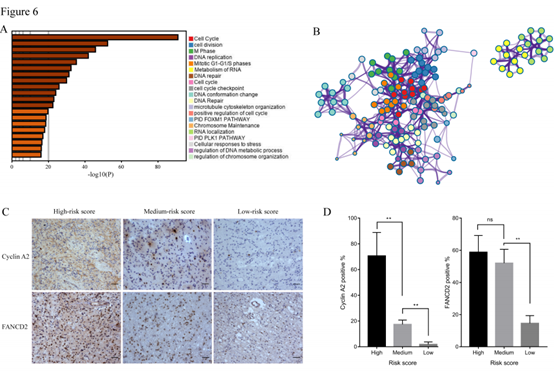 图6
图6
 该signature可区分患者的预后,高风险得分患者生存时间短。时间依赖的ROC曲线、Cox回归和Nomogram模型显示生存signature具有独立的预后效果和较高的预后准确性。结合IDH突变状态,这种风险特征可以进一步细分生存期不同的患者。相关基因的功能分析揭示了与signature相关的基因参与细胞周期和DNA修复的生物学过程,同时这些机制已在患者样本中得到证实。
该signature可区分患者的预后,高风险得分患者生存时间短。时间依赖的ROC曲线、Cox回归和Nomogram模型显示生存signature具有独立的预后效果和较高的预后准确性。结合IDH突变状态,这种风险特征可以进一步细分生存期不同的患者。相关基因的功能分析揭示了与signature相关的基因参与细胞周期和DNA修复的生物学过程,同时这些机制已在患者样本中得到证实。

1. Nuno M, Birch K, Mukherjee D, Sarmiento JM, Black KL, Patil CG. Survival
and prognostic factors of anaplastic gliomas. Neurosurgery 2013;73(3):458-65;
quiz 65 doi 10.1227/01.neu.0000431477.02408.5e.
2. Jiang T, Mao Y, Ma W, Mao Q, You Y, Yang X, et al. CGCG clinical practice
guidelines for the management of adult diffuse gliomas. Cancer letters
2016;375(2):263-73 doi 10.1016/j.canlet.2016.01.024.
3. Guan X, Vengoechea J, Zheng S, Sloan AE, Chen Y, Brat DJ, et al. Molecular
subtypes of glioblastoma are relevant to lower grade glioma. PloS one
2014;9(3):e91216 doi 10.1371/journal.pone.0091216.
4. Wesseling P, Capper D. WHO 2016 Classification of gliomas. Neuropathology
and applied neurobiology 2018;44(2):139-50 doi 10.1111/nan.12432.
5. Louis DN, Perry A, Reifenberger G, von Deimling A, Figarella-Branger D,
Cavenee WK, et al. The 2016 World Health Organization Classification of
Tumors of the Central Nervous System: a summary. Acta neuropathologica
2016;131(6):803-20 doi 10.1007/s00401-016-1545-1.
6. Gusyatiner O, Hegi ME. Glioma epigenetics: From subclassification to novel
treatment options. Seminars in cancer biology 2018;51:50-8 doi
10.1016/j.semcancer.2017.11.010.

文献解析(1)如何构建关系网络来更好的揭示一些相关的生物学机制?
文献解析(2)影响EGC病人使用ICI反应的关键因子是什么?
文献解析(3)是什么基因分类器竟能应用到头颈癌病人预后以及靶向治疗的药物反应评估中?
文献解析(5)表观遗传时钟与共识分子亚型和结直肠癌总体生存率的关系
- 发表于 2019-11-26 13:32
- 阅读 ( 4925 )
- 分类:文献解读
