文献解读(4)预测前列腺癌患者预后的新型分层框架
看教程不够直观,那就看视频吧! >>点击加载视频

无监督学习方法通常用于分析基因组平台数据。然而这些方法忽略了前列腺样本中的大量的异质成分。那么这篇文章的研究的目的就是使用更复杂的分析方法对前列腺癌转录组数据的结构进行反卷积,为该疾病提供新颖的临床可行信息。其发表在British Journal of Cancer上,影响因子5.416。

让我们先来通过摘要了解下这篇文章的主要内容,在这里作者将一个称为潜在过程分解(LPD)的无监督模型应用于8个前列腺癌临床系列(包括1785个以PSA失败和转移为临床终点的恶性肿瘤样本)的全基因组表达数据,该模型可以处理单个癌样中的异质性。结果显示PSA(前列腺癌生物标志物)失败与称为DESNT的表达signaure的水平相关,并且具有多数DESNT signature的患者转移风险增加。此外,作者开发了一种结合DESNT并识别出前列腺癌的三种新型分子亚型的分层框架。

1)转录组数据集
作者使用了八个公开的转录组芯片数据集,这些数据集来自前列腺癌男性前列腺切除术样本,并被称为MSKCC,CancerMap,CamCap,Stephenson,TCGA,Klein,Erho和Karnes。这其中包括原发性恶性组织标本1785份,正常组织标本173份(表1)。
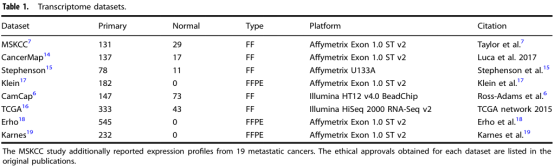
表1.转录组数据集
2)潜在过程分解
LPD 是一种无监督的贝叶斯方法,可将每个样本分解为组成子元素(signature)。每个特征是代表性的基因表达模式。LPD能够根据每个样本中特征的相对表示对复杂数据进行分类。LPD可以评估最可能的特征数,作者评估了在各种特征数量下计算出的数据的保留验证对数似然性,并使用统一和非统一先验的组合来选择特征的数量。作为输入,将每个数据集简化为探针,以检测MSKCC数据集中差异最大的500个基因。为了提高鲁棒性,对于每个数据集,LPD使用不同的种子运行100次。在100次运行中,作者选择生存对数质检验p值最接近模式的运行作为用于后续分析的代表性运行。
3)OAS-LPD
OAS-LPD(一个添加的样本-LPD)算法是LPD算法的修改版本,其中新样本被分解为LPD 特征,而无需重新训练模型。该算法主要用于对所有数据集的附加表达谱进行分类,一次一个样本。
4)统计检验
使用R进行所有的统计检验。为了表征样本,每个样本都被分配给该样本具有最大(γ)值的特征。
5)相关性
针对八个特征中的每一个,作者计算了MSKCC和CancerMap之间的表达谱之间的Pearson相关性:
(i)对于每个基因,随机选择一个相应的探针;
(ii)对于每种探针,将其在所有样本中的分布转换为标准正态分布;
(iii)在每个数据集中,测定分配给特征 j(基因亚群平均值)的样本中每个基因的平均表达;
(iv)计算每个特征的MSKCC基因亚组平均表达谱与CancerMap基因亚组平均表达谱之间的皮尔森相关性。
6)差异表达和甲基化特征
通过使用limma R包中的t检验(至少50/100次差异表达)来识别每个特征的差异表达探针集。使用methylMix R软件包, 并使用发现在该signature中差异表达的基因作为输入来鉴定预测转录的低甲基和高甲基化基因。
7)生存分析和列线图
使用Cox比例风险模型,对数秩检验和Kaplan–Meier进行生存分析,并以前列腺切除术后的生化复发为终点。对于列线图的构建,将Cox比例风险模型拟合到通过组合MSKCC,CancerMap和Stephenson数据集而获得的元数据集,并使用rms R软件包在CamCap上进行了验证。
8)检测基因组特征的过表达
在样本水平上检查了由癌症基因组图谱研究网络鉴定出的突变癌症基因。使用χ2检验确定分配给特定LPD signature的样本中这些特征的不足/过度表示。
9)通路过表达分析和signature相关性分析
使用clusterProfiler测试了GO生物学过程注释中每个signature中差异表达基因列表中的过表达(低表达)。使用全部8个数据集的完整组合数据集,计算17697个MSigDB v6.0基因集每个样本的Z分数。这些与DESNTγ值相关,并选择了绝对pearson相关性最高的前20组。使用FDR对来自通路过表达分析的结果p值进行了调整。

1. DESNT signature作为连续变量的存在与不良的临床预后相关
在之前的研究中,作者在前列腺癌切除术后收集的表达微阵列数据集中,LPD被检测到3到8个潜在signatures(也称为过程)。MSKCC数据集的分解给出了八个signatures。图1a说明了在每个MSKCC样本中鉴定出的DESNT表达signature的比例,如图1a和图1c所示,当DESNT signature最丰富时,各个癌症样本被指定为“ DESNT癌症”。并且相对于同一数据集中的其他癌症样本,DESNT癌症患者总是表现出较差的结果。这意味着存在包含DESNT siganture的癌症区域,会导致不良预后。接下来作者预测了包含较小的DESNT signature贡献的癌症样本,例如图1b所示的MSKCC数据集,显示较差的结果。
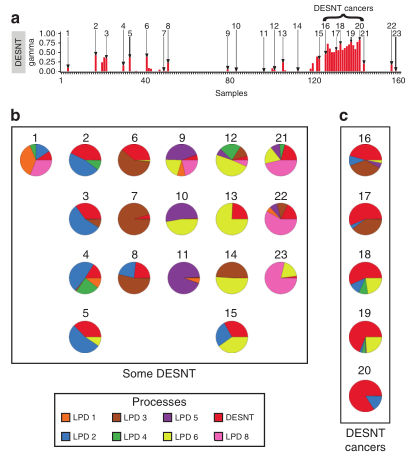
图1. MSKCC数据集的LPD分解
为了提高检验该预测的能力,作者结合了MSKCC,CancerMap,Stephenson和CamCap研究(n = 503)的转录组数据。当分配给DESNT signature的表达比例被视为连续变量时,与PSA复发存在显著关联。随着DESNT signature比例的增加,结果变得更糟。为了说明,作者根据DESNT的比例将癌症样本分为四类,其中47.4%的癌症样品至少含有一些DESNT癌症(图2a)。60个月时PSA无故障生存率分别为82.5%、67.4%、59.5%和44.9%,DESNT signature的比例分别为:<0.001;0.001-0.3;0.3-0.6;和>0.6(图2b)。
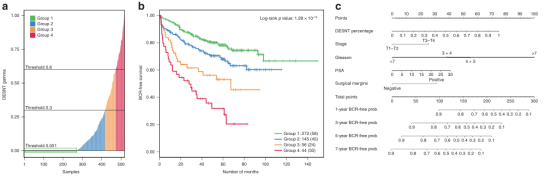
图2. 根据前列腺癌的百分比分层
2. 构建列线图
在Cox比例风险模型中将DESNT癌症的比例与其他临床变量合并,并拟合到318个癌症样本(MSKCC,CancerMap和Stephenson)的合并数据集中;CamCap癌症样本(n = 185)用于外部验证。结果显示DESNT的比例是临床预后恶化的独立预测因子。PSA水平和病理分期均低于统计显著性阈值。在内部验证时,Cox模型得到了1000个bootstrap修正的C指数,为0.747;在外部验证时,C指数为0.795。使用该模型,构造了列线图,以结合DESNT癌症信息和临床变量来预测前列腺切除术后1、3、5和7年生化复发的风险(图2c)。
3. 前列腺癌的新类别
在这一部分,作者希望确定LPD signatures是否以特定的临床或分子特征为特征,表明它们代表了前列腺癌的不同类别。使用源自MSKCC的基因signature模型的OAS-LPD应用于所有数据集(表1),并且每个样本都分配了最丰富的signature。与其他组相比,来自非癌性(良性)前列腺组织的样本分配给LPD2,LPD4和LPD8的频率更高。当合并具有临床数据的数据集(MSKCC,CancerMap,Stephenson和CamCap,图3a–c)时,分配给DESNT的原发性癌症的预后较差,而分配给LPD4的原发性癌症的PSA失败可改善预后。
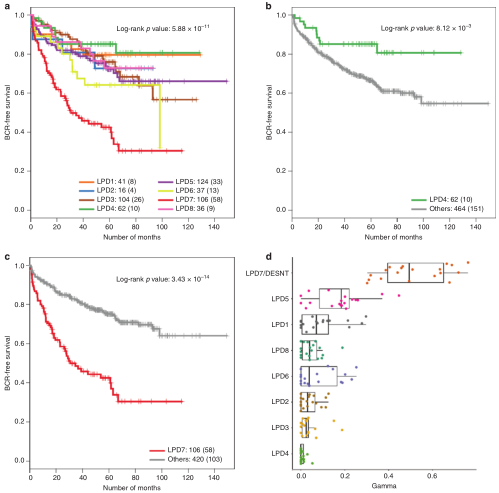 图3. 根据OAS-LPD组对临床结果的预测
图3. 根据OAS-LPD组对临床结果的预测
在ERG(前列腺癌生物标志物)状态可用的所有三个数据集中,具有ERG改变并分配给标志性LPD3的癌症样本也表现出更好的预后(与所有其他ETS阳性癌症样本相比)(图4b-d)。
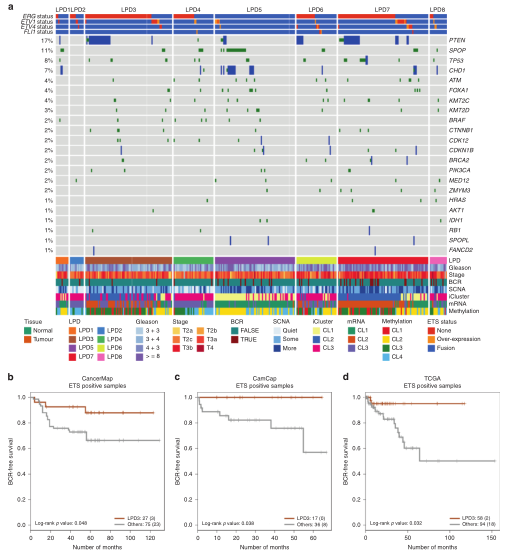
图4.LPD类别的基因组和临床特征
为了获得有关新的LPD类别的信息,作者检查了TGCA数据集分解过程中遗传改变的分布(图4a)。LPD3癌症样本中ETS和PTEN基因高表达,CDH1和SPOP基因的低表达(表2)。LPD5肿瘤样本显示了完全相反的基因改变模式:ETS和PTEN基因改变被抑制,SPOP和CHD1改变过表达(表2)。在TGCA数据集中观察到的分配给LPD3和LPD5的样本中,ETS基因改变的统计差异分布在CamCap和CancerMap数据集中得到了证实(表2)。
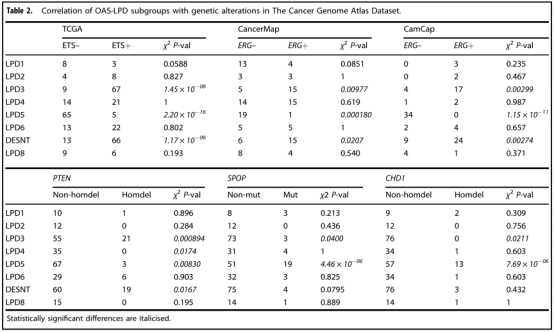
表2. TCGA中的OAS-LPD亚组与遗传变异的相关性
总之,作者确定了另外三种改变遗传或临床关联的前列腺癌类型:LPD3、LPD4和LPD5(图5),它们可能与药物靶向有关。
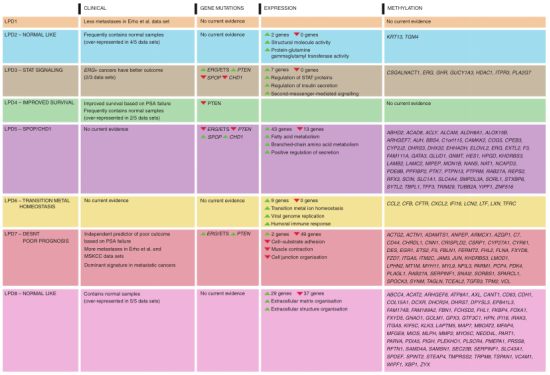
图5.前列腺癌的分析框架
4. 基因表达和DNA甲基化的模式改变
作者检查了分配给每个OAS-LPD signature的样本中所有八个数据集中表达水平明显改变的基因。LPD3癌症样本表现出七个通常过表达的基因,包括ERG,GHR和HDAC1。LPD5表现出47个明显高表达的基因和13个低表达的基因。LPD6-和LPD8癌症未能在这项研究中显示出遗传改变或临床结局的统计学显著变化,但确实具有特征性的基因表达模式改变。可用于TCGA数据集的差异甲基化数据与每个LPD组相关的基因的交叉引用表明,许多表达变化至少可以部分地通过DNA甲基化变化来解释(图5)。
5. 在这一部分,MSKCC研究包括来自19个转移性癌症样本的数据。对于每个转移样本,当使用OAS-LPD时,DESNT是最丰富的signature(图3d)。作者使用了两个数据集MSKCC和Erho表明检查了原发癌表达谱的患者在前列腺切除术后已进展为转移。在MSKCC数据集中发生转移的9位癌症患者中,有5位来自最常见DESNT signature的样本,在212位Erho数据集中发生转移的癌症患者中,有50位来自DESNT癌症。从以上研究中,作者得出结论,DESNT癌症发生转移的风险增加,与PSA失败的较高风险一致。

这些结果凸显了使用更复杂的方法来分析基因组数据的重要性,可能有助于药物靶向,并允许构建结合DESNT和其他临床因素的列线图用于临床管理。

- 发表于 2020-04-14 15:25
- 阅读 ( 5091 )
- 分类:文献解读
