拿一套GEO数据集用DECenter来花式做差异详解
看教程不够直观,那就看视频吧! >>点击加载视频
之前很多人使用DECenter出现各种奇奇怪怪的错误,然后程序猿一直希望将学习成本降到最低,经过多次努力,已更新到了第四版本
百度云下载地址:
64bit:链接:http://pan.baidu.com/s/1mi3OiwS 密码:ppra
32bit:链接:http://pan.baidu.com/s/1pL0unIr 密码:ayfd
这次就用V4来做一下一套GEO芯片数据的差异分析
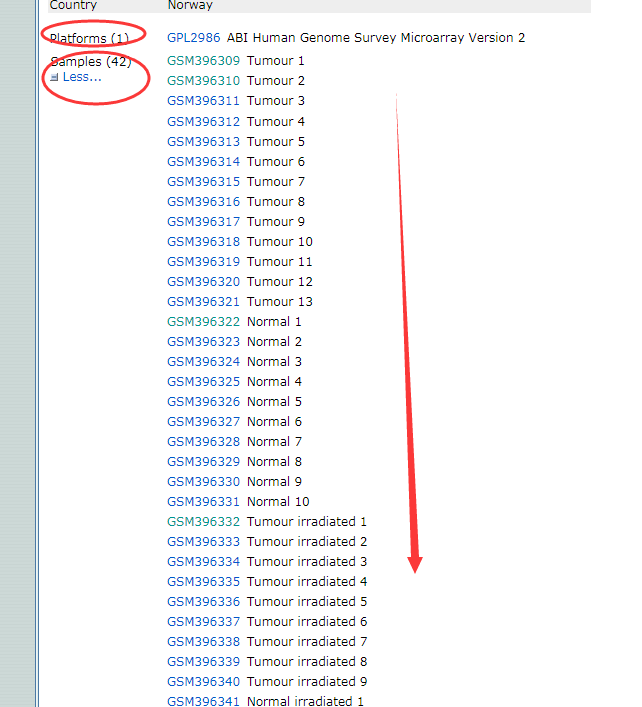
这套芯片数据为:GSE15781,这套芯片是直肠癌的数据,样本总共有42个,做差异分析之前我们都应该先看懂咱们自己的数据,如下图,从图中可以看出这套数据使用的平台是GPL2986,42个样本,分别有四类
1、肿瘤样本
2、正常样本
3、经过放疗的正常样本
4、经过放疗的癌症样本
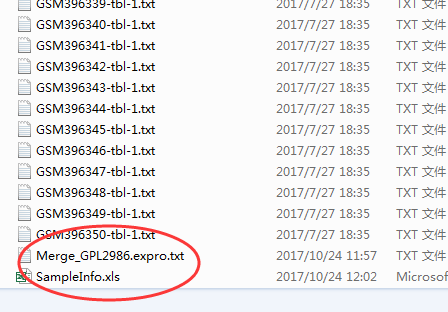
进一步的我们下下载这套GEO的数据集使用GEO芯片转换器进行数据提取,不会的回头看如何提取GEO的数据
最终我们会得到两个表:
 现在我们需要修改一下SampleInfo.xls文件以便对这些样本进行更好的分组比较,打开这个表格(Excel 会提示如:https://www.shengxin.ren/question/386),看到如下图的文件表格(注意你的数据中每个后面带了分号,我这里把分号替换掉了)
现在我们需要修改一下SampleInfo.xls文件以便对这些样本进行更好的分组比较,打开这个表格(Excel 会提示如:https://www.shengxin.ren/question/386),看到如下图的文件表格(注意你的数据中每个后面带了分号,我这里把分号替换掉了)

现在我们需要分组,假设我们设想的比较方式是这样的:
1、癌症样本与正常样本比较
2、放疗样本与非放疗样本比较
3、癌症放疗样本与非癌症放疗样本比较
那么从上面的三种比较方式我们可以看出需要以下几组样本
1、癌症样本,使用Tumor表示
2、正常样本,使用Normal表示
3、放疗样本,使用Irrad表示
4、非放疗样本,使用NoIrrad表示
5、癌症放疗样本,使用TumorIrrad
6、非癌症放疗样本,使用NoTumorIrrad
然后我们将这六组样本分别拿出来比如第一组,注意哦,我在Excel里面重新建了个表
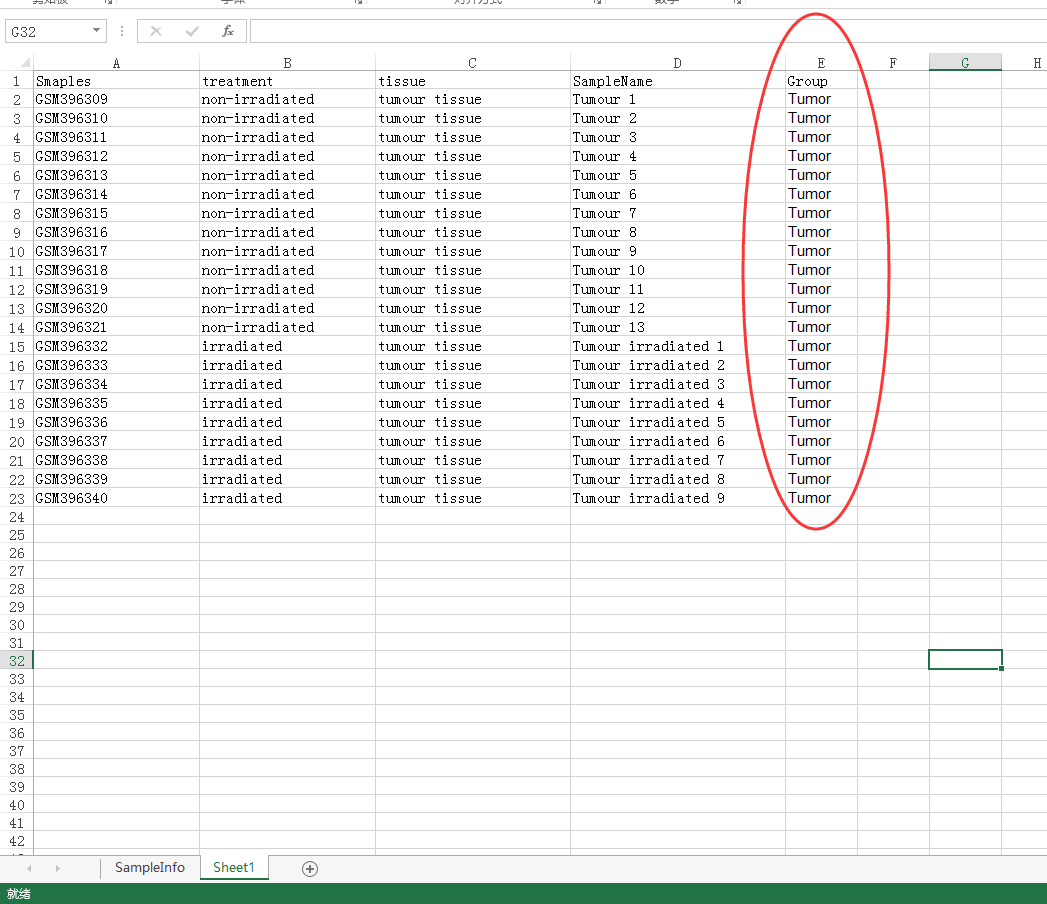
加入第二组样本,即正常样本如:
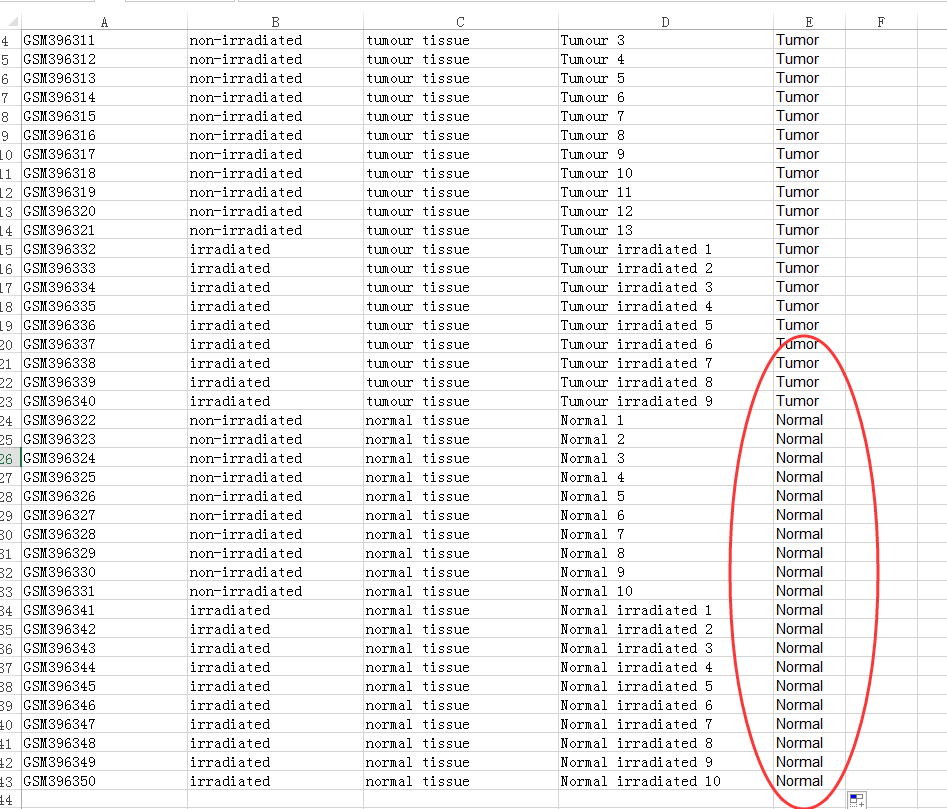
加入第三组样本,即放疗样本如:
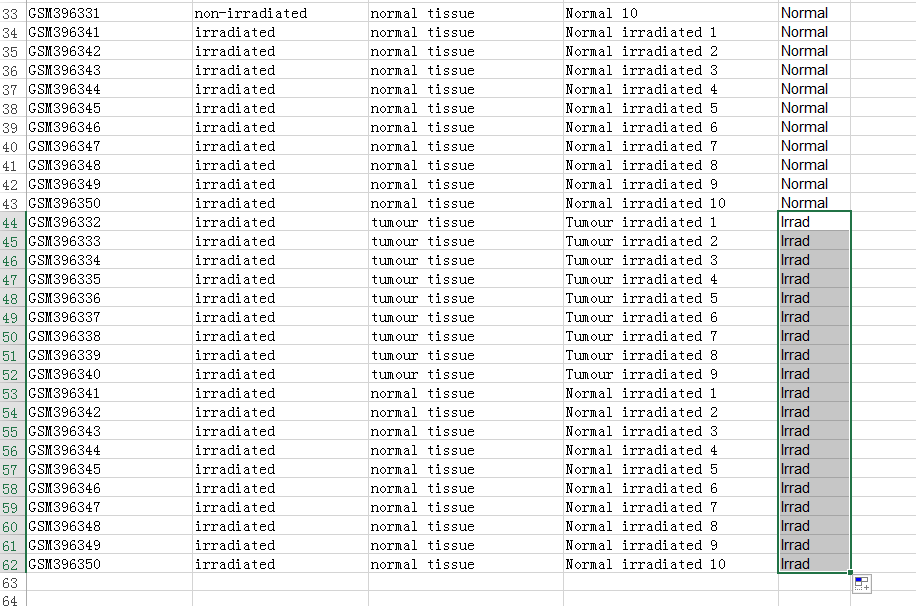 依次类推往表格后面加就行,所有组加完了另存为一个文本文件就行,比如下图:new_sample.txt
依次类推往表格后面加就行,所有组加完了另存为一个文本文件就行,比如下图:new_sample.txt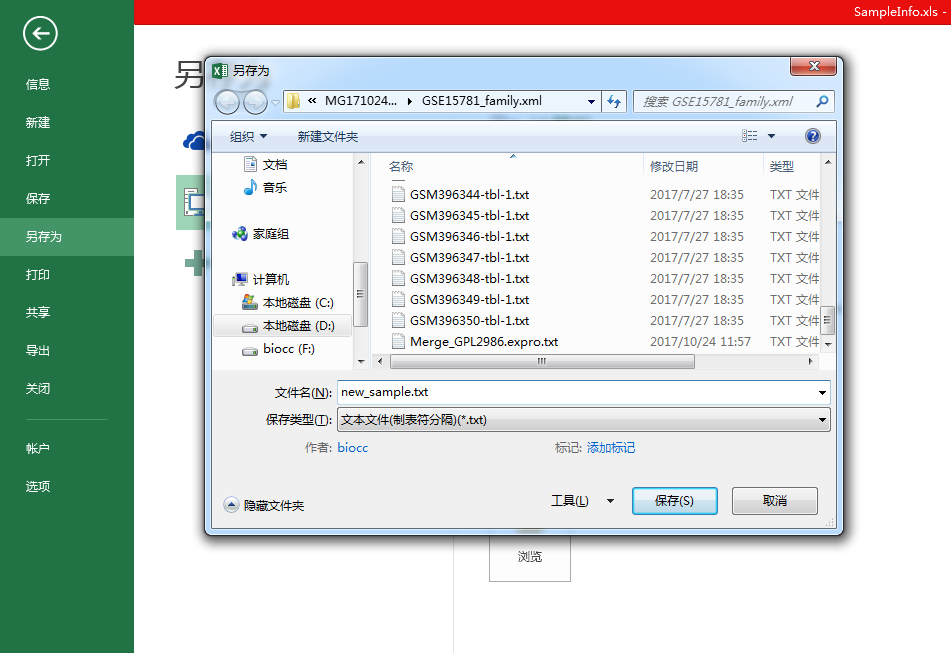
用文本打开new_sample.txt看看格式有没有坏,整齐的就好:
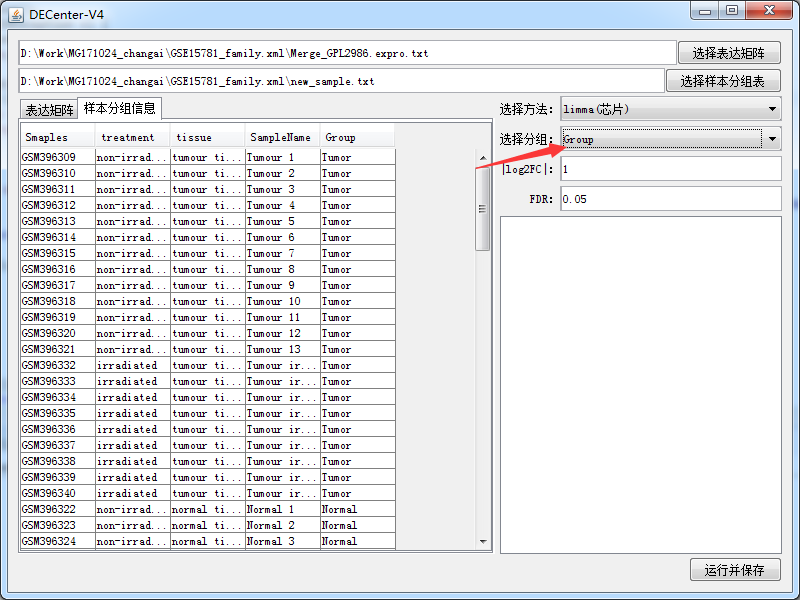
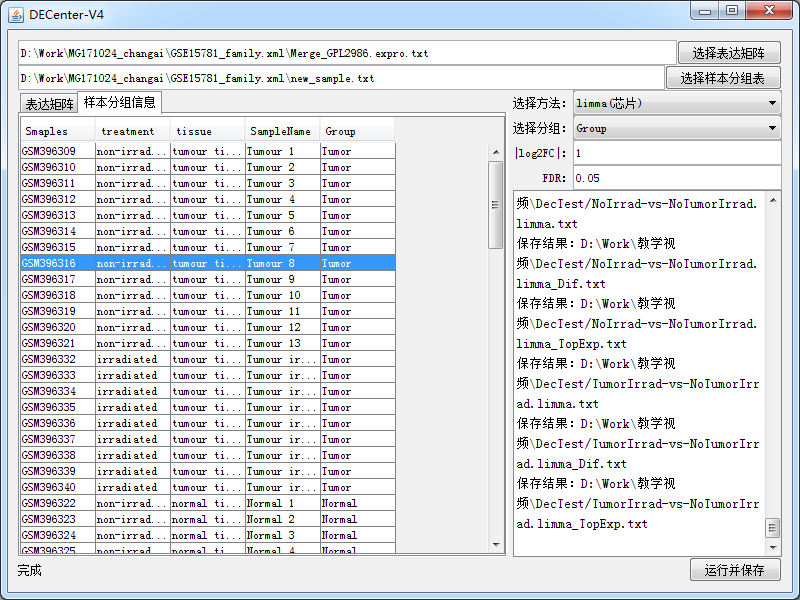
数据准备完毕了之后导入我们的DECenter软件如
 注意箭头处一定要选对,点击运行并选择结果保存目录。
注意箭头处一定要选对,点击运行并选择结果保存目录。
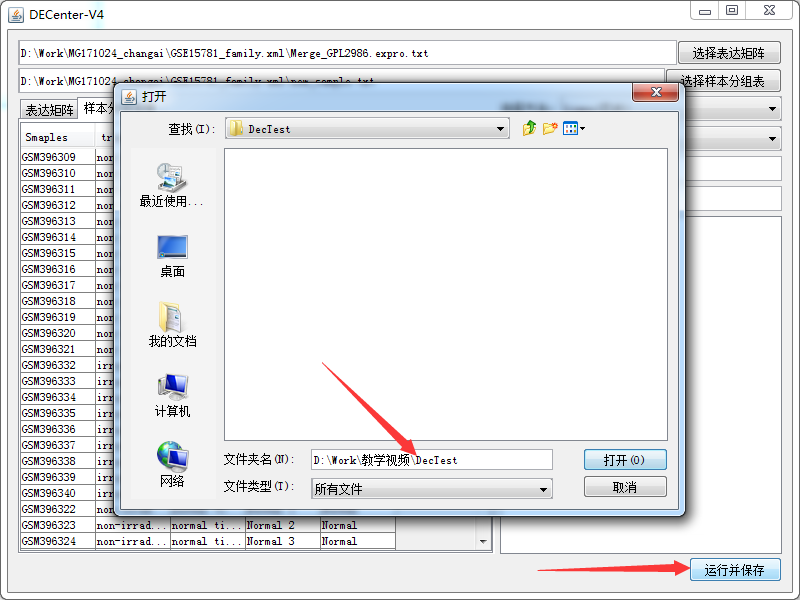
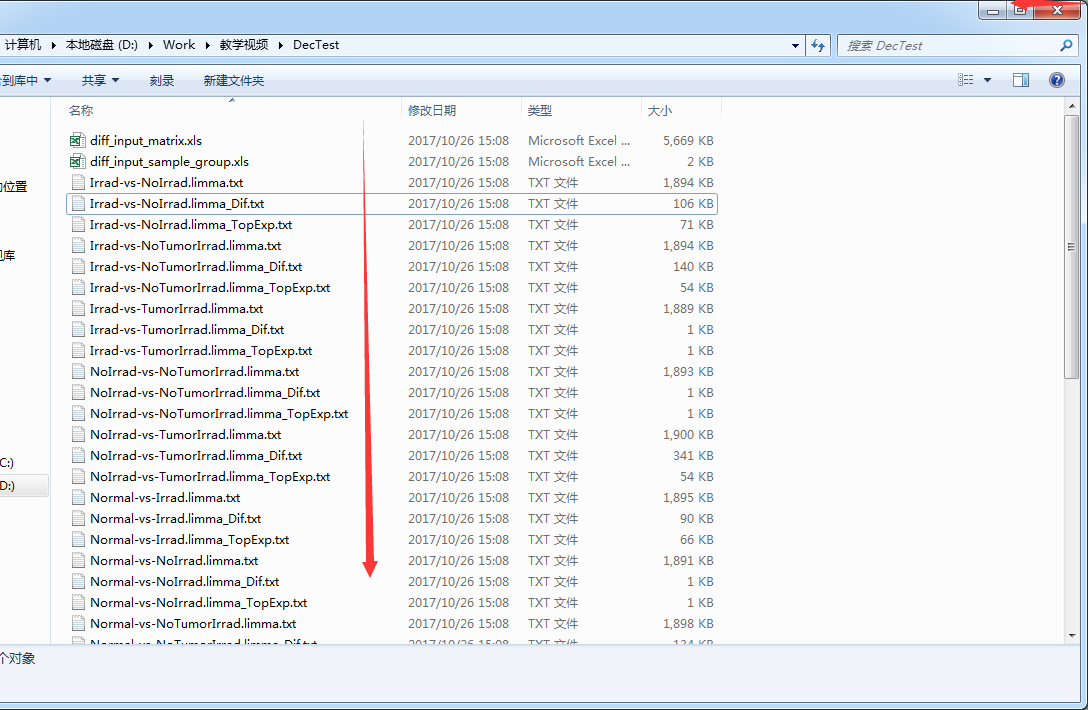
然后等待程序运行完成即可,根据数据量和电脑配置,时间长短不一,最终结果如下:
- 发表于 2017-10-26 15:20
- 阅读 ( 26748 )
- 分类:软件工具
你可能感兴趣的文章
- 转录组Count数据差异分析工具 7102 浏览
- 在GEO数据库如何找带有预后信息的数据?一分钟搞定,适用于找任何的样本特征数据 20966 浏览
- 当在文献中看到一个GEO数据集却在GEO中没有权限下载的时候,应该想到ArrayExpress 5749 浏览
- 如果导师丢给你一个基因让你去研究,不妨试一下这两个五分的套路 26049 浏览
- 零代码对不同类型的RNA Seq数据进行差异分析 6780 浏览
- 文章解读——基于20个基因的预后模型预测肺腺癌的生存 12382 浏览
