零代码对不同类型的RNA Seq数据进行差异分析
TCGA数据据是由美国国家癌症研究所(NCI)及国家人类基因组研究所(NHGRI)联合建立,其包含丰富的肿瘤数据类;ceRNA其主要基于竞争性内源RNA假说解释mRNA、假基因、长链非编码RNA之间如何通过miRNA反应元件进行“对话”。本节主要讲解TCGA中不同类型RNA-Seq数据差异分析方法。
看教程不够直观,那就看视频吧! >>点击加载视频
上一节课我们已经将三种类型的数据准备和预处理完成,那么本节将讲解怎么对三种数据进行降维,缩小目标基因范围,不错有的小伙伴已经猜到了,差异分析是我们目前最为普遍的方法,但是针对不多的数据类型该使用哪种统计方法比较合理呢,我们且听老师讲解
1、差异分析同样可以使用SangerBox中的DEcenter差异分析工具,没有用过的小伙伴可以点开连接看一下哦
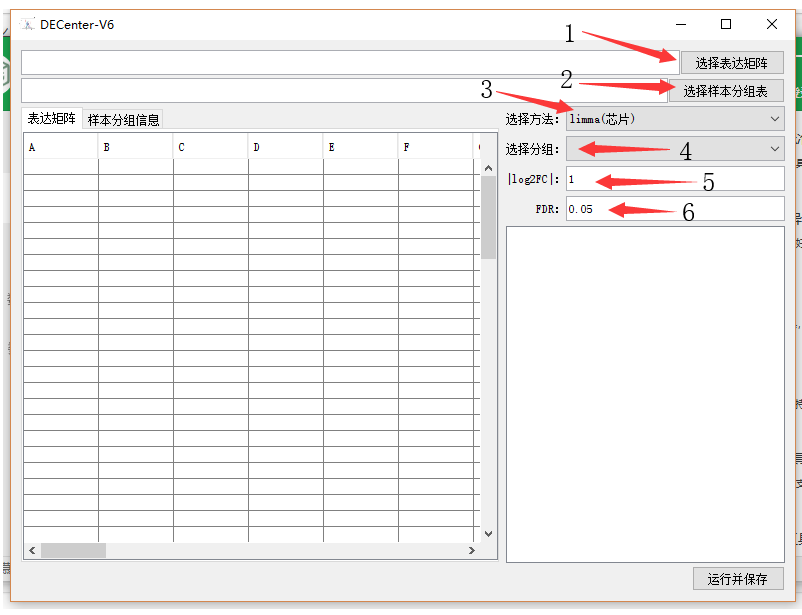
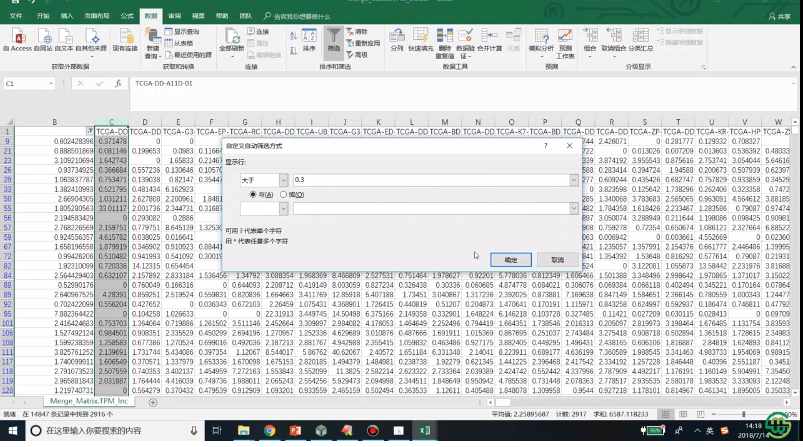
2、先是mRNA差异分析,输入表达数据比较容易,样本文件格式如何,生成的具体操作
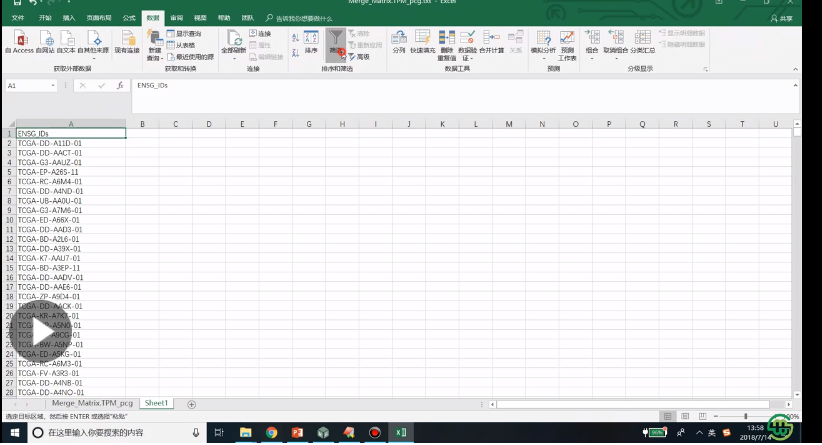
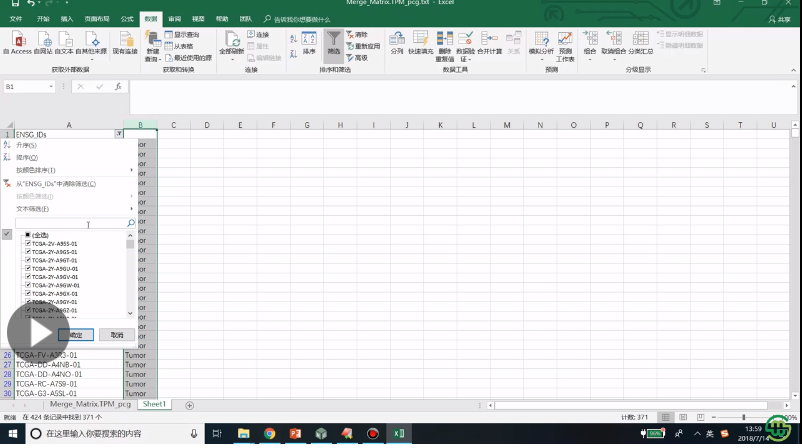
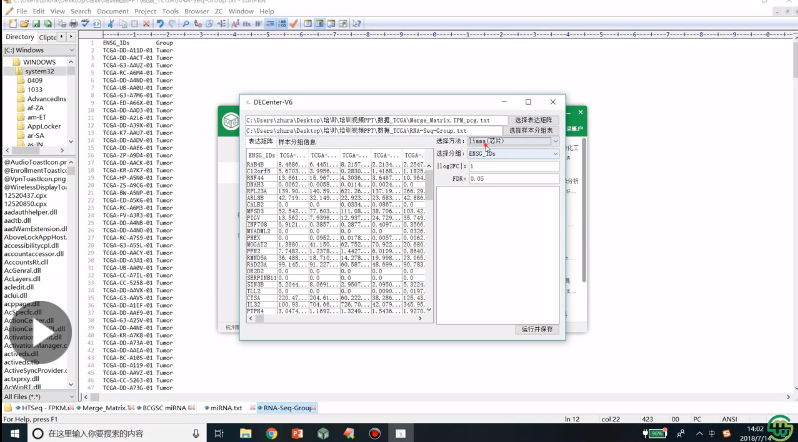
3、miRNA首先对数据进行过滤,通过excel的实现过程,然后是差异分析方法选择
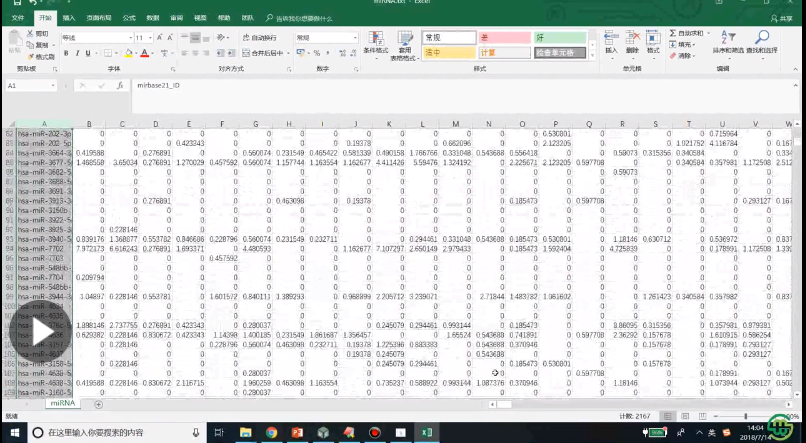
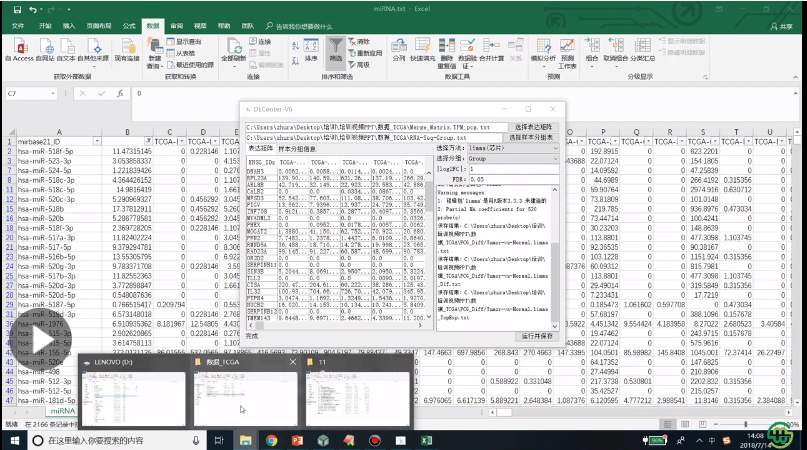
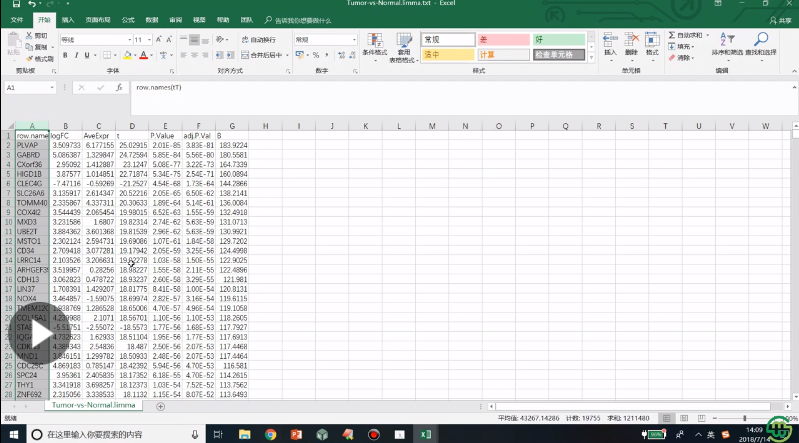
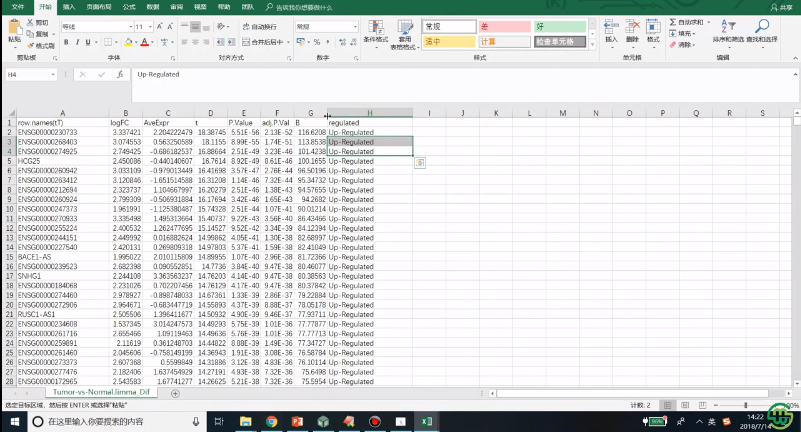
4、工具生成差异分析结果文件的解析
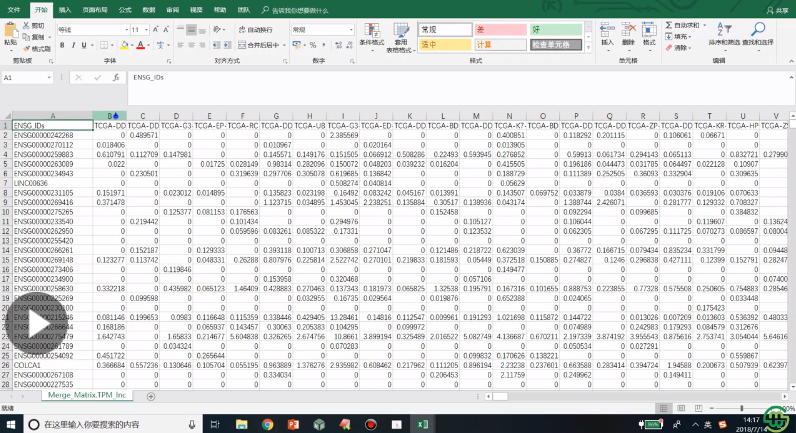
5、最后是lncRNA数据类型的寻找差异基因的步骤
本节课程完毕,下一节课将开始讲解miRNA与mRNA的网络构建

- 发表于 2018-11-16 13:31
- 阅读 ( 6838 )
- 分类:基因组学
你可能感兴趣的文章
- limma快速差异分析工具 8000 浏览
- 转录组Count数据差异分析工具 7256 浏览
- 快速差异分析工具——limma包 25927 浏览
- limma快速差异分析工具! 8162 浏览
- limma的两个弟弟:edgeR和DESeq2 8382 浏览
0 条评论
请先 登录 后评论
