边测序边分析可能不是梦
看教程不够直观,那就看视频吧! >>点击加载视频
听说过Real-time实时测序,那有没有听说过real-time实时分析?
相信接触过二代和三代测序的大家应该对Read-time Sequencing 并不陌生,在这个基因测序大范围普及的时代,Real-time Sequencing 扮演了不可或缺的角色。实时测序即边合成边测序,大部分二代测序比如Illumina, 454 SOliD Sequencing和三代测序PacBio SMRT sequencing, Oxford nanopore Seqeuencing都属于边合成边测序。传统的测序都是需要在全部测序完成之后,对低质量数据进行过滤,然后在进行基因组装和后续的分析。做过基因组装的人经常会因为数据量测不够而导致组装效果不理想,进而导致后续分析问题频出,又或者是因为数据量过多而导致时间和成本的大量浪费。最新发表在Nature Communication上的一篇文章开发了一种新的组装软件: npScarf。
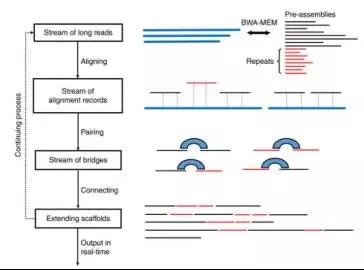
这种组装方法主要原理是基于短读长reads的测序数据,再用长读长的实时纳米孔数据将短reads建立关系连接起来。首先,npScarf将Illumina短reads分成两类:唯一个contigs和重复序列。之后将一次一次传输过来的长读长的纳米孔reads数据建立scaffolds和填充gaps。在收到数据的时候,nfScarf会将长reads比对到短的唯一的contigs上,并通过打分系统给他们打分,最后连接打分最高的两条reads会被连接起来并分配到相应的位置。
对于那些重复的序列,npScarf会将它们填充到scaffold 之间的gap区域。在组装过程中,该软件会实时计算和输出ScaffoldN50, Contigs个数等组装指标,方便我们实时监控组装的完整性。
至于这种组装方法的具体实行效果,文章对细菌K. pneumoniae等细菌种株的组装进行了比较。随着时间和深度的增加,组装的contig个数不断减少,Scaffold N50不断增加,在大概到达18~20X的时候,达到了比较理想的组装效果。
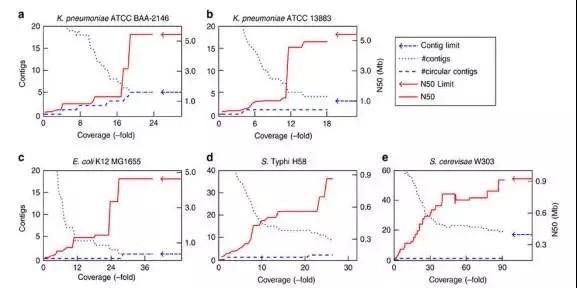
当然我们也很好奇这种方法和现有的装方法, 文章对不同数据量的长reads数据进行了全面的比较,我们选取了其中一张展示:
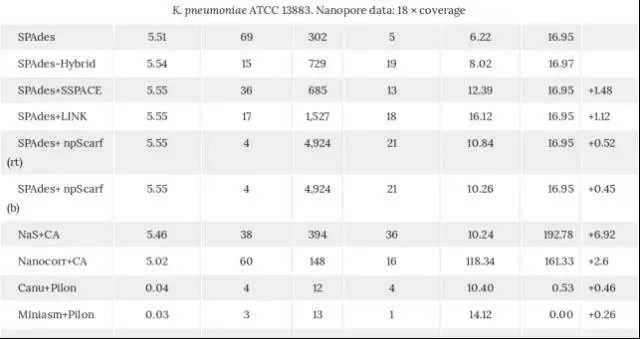
npScarf 不管在多少长reads的情况下,都获得了最完整的组装和比较好的准确性。
除此之外,在组装的同时,我们还可以进行一些相关的后续分析,比如基因组岛和质体上的基因鉴别。
目前为止,npScarf是第一款在测序进行的时候就可以组装的软件。它也是得益于纳米孔单分子测序技术的发展。他最主要的优点就是可以极大的提高测序效率,在获得完整的基因组情况下减少成本和测序时间,并且兼容后续的基因组分析。在测序的时候,研究人员可以根据组装的效果来随时决定是否已获得足够的数据来停止测序,从而避免了数据量不够而导致项目延期情况的发生。这种测序分析一体化的流程可以方便研究人员获取有效信息,避免冗余数据产生,极大加快研究速度。
在当前这个时间就是金钱的社会,尤其实文章发表时间的早晚与否决定了文章发表期刊的质量。选择一体化的组装和分析软件将来会是我们基因信息行业的一种趋势。
参考文献
Cao, M. D. et al. Scaffolding and completing genome assemblies in real-time with nanopore sequencing. Nat. Commun. 8, 14515 doi: 10.1038/ncomms14515 (2017).
- 发表于 2017-04-27 17:32
- 阅读 ( 4435 )
- 分类:基因组学
你可能感兴趣的文章
- 文献解读(4)NC甲状腺癌的刻画 7073 浏览
- 〖生物技术〗单细胞测序方法大比拼 5623 浏览
- 数据分析技巧、基因定位文献解读合辑 8827 浏览
- MECAT:PC组装人的基因组 9832 浏览
- 第一代测序技术介绍 8597 浏览
