文献解读(1)What?这篇文章为预测预后和发现抗转移药物提供了帮助?
看教程不够直观,那就看视频吧! >>点击加载视频


肾癌是泌尿系统常见的恶性肿瘤,多起源于肾小管上皮细胞,近年来其发病率在世界范围内呈上升趋势。根据最近的癌症统计报告,美国每年新增的确诊病例已达65,000例,其导致每年约15,000人死亡。透明细胞肾细胞癌(ccRCC)是最常见的散发性肾癌(约80%)的病理组织学类型,与其他类型的肿瘤相比,其生存预后较差。近20%的ccRCC病例在诊断开始时进展到晚期,转移性病例的5年总生存率(OS)下降到约10%。随着手术干预、放疗和免疫治疗的发展,肿瘤治疗的组合策略得到了很大的优化。然而,实际的临床效率仍有待提高,30%的ccRCC患者不可避免地出现复发和癌症相关进展。
因此,ccRCC转移或进展的分子机制及新的靶点的研究迫在眉睫。这次小编带来的这篇文章就是基于单细胞RNA-seq数据开发验证了一个转移相关的预后特征集。
标题:Development and validation of a metastasis-associated prognostic signature based on single-cell RNA-seq in clear cell renal cell carcinoma
杂志:AgING IF: 5.543 发表日期:2019.10.29

1) 数据:来自GEO的GSE73121数据集的121个细胞样本的原始转录数据;来自TCGA的537个ccRcc的表达谱;以及ICGC的91个患者的表达信息;同时也收集了这些样本的临床信息。
2) 单细胞数据的处理:推断基因组信息,过滤低质量细胞。
3) 在ccRcc样本队列中识别MAGs:首先从RNA-seq数据中检测marker基因。将TCGA-KIRC分为训练集和检验集。使用LASSO推断预后相关的hub基因。构建预后特征MAGs,生存分析,ROC评估。
4) 开发一个检测进展的个性化预测模型:整合MAG和其他临床因素,进行单因素以及多因素分析。
5) TMB的谱系以及相关分析
6) 两个MAG组的功能富集分析:GSEA

① 单细胞RNA-seq谱以及转移相关的marker基因的过滤
作者获取了3个亚群的121个细胞样本,如表1所示。对数据的质量控制流程如图1A所示。作者观察到检测的基因数量与测序深度显著正相关(图1B)。而图1C展示了TOP的显著差异表达基因。在图1D中作者使用主成分分析划分细胞。图F展示了t-SNE的聚类结果。得到MAGs之后将得到的两类细胞的热图如图1G所示,在图1H中我们可以看到明显的转录异质性。
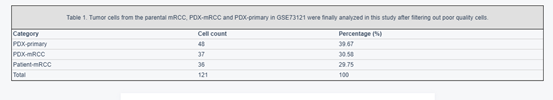
表1
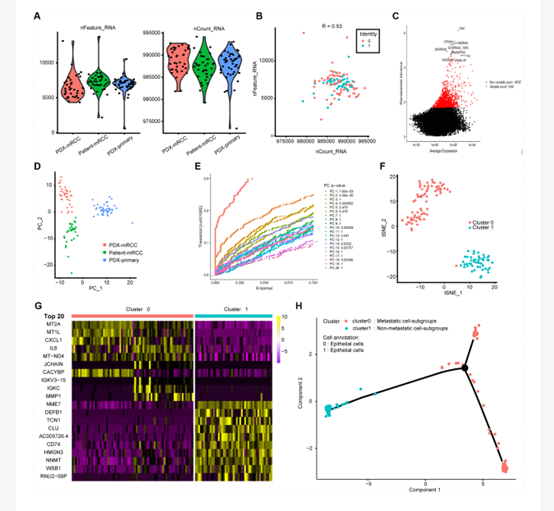
图1
② 在内部和外部ccRcc数据中对MAGs验证
作者首先整合44个差异hub MAGs的表达谱以及生存信息。使用LASSO筛选了17个预后相关基因(图2A,B)。样本对应的临床信息如表2所示。作者在两类中刻画了这17个预后相关的基因。接着使用多因素COX回归模型建立MAG特征。其AUC,ROC分别为0.763以及0.803。作者同时进行了生存分析以及外部数据验证。
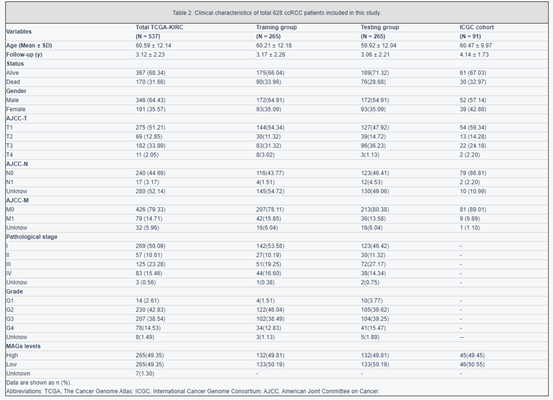
表2
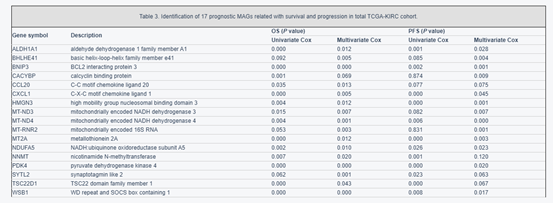
表3
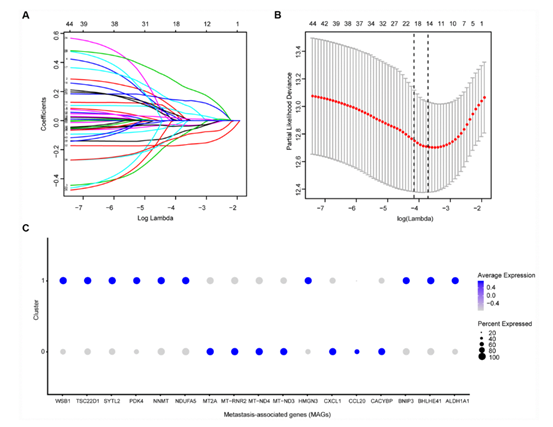
图2
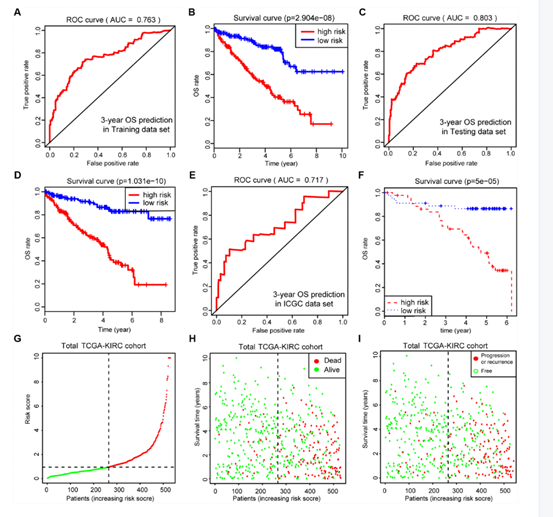
图3
③ MAGs和其他临床特征的相关性分析
作者为了探究MAGs在ccRCC中的临床意义,所以研究了MAGs与其他临床特征之间的潜在关系。Kruskal-Wallis检验显示,MAG评分升高与T分期增高(P = 7.586e-09)、淋巴结阳性率增高(P = 0.005)、晚期转移(P = 1.572e-06)、病理分期较差(P = 1.699e-08)和肿瘤进展分级(P = 1.643e-11)相关。此外,在TCGA-KIRC总队列中,MAG特征预测5年PFS较好,其AUC为0.752(图4F),并且通过log-rank检验证明MAG评分高的患者在肿瘤复发或进展方面具有更大的风险(图4G)。此外,作者在另一个ICGC数据集中验证了潜在的关系,发现MAG评分与T期(P = 4.364e-04)和转移状态(P = 3.436e-05)显著相关。
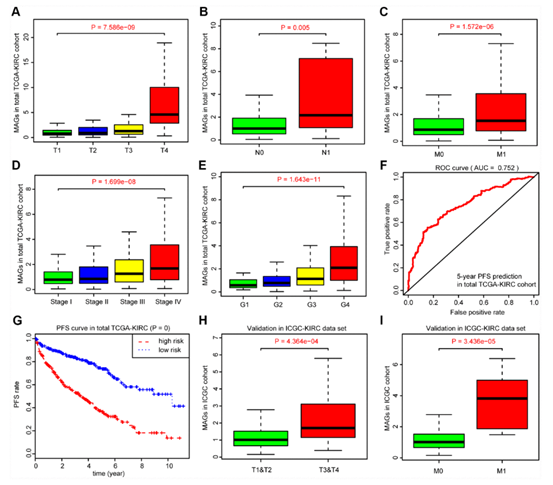
图4
④ 构建预测进展的MAG nomogram
然后,作者将MAG信号与其他独立临床变量相结合,构建一个全面的ccRCC进展监测模型。排除了超过半数的缺失病例的N期因素,并且忽略了在多变量Cox回归模型中没有统计学意义的变量。最终选择了年龄、肿瘤分级、病理分期和MAG特征四个独立的风险特征作为模型(图5A)。利用广义线性模型(GLM)回归算法,开发了包含这四个特征的MAG nomogram,如图5B所示。根据MAG nomogram评分的中位数将TCGA-KIRC队列分为高分组和低分组。根据预测的1年或3年进展/复发事件与实际观察结果之间的一致性,绘制校准曲线来描述拟合模型(图5C)。MAG nomogram预测1年和3年进展结果的auc分别达到0.848和0.837(图5D)。生存分析也表明,MAG nomogram是ccRCC PFS的显著预测因子,P = 0(图5E)。
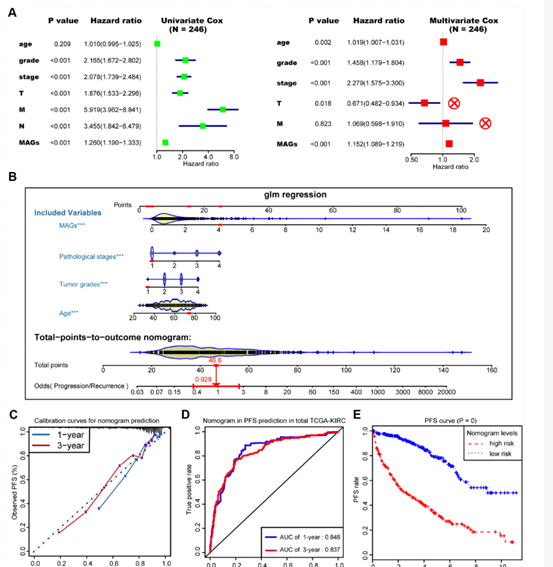
图 5
⑤ 两个nomogramm评分水平之间的体细胞突变负担差异
作者在TCGA-KIRC队列中定义并计算了TMB,并将其与相应的MAG nomogram评分进行匹配。此外,计算了分布在5%以上样本中的突变的差异突变率,经卡方检验发现,SETD2、BAP1和MTOR在高危组的突变数量明显多于低危组(图6A)。此外,Wilcoxon秩和检验表明,高TMB组的MAG nomogram风险评分显著高于低TMB组(P = 2.875e-05)。进一步分析了TMB在ccRCC中的生存意义,发现TMB水平越高,进展事件的风险越大,P = 0.01(图6C), OS结果越差,P = 0.035(图6D)。因此,推测高MAG nomogram评分的ccRCC患者存在较高的TMB水平,这也被证明是ccRCC的一个危险因素。
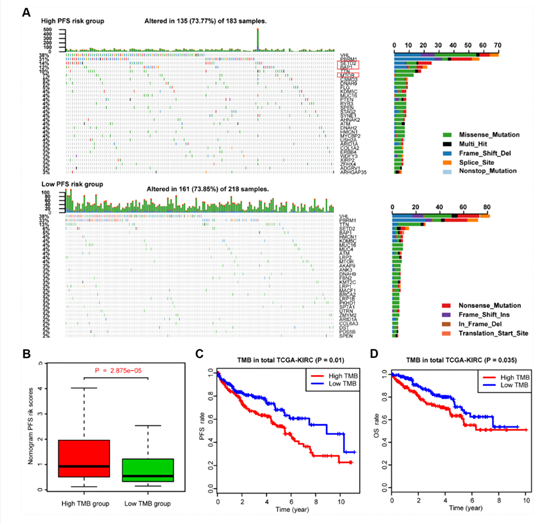
图6
⑥ GSEA
选择517例ccRCC患者的转录组数据,以MAG nomogram评分作为参考表型,进行基因集富集分析(GSEA)。观察到,氧化磷酸化、Wnt信号通路、MAPK信号通路和肾细胞癌串扰在高危组中上调。低危组P53信号通路、系统性红斑狼疮、果糖代谢均下调(图7)。所有这些异常通路均富集为恶性肿瘤的标志。
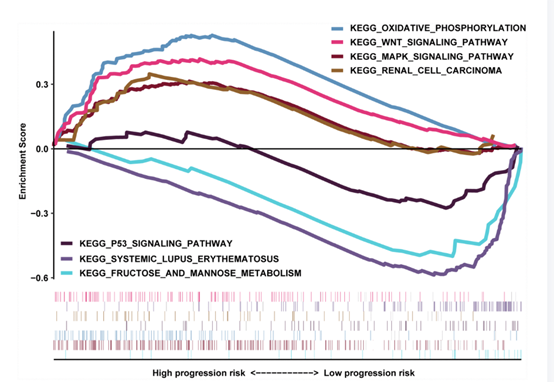
图7
 本研究从单细胞测序数据入手,结合多种生物信息方法,筛选了基于scRNA-seq的标记基因,并在大量ccRCC样本中进行了验证。不仅描述了pRCC和mRCC的基因组特征和异质性,还发现了一些MAGs,为预测预后和发现抗转移药物提供了可能的帮助。
本研究从单细胞测序数据入手,结合多种生物信息方法,筛选了基于scRNA-seq的标记基因,并在大量ccRCC样本中进行了验证。不仅描述了pRCC和mRCC的基因组特征和异质性,还发现了一些MAGs,为预测预后和发现抗转移药物提供了可能的帮助。

1. Barata PC, Rini BI. Treatment of renal cell carcinoma: current status and future directions. CA Cancer J Clin. 2017; 67:507–24. https://doi.org/10.3322/caac.21411 [PubMed]
2. Hakimi AA, Voss MH, Kuo F, Sanchez A, Liu M, Nixon BG, Vuong L, Ostrovnaya I, Chen YB, Reuter V, Riaz N, Cheng Y, Patel P, et al. Transcriptomic Profiling of the Tumor Microenvironment Reveals Distinct Subgroups of Clear Cell Renal Cell Cancer: Data from a Randomized Phase III Trial. Cancer Discov. 2019; 9:510–25. https://doi.org/10.1158/2159-8290.CD-18-0957 [PubMed]
3. Mitchell TJ, Turajlic S, Rowan A, Nicol D, Farmery JH, O’Brien T, Martincorena I, Tarpey P, Angelopoulos N, Yates LR, Butler AP, Raine K, Stewart GD, et al, and TRACERx Renal Consortium. Timing the Landmark Events in the Evolution of Clear Cell Renal Cell Cancer: TRACERx Renal. Cell. 2018; 173:611–623.e17. https://doi.org/10.1016/j.cell.2018.02.020 [PubMed]
4. Miao D, Margolis CA, Gao W, Voss MH, Li W, Martini DJ, Norton C, Bossé D, Wankowicz SM, Cullen D, Horak C, Wind-Rotolo M, Tracy A, et al. Genomic correlates of response to immune checkpoint therapies in clear cell renal cell carcinoma. Science. 2018; 359:801–06. https://doi.org/10.1126/science.aan5951 [PubMed]
5. Syafruddin SE, Rodrigues P, Vojtasova E, Patel SA, Zaini MN, Burge J, Warren AY, Stewart GD, Eisen T, Bihary D, Samarajiwa SA, Vanharanta S. A KLF6-driven transcriptional network links lipid homeostasis and tumour growth in renal carcinoma. Nat Commun. 2019; 10:1152. https://doi.org/10.1038/s41467-019-09116-x [PubMed]
6. Rojas JD, Lin F, Chiang YC, Chytil A, Chong DC, Bautch VL, Rathmell WK, Dayton PA. Ultrasound Molecular Imaging of VEGFR-2 in Clear-Cell Renal Cell Carcinoma Tracks Disease Response to Antiangiogenic and Notch-Inhibition Therapy. Theranostics. 2018; 8:141–55. https://doi.org/10.7150/thno.19658 [PubMed]
7. Li JK, Chen C, Liu JY, Shi JZ, Liu SP, Liu B, Wu DS, Fang ZY, Bao Y, Jiang MM, Yuan JH, Qu L, Wang LH. Long noncoding RNA MRCCAT1 promotes metastasis of clear cell renal cell carcinoma via inhibiting NPR3 and activating p38-MAPK signaling. Mol Cancer. 2017; 16:111. https://doi.org/10.1186/s12943-017-0681-0 [PubMed]
8. Dai J, Lu Y, Wang J, Yang L, Han Y, Wang Y, Yan D, Ruan Q, Wang S. A four-gene signature predicts survival in clear-cell renal-cell carcinoma. Oncotarget. 2016; 7:82712–26. https://doi.org/10.18632/oncotarget.12631 [PubMed]
9. Chang P, Bing Z, Tian J, Zhang J, Li X, Ge L, Ling J, Yang K, Li Y. Comprehensive assessment gene signatures for clear cell renal cell carcinoma prognosis. Medicine (Baltimore). 2018; 97:e12679. https://doi.org/10.1097/MD.0000000000012679 [PubMed]

- 发表于 2019-11-27 17:24
- 阅读 ( 4750 )
- 分类:文献解读
