一篇典型的WGCNA文章解读
我们鉴定了两个模块和10个hub基因,这些基因与OSCC的肿瘤发生有关。
这两个模块提供的参考将促进对OSCC中肿瘤发生机制的理解。
此外,hub基因可以作为生物标志物和治疗靶标,用于未来OSCC的精确诊断和治疗。
看教程不够直观,那就看视频吧! >>点击加载视频
A pplication of weighted gene co-expression network analysis to identify key modules and hub genes in oral squamous cell carcinoma tumorigenesis
摘要
目的:口腔鳞状细胞癌(OSCC)是世界上最常见的恶性疾病,然而,它的分子机制在很大程度上是未知的。
我们的目标是构建基因共表达网络,以确定与发病机制相关OSCC的关键模块和hub基因。
患者和方法:我们使用GSE30784数据集通过加权基因共表达网络分析(WGCNA)构建共表达网络。
使用DAVID对数据集进行Gene Ontology(GO)和Kyoto Encyclopedia of Genes and Genomes(KEGG)富集分析。 使用其他数据集筛选和验证hub基因。
结果:发现蓝绿色和棕色模块与肿瘤最显着相关。功能富集分析表明,蓝绿色模块与细胞间粘附,细胞外基质和胶原分解代谢过程有关。
10个hub基因(MMP1,TNFRSF12A,PLAU,FSCN1,PDPN,KRT78,EVPL,GGT6,SMIM5和CYSRT1)在转录和翻译水平上被鉴定和验证。
他们的基因改变和生存分析也被挖掘。
结论:我们鉴定了两个模块和10个hub基因,这些基因与OSCC的肿瘤发生有关。
这两个模块提供的参考将促进对OSCC中肿瘤发生机制的理解。
此外,hub基因可以作为生物标志物和治疗靶标,用于未来OSCC的精确诊断和治疗。
介绍
头颈部鳞状细胞癌(HNSC)是一种常见的恶性肿瘤,占头颈癌的90%,口腔鳞状细胞癌(OSCC)是HNSC的主要压型。
OSCC的特点是在口腔内发病率高,并与吸烟饮酒相关。
同样上皮细胞遗传改变也会引发OSCC。加权基因共表达网络分析是一种新型分析工具,可以找到潜在的基因表达模块。
使用加权基因共表达网络有助于理解分析OSCC发生的分子机制。
结果1 Microarray数据的表达分析
为了建立基因共表达网络,GSE30784的原始数据从GEO中下载。原始数据使用R进行背景相关性和标准化的预处理。
R包注释用来区配探针到基因symbol,匹配到多个基因的探针被去除,一个基因匹配到多个探针的,中值作为最终的表达值。
最终,作者获得了20027个基因。然后作者计算了每个基因的SD,将它们从大到小排序,前5000个基因被用来做WGCNA。
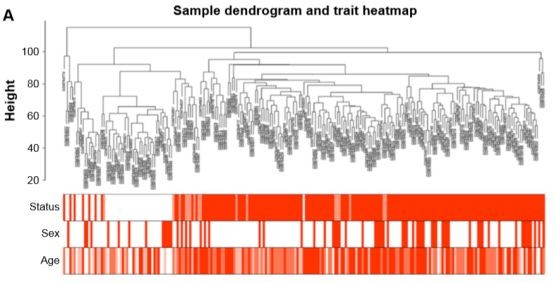
使用WGCNA包对这5000个基因进行聚类分析,如下图所示:可以看出,229个样本被整体分成两组。
结果2 权重共表达网络的建立和核心模块的识别
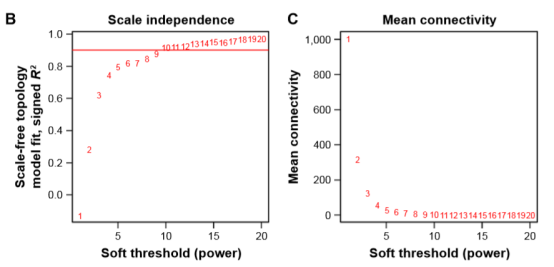
选择soft-thresholding power是构建WGCNA时的重要步骤。我们对1到20的soft-thresholding power进行了网络拓扑分析,
并确定了相对平衡的规模独立性和WGCNA的平均连通性。
如下图所示,选择阈值9,其为0.9的无标度拓扑拟合指数的最低功率,
以产生5,000个基因的分层聚类树。
作者将MEDissThres设置为0.25以合并类似的模块,并生成了11个模块。如下图所示:
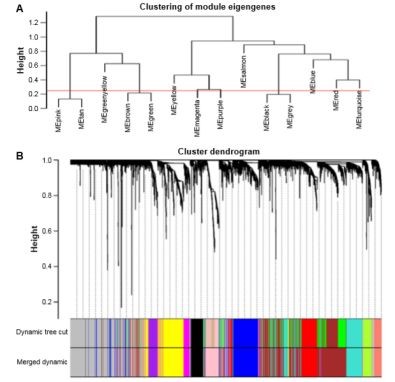
其中黑色模块中有223个基因,蓝色模块中有518个基因,棕色模块中有954个基因,黄绿模块中有139个基因,品红色模块中有172个基因,
粉红色模块中有302个基因,紫色模块中有143个基因,红色模块中的379个基因,浅澄色模块中的112个基因,
蓝绿色模块中有530个基因,黄色模块中有471个基因。将不能包含在任何模块中的基因放入灰色模块中,在随后的分析中将其除去。
结果3 模块之间的关联和关键模块的识别
本文分析了11个模块的交互关系,绘制了热图。如下图所示:
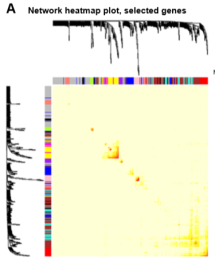
结果显示,每个模块都是相互独立的验证,证明了模块之间的高度独立性以及每个模块中基因表达的相对独立性。
此外,本文根据eigengenes的相关性对它们进行聚类,以探索所有模块的共表达相似性,如下图所示:
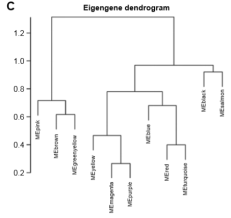
本文发现11个模块主要分为两个簇。根据邻接关系绘制的热图表明了类似的结果。如下图所示:
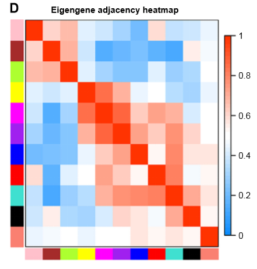
此外,与其他模块相比,蓝绿色模块和棕色模块的ME显示与疾病状态(正常,发育异常和OSCC)高度相关。
蓝绿色模块与疾病状态呈正相关,而褐色模块与疾病状态呈负相关,表明蓝绿色模块可能在OSCC的肿瘤发生中起重要作用,
棕色模块可能起到抗肿瘤的作用。我们将蓝绿色模块和棕色模块确定为与OSCC疾病状态最相关的模块。
下图分别显示出了蓝绿色和棕色模块中模块成员与基因显著性之间的相关性。
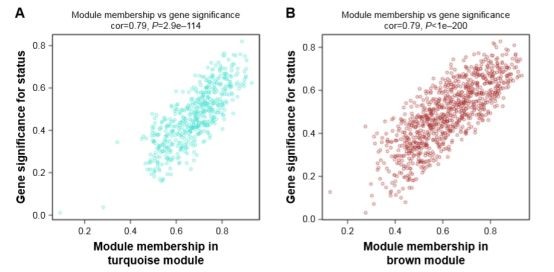
结果4 识别蓝绿色模块和棕色模块中的hub基因
本文使用Cytoscape将蓝绿色模块和棕色模块可视化为网络,并通过对节点度候选基因进行排序筛选出30个基因用于进一步分析。
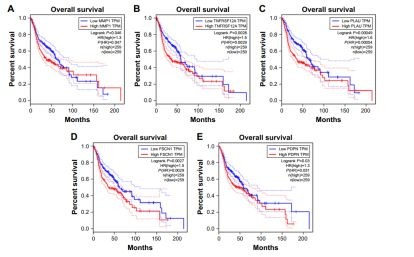
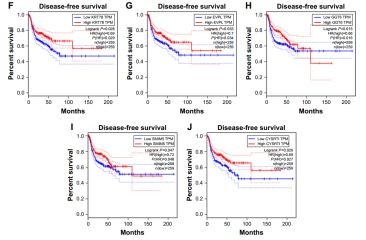
使用 GEPIA做生存分析。选择具有显著生存分析结果(p<0.05)的基因并按节点程度进行排序。
每个模块中的前五个基因被认为是hub基因。下图显示出hub基因的生存分析(A-E是蓝绿色模块中的hub基因,F-J是棕色模块中的hub基因)。
它们分别是蓝绿色模块中的MMP1,TNFRSF12A,PLAU,FSCN1和PDPN以及棕色模块中的KRT78,EVPL,GGT6,SMIM5和CYSRT1。
10个基因的生存分析在另一组数据集GSE41613中被验证。
结果5 2个核心模块的功能富集分析
使用DAVID做BP的GO富集分析,结果表明,褐色模块主要富集表皮的调节,如表皮发育,上皮细胞分化和角质形成细胞分化,与肿瘤发生呈负相关。
在蓝绿色模块中,富集分析的结果主要涉及恶性肿瘤的发生,如细胞迁移的正位调节,血管生成,细胞粘附,细胞-基质粘附和细胞外基质(ECM)组织。
对OSCC的肿瘤发生,侵袭,迁移和转移起重要作用。通路分析显示褐色模块富集在生物化合物代谢通路,
如类固醇生物合成,亮氨酸和异亮氨酸降解,抗生素的生物合成和甘油脂代谢通路中。
在蓝绿色模块中,与细胞粘附调节有关的途径显著富集,如粘着斑,PI3K-Akt信号通路和ECM受体相互作用通路,
这些与恶性肿瘤密切相关。对前面提到的DEG进行GO富集分析和KEGG通路分析。
WGCNA和DEGs的富集分析结果相似,这意味着结果是可靠的。
结果6 Hub基因的验证
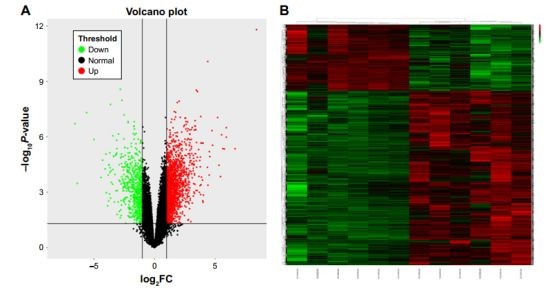
本文使用另一组数据集GSE74530来验证10个hub基因的表达状态。将阈值设置为logFC>| 1 | 和P<0.05,
以筛选差异表达基因。下图显示的分别是DEG的火山图和层次聚类热图。
- 发表于 2018-10-16 13:25
- 阅读 ( 36922 )
- 分类:转录组学
你可能感兴趣的文章
- WGCNA 一键式分析工具 15967 浏览
- 零代码系列之三教你如何进行功能富集 5959 浏览
- 零代码系列之三教你如何看懂WGCNA分析结果 20407 浏览
- 零代码系列之三教你如何做WGCNA数据准备 14031 浏览
- 用TCGA数据不用编程轻轻松松构建CeRNA网络 7684 浏览
- WGCNA分析,简单全面的最新教程 9261 浏览
3 条评论
请先 登录 后评论
