近期可以模仿的数据分析pipeline汇总
看教程不够直观,那就看视频吧! >>点击加载视频
文章标题是Genetic and Environmental Determinants of Immune Response to Cutaneous Melanoma
大家可以通过doi自行查看,doi.org/10.1158/0008-5472.CAN-18-2864
文章主要做的是一个事情是鉴定黑色素瘤的不同亚型,并且阐述不同亚型中差异基因的变化,基因组CNV的变化,还有某一基因在不同亚型中对预后的关系和对PDL1的免疫响应。
背景和论述的过程大家可以看一下原文,不太难懂,我们主要讲一下结论和分析的pipeline
重要结论:
1.基于免疫细胞得分构建了不同免疫响应亚型,并且亚型之间确实存在显著的预后差异。
2.通过对不同响应亚型组之间的差异表达基因进行富集分析,发现MYC,NFKB1基因与这种免疫响应之间的关系
3.CNV分析表明在不同的响应亚型组之间MYC,NFKB1基因的突变频率也有显著的差异
4.对各组进行临床特征分析发现smoking是高免疫响应亚型的风险因子
文章用到的数据比较多,包含TCGA带有临床特征的数据 GEO数据 CNV数据和突变数据。
第二篇文章来自Oncoimmunology,实时的影响因子是5.333
文章题目Two immune-enhanced molecular subtypes differ in inflammation, checkpoint signaling and outcome of advanced head and neck squamous cell carcinoma
文章链接:doi.org/10.1080/2162402X.2017.1392427
先看分析结果:
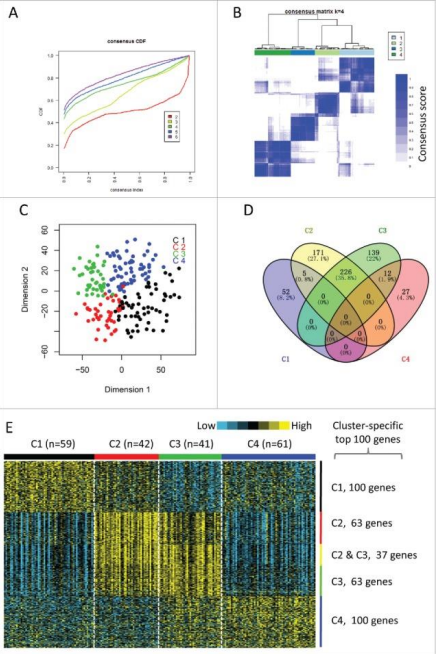
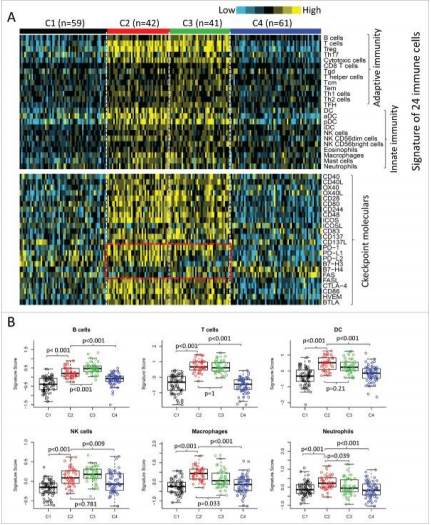
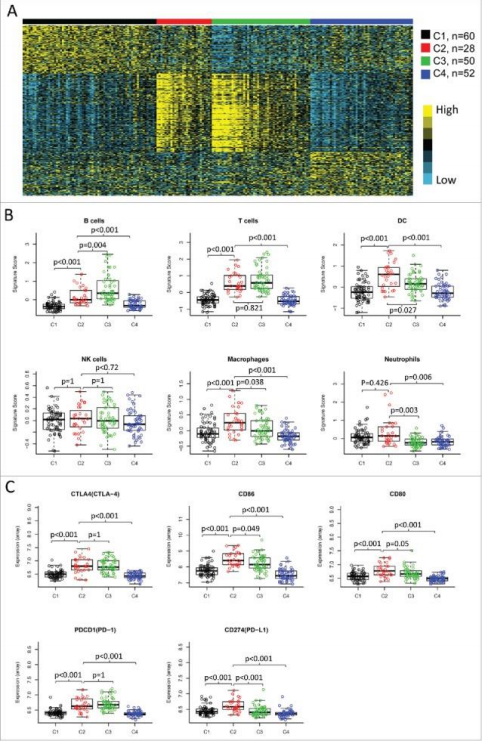
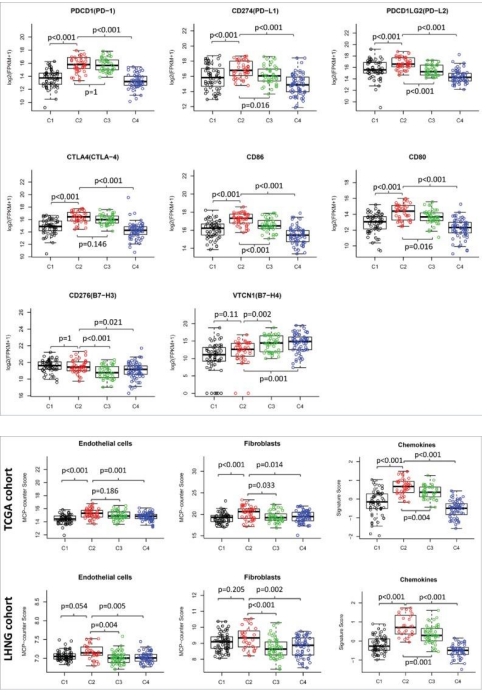
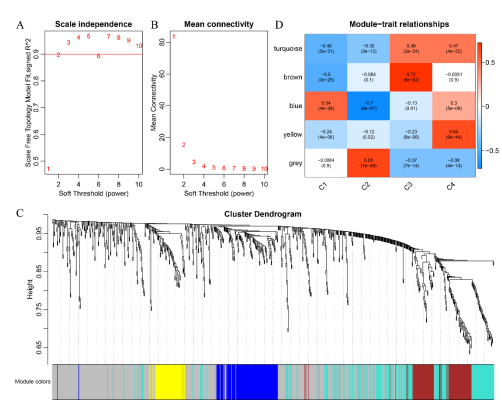
文章的分析方法也相对好梳理:
1、首先筛选具有预后信息的 TCGA\GEO 数据集 2、下载收集免疫相关基因集。 3、利用免疫相关基因集表达谱进行分子分型 4、主成分分析亚型特异性 5、筛选不同分子分型特异表达基因 6、分析不同分子分型中的临床特征 7、分析不同分子分型中免疫细胞、基质细胞评分等免疫浸润的关系,寻找 免疫增强/抑制相关亚型。 8、免疫检查点基因在不同亚型中的差异分析 9、不同亚型的预后差异分析 10、共表达筛选免疫增强/抑制亚型的特征基因 11、构建分类模型预测免疫增强/抑制亚型 12、外部数据集验证
第三篇文章来自,主要方向是肿瘤干细胞特征分析
文章来自比较高大上的CELL,文章的工作量很大,但我们模仿其中精髓就能做一篇不错的文章
标题是:
Machine Learning Identifies Stemness Features Associated with Oncogenic Dedifferentiation
链接是:doi.org/10.1016/j.cell.2018.03.034










文章结论的数量真的非常能体现这个分数段杂志的质量,但有些结果你在其他肿瘤里未必能做出来
如果想模仿其实也有办法,这里不把篇幅扩的太大了,感兴趣可以与小编私聊。
第四篇不是文章,而是整合了多篇文章的一个思路
具体参考的文章有:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6323227/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6412453/
https://www.nature.com/articles/s41598-019-39228-9
https://www.biorxiv.org/content/10.1101/253344v2
https://www.ncbi.nlm.nih.gov/pubmed/29512712
那么整合了这么多篇文章的思路究竟是什么样的呢?
1.准备数据
2.鉴定gene signature
3.功能注释
4.PPI网络分析挖掘hub gene
5.gene signatures 参与调控的 miRNA 分析
6.蛋白表达验证
7.药物相关靶点分析和潜在药物分析
看上去是不是挺简单的,但具体分析中的坑还是挺多的,不过好在这些坑小编团队已经提前踩过了,如果你想节省时间,欢迎与我们合作。
- 发表于 2019-10-28 19:51
- 阅读 ( 7037 )
- 分类:文献解读
你可能感兴趣的文章
- 芦笋基因组揭示了Y染色体的起源和进化 8959 浏览
- 基因组去哪儿了 8707 浏览
- 干货|基因注释方法论 9430 浏览
- TCGA CNV全攻略 19425 浏览
- 纯三代向日葵基因组登上Nature 6276 浏览

