文献分享(6)对表观遗传酶进行综合泛癌分析揭示了癌症中表观基因组失调的普遍模式
看教程不够直观,那就看视频吧! >>点击加载视频

 最近在癌症基因组学中最重要的发现之一是鉴定新的驱动突变,其通常靶向调节全基因组染色质和DNA甲基化标记的基因。然而,鲜为人知的是这些基因是否表现出超越癌症类型的表观基因组失调的模式。那么今天小编为大家带来的这篇文章就是对泛癌中表观遗传酶基因进行综合分析以表征癌症中表观基因组失调的模式,发表在Genome Biology上,影响因子13.214。
最近在癌症基因组学中最重要的发现之一是鉴定新的驱动突变,其通常靶向调节全基因组染色质和DNA甲基化标记的基因。然而,鲜为人知的是这些基因是否表现出超越癌症类型的表观基因组失调的模式。那么今天小编为大家带来的这篇文章就是对泛癌中表观遗传酶基因进行综合分析以表征癌症中表观基因组失调的模式,发表在Genome Biology上,影响因子13.214。

(1)EE基因列表:通过检索文献收集EE(epigenetic enzymes)基因,共212个。
(2)TCGA基因表达数据:从TCGA中下载10种癌症类型的RNA-Seq level3表达数据,定量为RSEM。
(3)TCGA DNAm数据:从TCGA中下载使用Illumina Infinium HumanMethylation450 BeadChip阵列生成的10种癌症类型的DNAm数据。
(4)Erlangen Illumina 450k乳腺癌DNAm数据:在Bavarian Breast Cancer Cases and Controls
Study 2中收集了21个与乳腺癌相邻的正常样品和165个乳腺癌样品的Illumina 450k DNAm数据。

1.全基因组EE基因的差异表达分析
在这一部分,作者对212个“表观遗传酶”(EE)基因的综合列表进行泛癌差异表达分析,然后作者计算了每个癌症样本的两个表观遗传不稳定指数(HyperZ和HypoZ),反映了DNAm模式与正常参考的全局偏差。简而言之,HyperZ指数测量给定癌症样品中启动子CpG岛(CGI)上的异常高甲基化,而HypoZ测量开放式探针上的异常低甲基化。接下来,作者利用TCGA肿瘤样本的匹配RNA序列和DNA序列数据,对EE基因的表达水平与这两个表观遗传不稳定性指数进行泛癌相关分析,以确定EE基因的表达变异与异常癌症DNAm的相关性。最后,作者使用EE基因的因果网络建模,其显示一致的差异表达和与癌症类型的HyperZ / HypoZ的相关性,以鉴定控制DNAm的全局变异的EE基因子集(HyperZ / HypoZ)。
 图一:鉴定癌症DNA甲基化组的主要表观遗传调节因子
图一:鉴定癌症DNA甲基化组的主要表观遗传调节因子
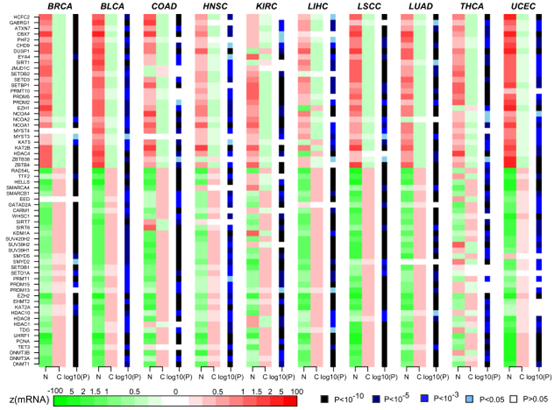 图二:泛癌表观遗传酶基因的差异表达分析
图二:泛癌表观遗传酶基因的差异表达分析
2.全基因组水平的DNA高甲基化和低甲基化仅在癌症中微弱相关
由于许多EE基因在癌症中异常表达,作者想要鉴定那些可能控制癌症中的异常DNAm模式的基因。对于每个癌症样品,作者分别获得总体HyperZ s和HypoZ s指数,其反映该样品中异常DNA高甲基化和低甲基化的整体水平。对于给定组织类型的所有癌症样品,作者绘制了HyperZ和HypoZ不稳定性指数的相关性,结果显示在这么多癌症类型中,两个DNAm指数之间缺乏强相关性。
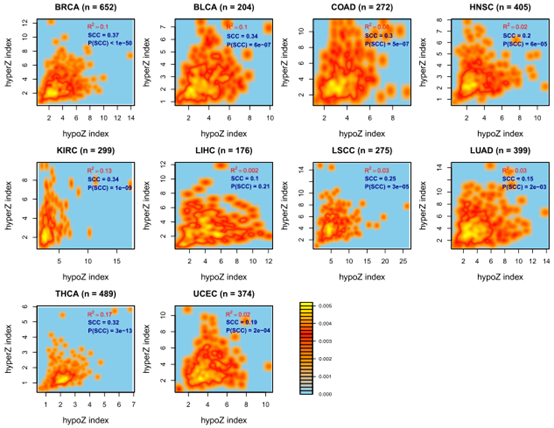
图3 全基因组低甲基化和高甲基化相关性较弱
3.EE基因表达与DNAm之间的泛癌相关性分析揭示了表观基因组失调的普遍模式
在发现异常DNA高甲基化和低甲基化通常不相关之后,作者接下来决定调查这两个不同的DNAm不稳定性指数是否由特定EE基因的表达模式决定。因此作者进行了EE基因表达与DNA甲基化之间的泛癌相关性分析。结果显示在十种癌症类型中至少有六种中表现出显著且定向一致的相关性的EE基因。
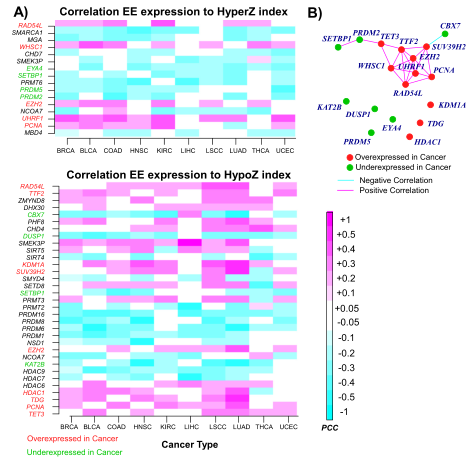 图4:EE表达与DNA甲基化之间的泛癌相关性分析
图4:EE表达与DNA甲基化之间的泛癌相关性分析
4.癌症DNAm的候选表观遗传调节因子
为了鉴定可能代表癌症中全局DNAm模式的主要调节因子的EE基因,作者关注那些在癌症类型中表现出一致的差异表达和DNAm不稳定性相关模式的EE基因。一共鉴定了18个基因,接下来作者验证这18个基因是否独立,计算了所有癌症样本中这18个基因之间mRNA表达的相关性,并将结果组合在荟萃分析中,揭示了正相关癌基因的核心簇,其中包括UHRF1 ,EZH2,TTF2,SUV39H2,PCNA,WHSC1和RAD54L(图4b)。
由于EE基因表达与全球DNAm指数之间的相关性可能不代表直接影响(图5a)。为了确定癌症中DNAm模式的更可能的驱动因素,作者设计了一种实现部分相关的因果网络建模策略以消除间接相关性。首先,作者分别为18个EE基因中的每一个实施了这一策略。这表明,对于大多数情况,EE基因的启动子DNAm无法解释它们的表达与表观遗传不稳定性指数之间的关联。接下来,作者在推理过程中使用所有先前确定的18个EE基因实施了因果网络策略。 总结每种癌症类型的推断的部分相关网络(图5b)预测UHRF1和WHSC1的过表达和CBX7的低表达可能分别是癌症DNA高甲基化和DNA低甲基化的关键驱动因素(图5C)。
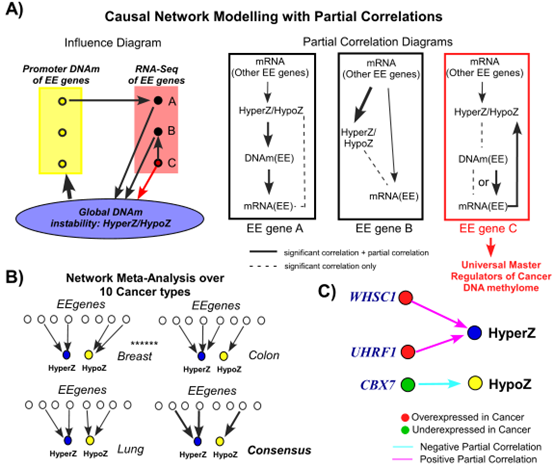 图5因果网络建模荟萃分析
图5因果网络建模荟萃分析
5.癌症甲基化组的候选调节剂影响不同癌症类型的相同基因座的DNAm水平
最后,作者探究了预测的调节因子(UHRF1,WHSC1和CBX7)是否影响癌症类型的相同基因组位点的DNAm。为此,作者根据它们与UHRF1,WHSC1或CBX7的基因表达的关联水平,对先前用于构建HyperZ和HypoZ指数的基因组区域进行排序。结果表明那些DNAm水平与UHRF1或WHSC1的基因表达增加最强相关的基因组位点在大多数其他癌症类型中也显著高度排名。类似地,其DNAm水平与CBX7的基因表达降低最强相关的基因座在其他癌症类型中也是最显著的排名。因此,作者可以看到这些候选主调节因子倾向于影响相同的基因组位点,导致相似的DNAm模式,与组织类型无关。
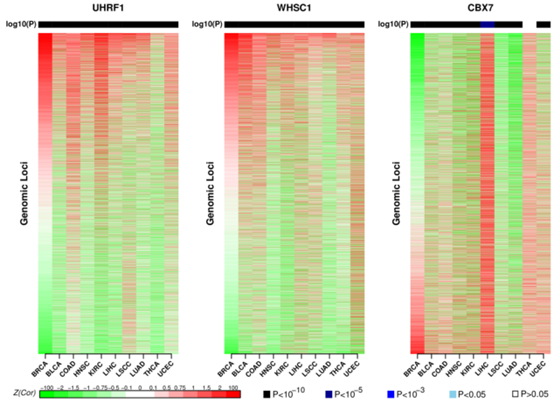 图6 EE基因表达与不同癌症类型的个体基因组位点的DNAm水平的相关热图
图6 EE基因表达与不同癌症类型的个体基因组位点的DNAm水平的相关热图

该研究表明存在超越癌症类型的表观基因组失调的普遍模式,并且全基因组DNA低甲基化和高甲基化的肿瘤内水平由不同的过程控制。

文献分享(3)sEV相关基因表达特征(3-PPI-Mod)与肿瘤内缺氧状态显著相关???
文献分享(4)联合抑制PD-L2和TGF-β2可能会提高免疫治疗的效果?
文献分享(7)circRNA特征预测II / III期结肠癌术后复发
文献分享(8)RNA-Seq分析人肺腺癌和鳞状细胞癌中的环状RNA
文献分享(9)m6A调控子在33种癌症类型中的分子特征和临床相关性
- 发表于 2019-11-25 19:34
- 阅读 ( 3816 )
- 分类:文献解读
你可能感兴趣的文章
- 文献解读(3)what?铁代谢的分子治疗可能对癌症的治疗有帮助? 5220 浏览
- Weinberg教授继的癌症综述 4869 浏览
