文献分享(5)marker识别并分型
看教程不够直观,那就看视频吧! >>点击加载视频


乳头状甲状腺癌(PTC)是一种异质性疾病,具有不同的临床结局,因此识别具有临床相关性的生物标志物对其至关重要。今天小编要和大家分享的就是一篇识别PTC中的基因signature的文章【Immune Gene Signature Delineates a Subclass of Papillary Thyroid Cancer with Unfavorable Clinical Outcomes;2018.18; Cancer; IF:6.162】
在这项研究中,作者从TCGA等获得了包括PTC癌旁组织在内的大规模表达数据,使用基于去卷积,非负矩阵分解,以及免疫组化等手段对PTC进行了全面精细的刻画。

1.公开PTC转录组数据:
1)作者在TCGA获得甲状腺癌的表达数据。
2)作者同样得到了癌旁组织的对照数据。
3)作者同时在TCGA之外收集了180例甲状腺癌的表达数据,作为验证数据。
2. 文章研究用到的主要方法流程:
1) 转录组测序以及数据预处理
2) PTC的非负矩阵分解(NMF)来分析metagene特征
3) 功能注释:GSEA
4) 免疫谱系:使用CIBERSORT方法评估肿瘤浸润的免疫细胞的类型
5) 免疫组化分析,对转录组的分析结果进行验证
6) 分析不同分组的预后情况,主要包括单因素分析以及多因素分析,生存分析等
7) 使用方差分析卡方检验等统计学方法分析组间表达差异

1.将PTC表达谱去卷积得到关键metagene特征
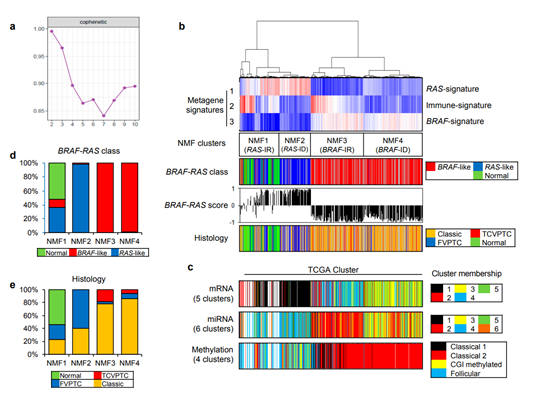 图1
图1
作者在第一步首先进行了关键metagene signature的识别。作者使用非负矩阵分解的方法对PTC的表达谱数据进行聚类。图1a的系数图衡量了metagene的稳定性,可以看出在表达谱中至少存在3个metagene特征。图1b可以看出基于这些特征水平可以使用NMF将表达谱分成四类。图1c将作者得到的四个类与之前研究提出的PTC分类进行比较。图1d展示了这四类中类BRAF以及类RAS的比例,而图e则展示了这四类的组织学分类情况。
2.免疫相关Metagene特征的功能注释
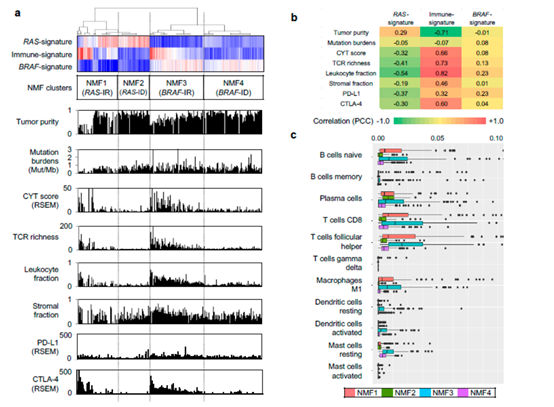 图2
图2
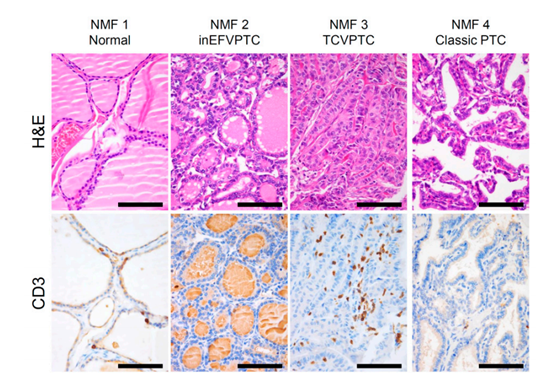 图3
图3
作者在这部分又对之前得到的metagene特征进行了功能注释。作者主要使用GSEA进行功能富集。除此之外作者进一步评估了与免疫细胞丰度相关的其他基因组病理学特征,如肿瘤纯度、总突变量、CYT活性评分等,如图2a所示。图2b则展示了对这三个metagene signature与免疫特征相关性的衡量结果。作者进一步使用CIBERSORT算法评估了免疫细胞的亚型,评估结果如图2c所示。图3则展示了这四类的免疫组化情况,对之前的评估结果进行了验证。
3.在TCGA数据中基于NMF分类的PTC的预后
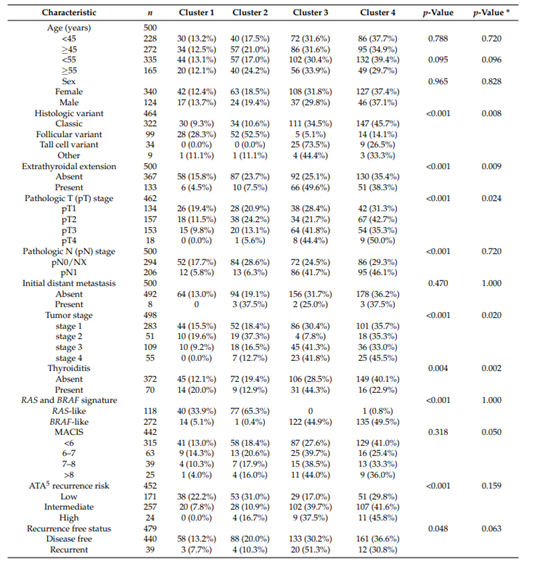 表2
表2
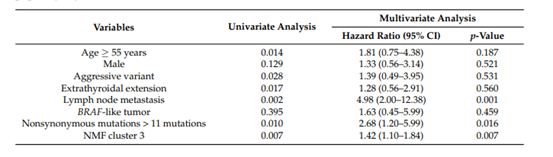 表3
表3
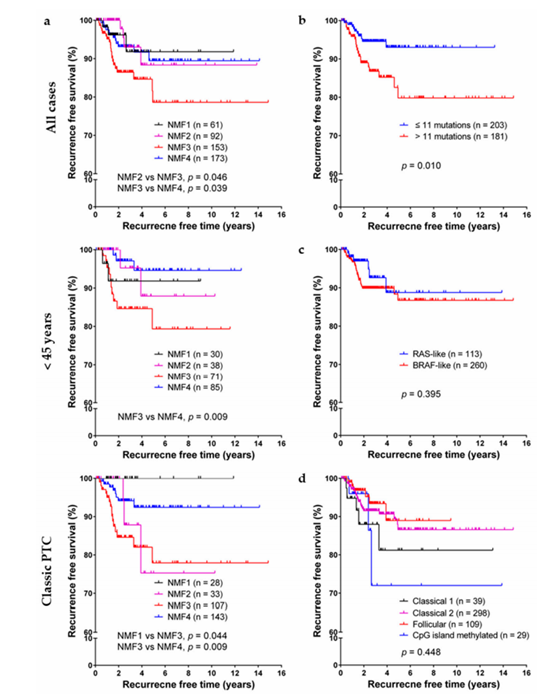 图4
图4
作者在这一部分对TCGA数据分析中得到的不同PTC亚型进行了预后分析,来研究其临床结局。NMF分类的不同亚型的患者的基本特征展示在表1中。表2,3则展示了对这些临床特征进行单因素分析以及多因素分析的结果。接下来作者又在不同水平进行了生存分析,来研究不同分组的预后情况,生存分析的结果如图4所示。
4.使用两套外部数据进行NMF分类结果的验证
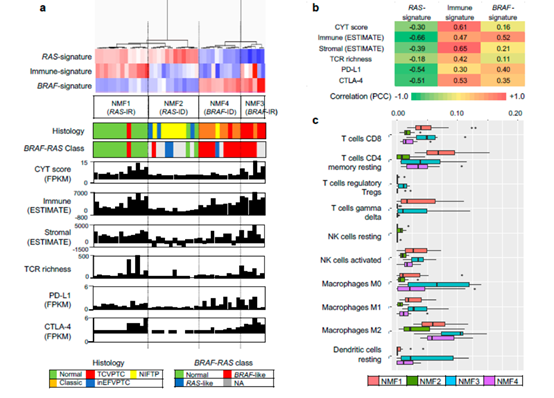 图5
图5
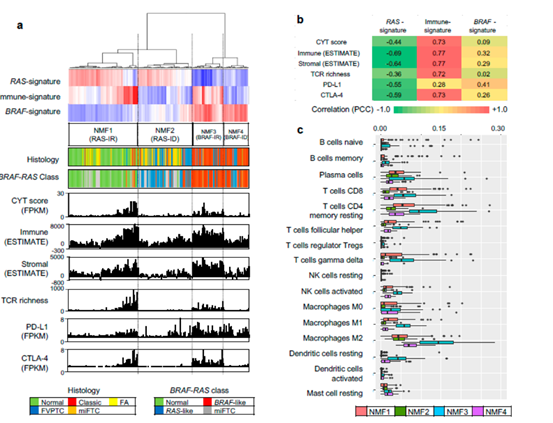 图6
图6
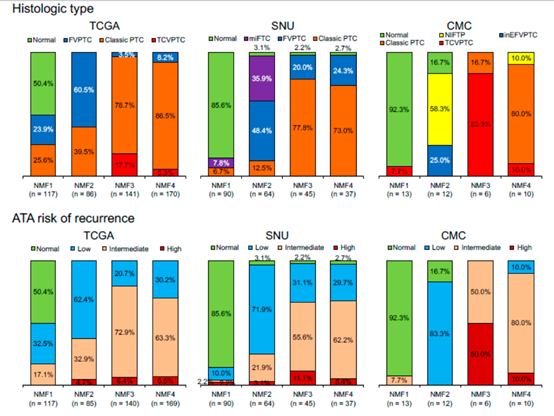 图7
图7
在文章的最后一部分作者使用非TCGA的外部数据,对文章的研究结果进行了验证。作者对两套验证数据采用了与TCGA数同样的操作流程,从图5,以及图6中可以看出验证结果支持了作者发现的免疫相关的基因特征,接着作者又对这三套数据在组织类型等层面进行了综合分析。
 作者收集了TCGA数据以及其他的验证数据,对PTC的转录组数据进行了详细分析,使用非负矩阵分解,去卷积等方法,识别出了PTC中与免疫相关的基因signature,能够对PTC进行性分型,并对不同亚型进行了生存分析以及多层面刻画。概括来说这是一篇marker识别并分型的文章,其中无论是研究的方法还是流程以及角度都值得我们学习借鉴。
作者收集了TCGA数据以及其他的验证数据,对PTC的转录组数据进行了详细分析,使用非负矩阵分解,去卷积等方法,识别出了PTC中与免疫相关的基因signature,能够对PTC进行性分型,并对不同亚型进行了生存分析以及多层面刻画。概括来说这是一篇marker识别并分型的文章,其中无论是研究的方法还是流程以及角度都值得我们学习借鉴。

文献分享(3)sEV相关基因表达特征(3-PPI-Mod)与肿瘤内缺氧状态显著相关???
文献分享(4)联合抑制PD-L2和TGF-β2可能会提高免疫治疗的效果?
文献分享(6)对表观遗传酶进行综合泛癌分析揭示了癌症中表观基因组失调的普遍模式
文献分享(7)circRNA特征预测II / III期结肠癌术后复发
- 发表于 2019-11-25 19:26
- 阅读 ( 6342 )
- 分类:文献解读
你可能感兴趣的文章
- 多因素生存分析工具 5080 浏览
- 一键式的批量生存分析工具 4863 浏览
- KaplanMeier生存曲线绘制工具 11242 浏览
- 文献分享(3)sEV相关基因表达特征(3-PPI-Mod)与肿瘤内缺氧状态显著相关??? 4581 浏览
- 生信文献解读(1)多组学数据的生存分析识别胰腺癌中的预后标志物 10148 浏览
