一键式WGCNA,WGCNA的简易便捷分析软件
看教程不够直观,那就看视频吧! >>点击加载视频
WGCNA加权基因共表达网络分析主要用来分析不同样品之间基因关联模式的系统生物学方法,WGCNA适用于复杂的数据模式,15个样品以上的数据,利用数千或近万个变化最大的基因或全部基因的信息识别感兴趣的基因集,并与表型进行显著性关联分析,优势在于一是充分利用了信息,二是把数千个基因与表型的关联转换为数个基因集与表型的关联,免去了多重假设检验校正的问题。
首先介绍一下工具界面每一部分的说明,直接上图:
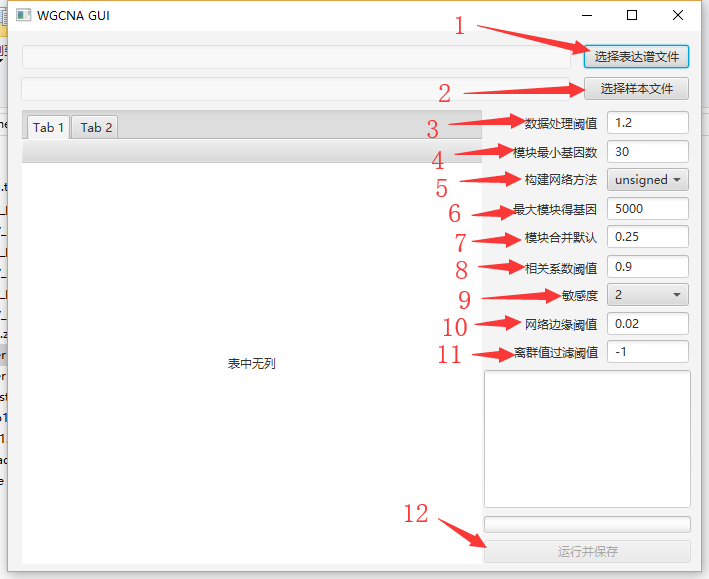
1、待分析表达数据,通常行为基因,列为样本,事先需要标准化好,如有批次效应,同样需要处理 测试数据 ;
2、对应样本的表型数据,倘若没有暂时先伪造一个,测试数据;
3、对表达数据进行过滤,这里使用方差进行过滤,过滤掉差异不是很明显的基因,这里默认1.2,可根据自己数据进行调整;
4、网络构建时一个模块中最少需要包含的基因个数,值越小,小的模块就会被保留下来;
5、构建网络的方法,三种方式:1)无向网络的边属性计算方式为abs(cor(genex, geney)) ^ power;2)有向网络的边属性计算方式为(1+cor(genex, geney)/2) ^ power;3)sign
hybrid的边属性计算方式为cor(genex, geney)^power if cor>0 else 0,通常使用最多的是无向网络,当然这个需要根据数据情况来判定,这个可以看下陈老师博文;
6、网络构建时运行过程中最大模块,这个需要根据自己的电脑性能来断定,性能好多给些,差就少给一点,不会对结果有什么影响;
7、合并模块参数,这个根据想要模块个数来设定,越大得到的模块数越少;
8、相关性分析,采用pearson计算方法;
9、敏感度0-3,如果我们给定基因个数并不是很多或者得到模块太少可以调大这个参数这个值越大,得到的模块就越多;
10、边缘的临接阈值,默认0.02,同样可以根据自己的需要调整,导出后在cytoscape中再调整也是可以的;
11、离群值是否过滤,这个在软阈值筛选前检测,想要过滤离群样本除了-1随便给数值;
12、点击运行保存耐心等待结果就可以了,这个运行时间跟数据量多少,电脑性能时密切相关,如果样本量大,可以运行上然后看看最新文章。
这里小编上传了一套测试数据,各位可以下载尝试分析下,自己需要分析的数据就可以仿照测试数据的格式编辑,运行解说后,在给定分析目录下会有一个Report.html,打开它就可以看到报告啦!!报告部分截图如下

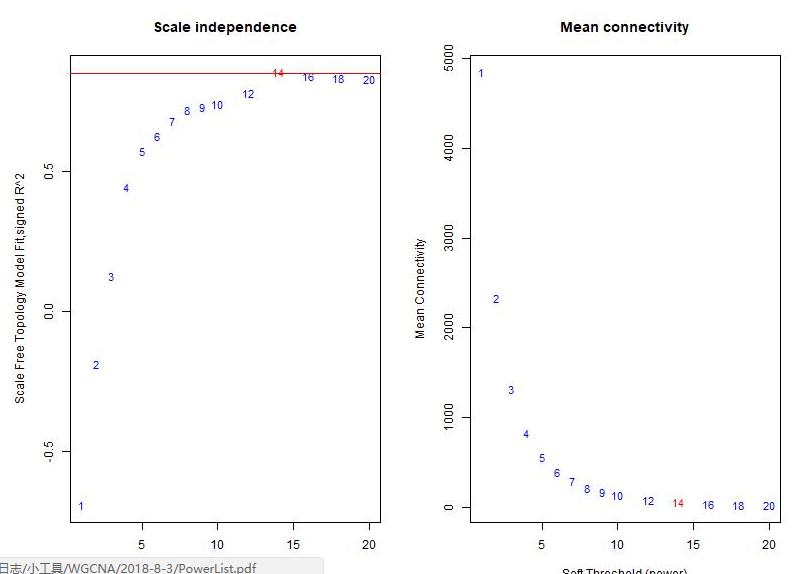
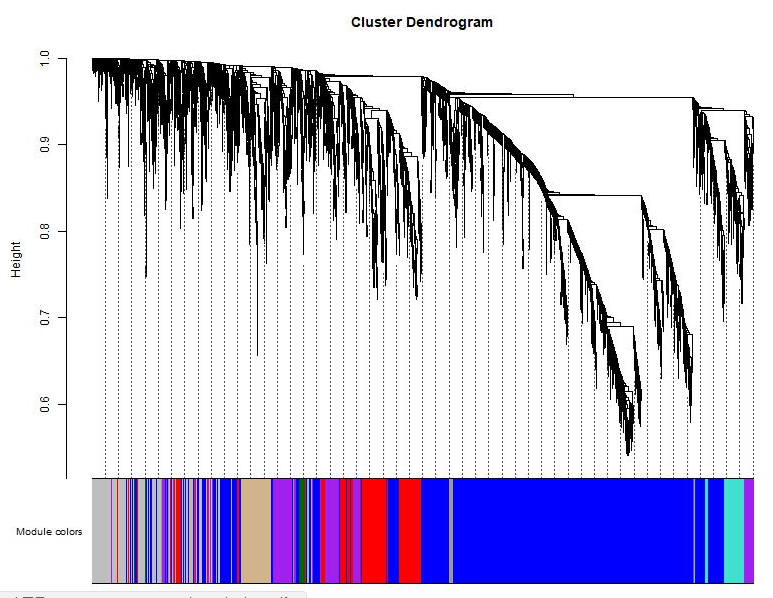
如果打开这个文件提示WGCNA尚未运行完毕,说明分析没有结束;已经提示运行完成,就是运行出现异常,检查右下角的小窗口看看提示信息,自己解决不了可以地下留言或社区提问题,记得留下报错截图,不然技术老师也很难发现问题所在 ^-^
- 发表于 2018-09-03 09:05
- 阅读 ( 22841 )
- 分类:基因组学
你可能感兴趣的文章
- WGCNA 一键式分析工具 16022 浏览
- 零代码系列之三教你如何进行功能富集 5971 浏览
- 零代码系列之三教你如何看懂WGCNA分析结果 20419 浏览
- 零代码系列之三教你如何做WGCNA数据准备 14046 浏览
- 用TCGA数据不用编程轻轻松松构建CeRNA网络 7698 浏览
- WGCNA分析,简单全面的最新教程 9294 浏览
